Korunan imza indelleri - Conserved signature indels - Wikipedia
Korunan imza ekleme ve silme işlemleri (CSI'lar) protein dizilerinde filogenetik ilişkileri anlamak için önemli bir moleküler belirteç kategorisi sağlar.[1][2] Nadir genetik değişikliklerin neden olduğu CSI'lar, genellikle tanımlanmış büyüklükte olan ve güvenilirliklerini sağlamak için her iki tarafta korunmuş bölgelerle çevrelenmiş yararlı filogenetik belirteçler sağlar. Süre Indels rastgele ekler veya çıkarmalar olabilir, CSI'lar yalnızca proteinin korunmuş bölgelerinde bulunan protein indelleri olarak tanımlanır.[2][3][4][5]
Belirli bir kullanıcıyla sınırlı olan CSI'lar clade veya türler grubu, genellikle iyi filogenetik belirteçler ortak evrimsel köken.[2] Bu tür değişikliklerin nadirliği ve oldukça spesifik doğası nedeniyle, her ikisinden de bağımsız olarak ortaya çıkma olasılığı daha düşüktür. yakınsak veya paralel evrim (yani homoplazi) veya sinapomorfi. Farklı bölgelerdeki veya farklı türler arasındaki evrim oranlarındaki farklılıklar gibi diğer karıştırıcı faktörler de genellikle bir CSI'nın yorumlanmasını etkilemez.[2][3] Bir grup dışı türde CSI'lerin varlığını veya yokluğunu belirleyerek, CSI'nın atalarından kalma formunun bir eklenti mi yoksa silinme mi olduğu sonucuna varılabilir ve bu, organizmalar arasında köklü bir filogenetik ilişki geliştirmek için kullanılabilir.[1][2]
Tanımlanan çoğu CSI'nın yüksek tahmin değeri sergilediği bulunmuştur ve genellikle orijinal olarak tanımlanan tür sınıfları için özgünlüğü muhafaza ederler. Bu nedenle, varlığına veya yokluğuna bağlı olarak, bu gruplara ait hem bilinen hem de daha önce bilinmeyen türlerin farklı ortamlarda belirlenmesi mümkün olmalıdır.[3]
Türler
Gruba özgü

Gruba özgü CSI'lar genellikle belirli bir gruba ait farklı türler tarafından paylaşılır. Takson (örneğin, cins, aile, sınıf, sıra, filum) ancak diğer gruplarda mevcut değillerdir. Bu CSI'lar büyük olasılıkla türler grubunun bir atasına, takson ayrıldı. Belirli bir taksonun üyelerini diğer tüm organizmalardan ayırmak için moleküler araçlar sağlarlar.[2][5]
Şekil 1 X taksonuna ait tüm türlerde bulunan bir 5aa CSI örneğini göstermektedir. Bu, başka hiçbir türde bulunmadığından bu taksonun ayırt edici bir özelliğidir. Bu imza, muhtemelen bu taksondaki türlerin ortak bir atasında tanıtıldı. Benzer şekilde diğer gruba özgü imzalar (gösterilmemiştir) A1 ve A2 veya B1 ve B2, vb. Veya X1 ve X2 veya X3 ve X4, vb. Tarafından paylaşılabilir. A, B, C, D ve X, bu diyagramda çeşitli bakteriyel veya Ökaryotik filum.[6]
Gruba özgü CSI'lar geçmişte bir dizi bakteri filumunun ve içindeki alt grupların filogenetik ilişkisini belirlemek için kullanılmıştır. Örneğin, 3 amino asitli bir eklenti, filumun üyeleri tarafından benzersiz bir şekilde paylaşıldı Termotoga temelde 50S ribozomal protein L7 / L12 yüksek oranda korunmuş bir bölge içinde (82-124 amino asit). Bu, başka hiçbir bakteri türünde mevcut değildir ve filumun üyelerini karakterize etmek için kullanılabilir. Termotoga diğer tüm bakterilerden. Filum içindeki alt grupları karakterize etmek için gruba özgü CSI'lar da kullanılmıştır. Termotoga.[7]
Çoklu grup veya ana hat

Ana hat CSI'lar, korunmuş bir ekleme veya silme işleminin birkaç ana filum tarafından paylaşıldığı, ancak diğer filumlarda bulunmayanlardır.[2]
şekil 2 X, Y ve Z filumlarına ait türlerde yaygın olarak bulunan, ancak diğer filumlarda (A, B ve C) bulunmayan korunmuş bir bölgede bulunan 5aa CSI örneğini gösterir. Bu imza, X, Y ve Z taksonlarının ve ayrıca A, B ve C'nin spesifik bir ilişkisini gösterir. Böyle bir indelin varlığına veya yokluğuna dayanarak, grup dışı türlerde (yani Archaea), indel bir ekleme veya bir silme işlemidir ve bu iki gruptan A, B, C veya X, Y, Z hangisinin atasıdır.[6]
Geçmişte bir dizi bakteri filumunun filogenetik ilişkisini belirlemek için ana hat CSI'lar kullanılmıştır. Korunan bir bölge içinde yaklaşık 150-180 amino asitlik büyük CSI Girase B (529-751 amino asitleri arasında), yaygın olarak çeşitli Proteobakteriler, Chlamydiales, Planctomycetes ve Aquificales Türler. Bu CSI, diğer atalara ait bakteri filumlarında ve ayrıca Archaea.[8] Benzer şekilde, yaklaşık 100 amino asitlik büyük bir CSI RpoB homologlar (amino asitler 919-1058 arasında), aşağıdakilere ait çeşitli türlerde mevcuttur Proteobakteriler, Bacteroidetes-Klorobi, Chlamydiales, Planctomycetes ve Aquificales. Bu CSI, diğer atalara ait bakteri filumlarında ve ayrıca Archaea.[9][10] Her iki durumda da CSI'dan yoksun grupların atalarının olduğu sonucuna varılabilir.
CSI'lara dayalı evrimsel çalışmalar


Bakteriyel filogenide önemli bir konu, farklı bakteri türlerinin birbirleriyle nasıl ilişkili olduğunu ve ortak bir atadan dallanma sıralarını anlamaktır. Şu anda çoğu filogenetik ağaç, 16S rRNA veya diğer genler / proteinler. Bu ağaçlar her zaman anahtar filogenetik soruları yüksek derecede kesinlik ile çözemezler.[11][12][13][14][15] Bununla birlikte, son yıllarda evrensel olarak dağıtılmış birçok proteindeki korunmuş indellerin (CSI) keşfi ve analizleri bu arayışa yardımcı olmuştur. Onlara yol açan genetik olayların, önemli evrimsel dallanma noktalarında meydana geldiği varsayılmaktadır ve bunların tür dağılım modelleri, farklı bakteri filumları arasındaki dallanma düzeni ve karşılıklı ilişkiler hakkında değerli bilgiler sağlar.[1][2][7]
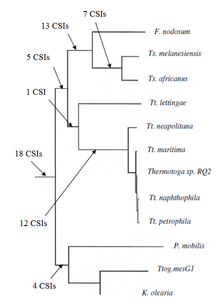
Termotoga
Son zamanlarda grubun filogenetik ilişkisi Termotoga CSI yaklaşımına göre karakterize edildi. Daha önce hiçbir biyokimyasal veya moleküler belirteçler bu filumun türlerini diğer tüm bakterilerden açıkça ayırt edebildiği biliniyordu. Tüm Thermotogae filumuna veya farklı alt gruplarına özgü 60'tan fazla CSI keşfedildi. 18 CSI, çeşitli Thermotogae türlerinde benzersiz bir şekilde bulunur ve filum için moleküler belirteçler sağlar. Ek olarak, çeşitli termotoga alt gruplarına özgü birçok CSI vardı. 12 CSI, Tt haricinde çeşitli Thermotoga türlerinden oluşan bir grup için spesifikti. Lettingae. 14CSI'ler, Fervidobacterium ve Thermosipho cinslerinden oluşan bir küme için spesifiktir ve 18 CSI, Thermosiphon cinsine özgüdür.[kaynak belirtilmeli ]
Son olarak, Thermotogae türlerinin bir kısmı veya tamamı veya diğer taksonlardan bazı türler tarafından paylaşılan 16 CSI rapor edilmiştir. Archaea, Aquificae, Firmicutes, Proteobakteriler, Deinococcus, Fusobacteria, Diktiyoglomus, Klorofleksi ve ökaryotlar. Bu CSI'lardan bazılarının ortak varlığı, yanal gen transferi (LGT) bu gruplar arasında. Bununla birlikte, diğer taksonlarla ortak olarak paylaşılan CSI'lerin sayısı, Thermotogae için özel olanlardan çok daha azdır ve herhangi bir spesifik model göstermezler. Dolayısıyla Termotoga ayrımı üzerinde önemli bir etkisi yoktur.[7]
Archaea
Mezofilik Crenarchaeotes yakın zamanda yeni bir filumun içine yerleştirildi Archaea aradı Thaumarchaeota. Bununla birlikte, bu arke grubunu Crenarchaeota filumundan ayıran çok az moleküler işaret vardır. Bu filumları moleküler terimlerle ayırt etmek için CSI yaklaşımını kullanan ayrıntılı bir filogenetik çalışma yapılmıştır. 6 CSI, çeşitli Thaumarchaeota'da benzersiz bir şekilde bulundu. Cenarchaeum symbiosum, Nitrosopumilus maritimus ve bir takım kültürlenmemiş deniz crenarchaeotları. Thaumarchaeota ve Crenarchaeota'ya ait türler arasında yaygın olarak paylaşılan 3 CSI bulundu. Ek olarak, Crenarchaeota-3 CSI'ların farklı sıralarına özel bir dizi CSI bulundu. Sulfolobales, 5 CSI için Termoprotealler, son olarak 2 CSI için ortak Sulfolobales ve Desulfurococcales. Tanımlanan imzalar, Crenarchaeota ve Thaumarchaeota'yı ayırt etmek için yeni araçlar sağlar, ayrıca ilgili türlerin sınıflandırılması ve tanımlanması için bir araç olarak kullanılabilirler.[16]
Pastöreliler
Siparişin üyeleri Pastöreliler şu anda temel olarak 16srRNA ağacının dallanmasındaki konumlarına göre ayırt edilmektedir. Şu anda bu düzenin üyelerini diğer bakterilerden ayırabilen çok az sayıda moleküler belirteç bulunmaktadır. Son zamanlarda bu sırayla türler arasındaki filogenetik ilişkileri aydınlatmak için bir CSI yaklaşımı kullanıldı; Türlerin tümü veya çoğu tarafından benzersiz şekilde paylaşılan 40'tan fazla CSI keşfedildi. Bu Pasteurellales içinde iki ana sınıf oluşur: Clade I, Toplayıcı karakter, Pastörella, Actinobacillus succinogenes, Mannheimia succiniciproducens, Haemophilus influenzae ve Haemophilus somnus, 13 CSI tarafından desteklenmiştir. Actinobacillus pleuropneumoniae'yi kapsayan Clade II, Actinobacillus minör, Haemophilus ducreyi, Mannheimia haemolytica ve Haemophilus parasuis, 9 CSI tarafından desteklenmiştir. Bu sonuçlara dayanarak, Pasteurellale'lerin mevcut tek ailesinden iki farklı aileye ayrılması önerildi. Ek olarak, açıklanan imzalar keşfedilmemiş Pasteurellales türlerini tanımlamak için yeni araçlar sağlayacaktır.[17]
Gammaproteobacteria
Sınıf Gammaproteobacteria en büyük bakteri gruplarından birini oluşturur. Şu anda diğer bakterilerden yalnızca şu şekilde ayırt edilmektedir: 16 sn rRNA temelli filogenetik ağaçlar. Sınıfa veya farklı alt gruplarına özgü hiçbir moleküler özellik bilinmemektedir. Bu sınıfın filogenisini daha iyi anlamak için CSI tabanlı detaylı bir çalışma yapılmıştır. İlk olarak, evrensel olarak dağıtılmış bir dizi proteinin birleştirilmiş dizilerine dayanan bir filogenetik ağaç oluşturuldu. Farklı dallanma sırası emirler of sınıf Gammaproteobacteria (en yakın zamandan en erken ayrılana) şunlardı: Enterobakteriyeller >Pastöreliler >Vibrionales, Aeromonadales >Alteromonadales >Oceanospirillales, Pseudomonadales >Chromatiales, Lejyonelleler, Metilokoklar, Xanthomonadales, Kardiyobakteriyeller, Thiotrichales. Ek olarak, Gammaproteobacteria sınıfının çoğu türüne özgü 4 CSI keşfedildi. İçinde 2 aa silme AICAR transformilaz hariç tüm gammaproteobacteria tarafından benzersiz bir şekilde paylaşıldı Francisella tularensis. İçinde 4 aa silme RNA polimeraz b-alt birimi ve içinde 1 aa silme ribozomal protein L16 siparişlere ait çeşitli türlerde benzersiz olarak bulundu Enterobakteriyeller, Pastöreliler, Vibrionales, Aeromonadales ve Alteromonadales, ancak diğer gammaproteobacteria'da bulunamadı. Son olarak, içinde 2 aa silme lösil-tRNA sentetaz Gammaproteobacteria sınıfının yukarıdaki siparişlerinde ve takımın bazı üyelerinde yaygın olarak mevcuttu Oceanospirillales.[18] Başka bir CSI tabanlı çalışma da Xanthomonadales takımına özel 4 CSI tanımlamıştır. Birlikte ele alındığında bu iki gerçek, Xanthomonadales'in bir monofiletik grup Bu, diğer Gammaproteobacteria'nın atasıdır, bu ayrıca Xanthomonadales'in bağımsız bir alt bölüm olduğunu ve Gammaproteobacteria sınıfındaki en derin dallanan soylardan birini oluşturduğunu gösterir.[4][18]
Mantarlar
Arasındaki tam filogenetik ilişki bitkiler, hayvanlar ve mantarlar iyi anlaşılmadı. Bu ilişkiyi aydınlatmak için küçük bir CSI tabanlı çalışma yapıldı. Hayvanları ve mantarları bir monofiletik grup olarak bir araya getirmek ve bitkileri dışlamak için dört CSI kullanıldı. Bu CSI'lar iki temel hücresel proteinde bulundu, uzama faktörü l ve enolase. Bununla birlikte, geleneksel olarak, mantarlar ve hayvanlar arasındaki bu özel ilişki desteklenmemiştir.[1]
Referanslar
- ^ a b c d Baldauf, S. L. (1993). "Hayvanlar ve Mantarlar Birbirlerinin En Yakın Akrabalarıdır: Birden Fazla Proteinden Eşlik Eden Kanıtlar". Ulusal Bilimler Akademisi Bildiriler Kitabı. 90 (24): 11558–11562. Bibcode:1993PNAS ... 9011558B. doi:10.1073 / pnas.90.24.11558. PMC 48023. PMID 8265589.
- ^ a b c d e f g h Gupta, Radhey S. (1998). "Protein Filogenileri ve İmza Dizileri: Arkaebakteriler, Öbakteriler ve Ökaryotlar Arasındaki Evrimsel İlişkilerin Yeniden Değerlendirilmesi". Mikrobiyoloji ve Moleküler Biyoloji İncelemeleri. 62 (4): 1435–91. doi:10.1128 / MMBR.62.4.1435-1491.1998. PMC 98952. PMID 9841678.
- ^ a b c Gupta, Radhey S .; Griffiths, Emma (2002). "Bakteriyel Filogenide Kritik Sorunlar". Teorik Popülasyon Biyolojisi. 61 (4): 423–34. doi:10.1006 / tpbi.2002.1589. PMID 12167362.
- ^ a b Cutiño-Jiménez, Ania M .; Martins-Pinheiro, Marinalva; Lima, Wanessa C .; Martin-Tornet, Alexander; Morales, Osleidys G .; Menck, Carlos F.M. (2010). "Xanthomonadales'in korunmuş protein imza dizilerine göre evrimsel yerleştirilmesi". Moleküler Filogenetik ve Evrim. 54 (2): 524–34. doi:10.1016 / j.ympev.2009.09.026. PMID 19786109.
- ^ a b Rokas, Antonis; Hollanda, Peter W.H. (2000). "Filogenetik için bir araç olarak nadir görülen genomik değişiklikler". Ekoloji ve Evrimdeki Eğilimler. 15 (11): 454–459. doi:10.1016 / S0169-5347 (00) 01967-4. PMID 11050348.
- ^ a b Gupta, Radhey. "Protein Dizilerinde Korunan Ekler ve Silmeler". Bakteriyel Filogeni. Gupta RS. Alındı 2 Nisan 2012.[güvenilmez tıbbi kaynak? ]
- ^ a b c Gupta, Radhey S .; Bhandari, Vaibhav (2011). "Phylum Thermotogae ve alt grupları için filogeni ve moleküler imzalar". Antonie van Leeuwenhoek. 100 (1): 1–34. doi:10.1007 / s10482-011-9576-z. PMID 21503713.
- ^ Griffiths, E .; Gupta, R. S. (2007). "Filogeni ve proteinlerdeki paylaşılan korunmuş ekler, Verrucomicrobia'nın klamydiae'nin bilinen en yakın serbest yaşayan akrabaları olduğuna dair kanıt sağlar". Mikrobiyoloji. 153 (8): 2648–54. doi:10.1099 / mic.0.2007 / 009118-0. PMID 17660429.
- ^ Gupta, Radhey S. (2003). "Fotosentetik bakteriler arasındaki evrimsel ilişkiler". Fotosentez Araştırması. 76 (1–3): 173–83. doi:10.1023 / A: 1024999314839. PMID 16228576.
- ^ Griffiths, Emma; Gupta, Radhey S. (2004). "Çeşitli proteinlerdeki imza dizileri, Düzenin geç ayrışmasına kanıt sağlıyor Aquificales" (PDF). Uluslararası Mikrobiyoloji. 7 (1): 41–52. PMID 15179606.
- ^ Brown, James R .; Douady, Christophe J .; Italia, Michael J .; Marshall, William E .; Stanhope, Michael J. (2001). "Büyük birleşik protein dizisi veri setlerine dayalı evrensel ağaçlar". Doğa Genetiği. 28 (3): 281–5. doi:10.1038/90129. PMID 11431701.
- ^ Cavalier-Smith, T (2002). "Arkaebakterilerin neomuran kökeni, evrensel ağacın negatif bakteriyel kökü ve bakteriyel büyük sınıflandırma". Uluslararası Sistematik ve Evrimsel Mikrobiyoloji Dergisi. 52 (1): 7–76. doi:10.1099/00207713-52-1-7. PMID 11837318.
- ^ Ciccarelli, F. D .; Doerks, T; Von Mering, C; Creevey, CJ; Snel, B; Bork, P (2006). "Son Derece Çözülmüş Bir Hayat Ağacının Otomatik Olarak Yeniden İnşasına Doğru". Bilim. 311 (5765): 1283–7. Bibcode:2006Sci ... 311.1283C. CiteSeerX 10.1.1.381.9514. doi:10.1126 / science.1123061. PMID 16513982.
- ^ Daubin, V .; Gouy, M; Perrière, G (2002). "Bakteriyel Filogeniye Filogenomik Bir Yaklaşım: Ortak Bir Tarihi Paylaşan Bir Gen Çekirdeğinin Kanıtı". Genom Araştırması. 12 (7): 1080–90. doi:10.1101 / gr.187002. PMC 186629. PMID 12097345.
- ^ Eisen, Jonathan A. (1995). "Bakterilerin Moleküler Sistematik Çalışmaları için Model Bir Molekül Olarak RecA Proteini: RecA Ağaçlarının ve Aynı Türlerden 16S rRNA'ların Karşılaştırılması". Moleküler Evrim Dergisi. 41 (6): 1105–23. Bibcode:1995JMolE..41.1105E. doi:10.1007 / bf00173192. PMC 3188426. PMID 8587109.
- ^ Gupta, Radhey S .; Shami, Ali (2010). "Crenarchaeota ve Thaumarchaeota için moleküler imzalar". Antonie van Leeuwenhoek. 99 (2): 133–57. doi:10.1007 / s10482-010-9488-3. PMID 20711675.
- ^ Naushad, Hafız Sohail; Gupta, Radhey S. (2011). "Pasteurellales takımına özgü olan ve ana sınıflarından ikisini ayıran protein dizilerindeki moleküler imzalar (korunmuş indeller)". Antonie van Leeuwenhoek. 101 (1): 105–24. doi:10.1007 / s10482-011-9628-4. PMID 21830122.
- ^ a b Gao, B .; Mohan, R .; Gupta, R. S. (2009). "Gammaproteobakteriler arasındaki evrimsel ilişkileri aydınlatan filogenomikler ve protein imzaları". Uluslararası Sistematik ve Evrimsel Mikrobiyoloji Dergisi. 59 (2): 234–47. doi:10.1099 / ijs.0.002741-0. PMID 19196760.
