Çoklu tür birleştirme süreci - Multispecies coalescent process
Çoklu Tür Birleştirme Süreci bir örneklem için şecere ilişkilerini tanımlayan stokastik bir süreç modelidir. DNA birkaç türden alınan diziler.[1] Uygulamayı temsil eder birleşik teori birden fazla tür durumunda. Çoklu türlerin birleşimi, tek bir gen için türler arasındaki ilişkilerin ( gen ağacı) türlerin daha geniş tarihinden farklı olabilir ( tür ağacı). Teori ve pratiği için önemli çıkarımlara sahiptir. filogenetik[2][3] ve genom evrimini anlamak için.
Bir gen ağacı rekombinasyon olmayan bir lokus için bir dizi örneği arasındaki evrimsel ilişkileri tanımlayan ikili bir grafiktir. Gibipecies ağacı Ağaç benzeri evrimi varsayarak bir dizi tür arasındaki evrimsel ilişkileri açıklar. Bununla birlikte, çeşitli süreçler arasında uyumsuzluklara yol açabilir. gen ağaçları ve tür ağaçları. Çoklu Tür Birleştirici model, tür soyoluşlarının atalarının hesaba katılması için bir çerçeve sağlar. çok biçimlilik ve gen ağacı türü ağaç fikir ayrılığı. İşlem aynı zamanda Sansürlü Birleştirici.[4]
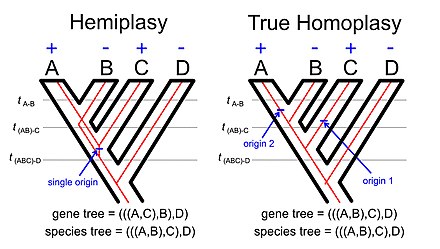
Gen ağaçları ile tür ağacı arasındaki uyumsuzluk, karakterlerin homoplastik (ayrı soylarda bağımsız olarak kazanılacak veya kaybedilecek), bu karakterler aslında tek bir kökene sahip olduğunda türler arasındaki ilişkiler göz önüne alındığında; bu fenomen denir hemiplazi.[5] Hemiplaziye odaklanan birçok çalışma, nükleotid veya amino asit ikameleri gibi genomik karakterlere odaklanmıştır,[6] indel[7][8] veya karyotipik farklılıklar[9] (ancak sonuncusunun hemiplaziye diğer birçok karakterden daha az maruz kaldığı düşünülmektedir). Bununla birlikte, fenotipik karakterler aynı zamanda hemiplazi sergileme potansiyeline de sahiptir.[10]
Gen ağaç türleri ağaç uyumu

Köklü üç taksonlu bir ağacı, önemsiz olmayan en basit filogenetik ağacı düşünürsek, üç farklı ağaç topolojisi vardır.[11] ama dört olası gen ağacı.[12] Daha az sayıda topolojiye rağmen dört farklı gen ağacının varlığı, birleşme zamanlarında farklılık gösteren topolojik olarak özdeş gen ağaçlarının olduğu gerçeğini yansıtır. Tip 1 ağaçta, A ve B türlerindeki aleller, A-B soyunu C soyundan ayıran türleşme olayından sonra birleşir. Tip 2 ağaçta, A ve B türlerindeki aleller, A-B soyunu C soyundan ayıran türleşme olayından önce birleşir (başka bir deyişle, tip 2 ağaç bir derin birleşme ağaç). Tip 1 ve tip 2 gen ağaçlarının her ikisi de tür ağacıyla uyumludur. Diğer iki gen ağacı ağaç türünden farklıdır; iki uyumsuz gen ağacı da derin birleşme ağaçlar.
Zamanların birleşmeye dağılımı aslında tüm bu ağaçlar için süreklidir. Başka bir deyişle, aynı gen ağacına sahip herhangi iki lokus için tam birleşme süresi farklı olabilir. Bununla birlikte, ağaçların birleşmenin en erken türleşme olayından önce mi yoksa sonra mı gerçekleştiğine bağlı olarak kırılması uygundur.
Birleştirici birimler cinsinden iç dal uzunluğu verildiğinde, her gen ağacının olasılığını hesaplamak basittir.[13] Diploid organizmalar için birleşik birimlerdeki dal uzunluğu, türleşme olayları arasındaki nesil sayısının etkili popülasyon büyüklüğünün iki katına bölünmesiyle elde edilir. Derin birleşme ağacının üçü de eşitlenebilir olduğundan ve bu derin birleşme ağaçlarından ikisi uyumsuz olduğundan, köklü üç taksonlu bir gen ağacının tür ağacına uygun olma olasılığının görülmesi kolaydır:
Birleştirici birimlerde dal uzunluğunun (T) ayrıca alternatif bir biçimde yazılmıştır: nesillerin sayısı (t) etkili popülasyon büyüklüğünün (Ne). Pamilo ve Nei[13] ayrıca, dört ve beş taksonun köklü ağaçları için uygunluk olasılığını ve daha büyük ağaçlar için uygunluk olasılığına ilişkin genel bir üst sınırı türetmiştir. Rosenberg[14] tüm topolojiler seti için kullanılan denklemlerle takip edildi (takson sayısı arttıkça mümkün hale gelen çok sayıda farklı filogenetik ağaç olmasına rağmen[11] takson sayısı çok sınırlı olmadığı sürece bu denklemleri kullanışsız hale getirir).
Hemiplazi olgusu, gen ağacı-tür ağaç uyumsuzluğunun altında yatan temel fikrin doğal bir uzantısıdır. Tür ağacına uymayan bazı karakterlerin dağılımını düşünürsek, yansıtabilir. homoplazi (karakterin birden çok bağımsız kökeni veya birden çok kayıpla takip edilen tek bir kökeni) veya hemiplaziyi (tür ağacıyla aynı fikirde olmayan bir gen ağacıyla ilişkili özelliğin tek bir kökeni) yansıtabilir.
Denilen fenomen eksik soy sıralaması (bilimsel literatürlerde genellikle kısaltılmış ILS[7]) fenomenle bağlantılıdır. Hemiplazinin resmini köklü bir dört takson ağacı kullanarak (sağdaki resme bakın) incelersek, A, B ve C taksonlarının ortak atası ile A ve B taksonlarının ortak atası arasındaki soy, polimorfik olmalıdır. türetilmiş özelliğe sahip alel (örneğin, bir yeri değiştirilebilir eleman yerleştirme[15]) ve atadan kalma özelliğe sahip alel. Eksik soy sıralama kavramı, nihayetinde bir veya daha fazla türleşme olayında polimorfizmlerin kalıcılığını yansıtır.
Çoklu türlerin birleşmesinin matematiksel açıklaması
Çok türlü birleşik model altındaki gen ağaçlarının olasılık yoğunluğu, çok konumlu dizi verilerini kullanarak parametre tahmini için kullanımı ile birlikte tartışılmıştır.
Varsayımlar
Tür soyoluşunun bilindiği varsayılmaktadır. Türlerin ayrışmasından sonra, hiçbir göç, melezleme veya introgresyon olmaksızın tam izolasyon da varsayılır. Lokus içindeki tüm sitelerin aynı gen ağacını paylaşması için (topoloji ve birleşme süreleri) rekombinasyon olmadığını varsayıyoruz.
Veri ve model parametreleri
Bu yöntemin modeli ve uygulaması herhangi bir tür ağacına uygulanabilir. Örnek olarak, tür ağacı harika maymunlar: insan (H), şempanze (C), goril (G) ve orangutan (O) kabul edilir. Tür ağacının (((HC) G) O)) topolojisinin bilindiği ve analizde sabitlendiği varsayılır (Şekil 1).[4] İzin Vermek veri kümesinin tamamı, nerede lokustaki dizi hizalamasını temsil eder , ile Toplamda loci.
Mevcut bir türün popülasyon büyüklüğü, yalnızca o türden bazı lokuslarda birden fazla birey örneklendiğinde dikkate alınır.
Şekil 1'deki örnek için modeldeki parametreler üç diverjans süresini içerir , ve ve nüfus büyüklüğü parametreleri insanlar için; şempanzeler için; ve , ve üç ata türü için.
Sapma zamanları ('ler) tür ağacındaki ata düğümünden şimdiki zamana kadar alan başına beklenen mutasyon sayısı ile ölçülür (Şekil 1, Rannala ve Yang, 2003).
Bu nedenle, parametreler .
Olasılığa dayalı çıkarım
Gen şecere her lokusta ağaç topolojisi ile temsil edilir ve birleşen zamanlar . Verilen parametreler olasılık dağılımı tarafından verilen model altında birleştirme süreci ile belirlenir
Veri olasılığı lokustaki gen ağacı ve birleşme süreleri (ve dolayısıyla dal uzunlukları) verildiğinde, Felsenstein'ın filogenetik olasılığıdır.[16] Lokuslar boyunca bağımsız evrim varsayımı nedeniyle,
Tarafından Bayesci çıkarım eklem temelli koşullu dağılım
Sonra arka dağıtım nın-nin tarafından verilir
burada entegrasyon, olası tüm gen ağacı topolojileri üzerinde toplamı ve her lokustaki birleşme süreleri boyunca entegrasyonu temsil eder.[17]
Gen şecere dağılımı
Ortak dağıtımı doğrudan bu bölümde türetilmiştir. Farklı türlerden iki dizi, yalnızca iki türe atası olan bir popülasyonda birleşebilir. Örneğin, H ve G dizileri, HCG veya HCGO popülasyonlarında birleşebilir, ancak H veya HC popülasyonlarında birleşemez. Farklı popülasyonlardaki birleşik süreçler farklıdır.
Her popülasyon için şecere, zaman içinde popülasyonun sonuna kadar geriye doğru izlenir. ve soyların sayısı nüfusu ve ondan ayrılan soy sayısını girmek kaydedildi. Örneğin, ve H popülasyonu için (Tablo 1).[4] Bu sürece bir sansürlü birleşen süreç çünkü bir popülasyon için birleştirme süreci, popülasyona giren tüm soylar birleşmeden önce sona erdirilebilir. Eğer nüfus oluşur bağlantısız alt ağaçlar veya soylar.
Site başına bir mutasyonu biriktirmek için geçen süre olarak tanımlanan bir zaman birimi ile, herhangi iki soy bu oranda birleşir . Bekleme süresi soyların sayısını azaltan bir sonraki birleşme olayına kadar -e üstel yoğunluğa sahiptir
Eğer , sonuncusu ile popülasyonun sonu arasında birleşik bir olayın meydana gelmeme olasılığı ; yani zaman aralığı boyunca . Bu olasılık ve 1 ise .
(Not: Zaman aralığında hiçbir olay olmaması olasılığının oranlı bir Poisson süreci için dır-dir . Burada birleşme oranı olduğunda soylar .)
Ek olarak, popülasyondaki belirli bir gen ağacı topolojisinin olasılığını türetmek için, eğer bir örneklemde bir birleşik olay meydana gelirse soylar, belirli bir soy çiftinin birleşme olasılığı .
Bu olasılıkları çarparak, ortak olasılık dağılımı popülasyondaki gen ağacı topolojisinin ve birleşme zamanlarının gibi
- .
Lokus için gen ağacının olasılığı ve birleşme zamanları, tüm popülasyonlardaki bu tür olasılıkların ürünüdür. Bu nedenle, Şekil 1'deki gen şecere,[4][18] sahibiz
Filogenetik tahmin üzerindeki etkisi
Çoklu türlerin birleşmesinin moleküler filogenetik teorisi ve pratiği için derin etkileri vardır.[2][3] Tek tek gen ağaçları tür ağacından farklı olabildiğinden, tek bir lokus için ağacı tahmin edemez ve gen ağacının tür ağacına karşılık geldiği varsayılamaz. Aslında, herhangi bir tekil gen ağacının, makul sayıda takson düşünüldüğünde, en azından bazı ilişkiler için tür ağacından farklı olacağı hemen hemen kesin olabilir. Bununla birlikte, gen ağacı-tür ağacı uyumsuzluğu, tür ağacını tahmin etmek için tek bir gen ağacının kullanılamayacağı şeklindeki basit gözlemin ötesine geçen tür ağacı tahmini teorisi ve pratiği üzerinde bir etkiye sahiptir, çünkü parametre uzayının en çok olduğu yer vardır. sık gen ağacı tür ağacıyla uyumsuzdur. Parametre uzayının bu kısmına anormal bölge[19] ve gen ağacından daha sık ortaya çıkması beklenen uyumsuz gen ağaçları. türüne uyan ağaç denir anormal gen ağaçları.
Anomali bölgesinin varlığı, çok sayıda gen ağacının basitçe tahmin edilemeyeceği ve gen ağacının en fazla sayıda geri kazanılanın tür ağacı olduğu varsayılamayacağı anlamına gelir. Elbette, tür ağacını gen ağaçlarının "demokratik oyu" ile tahmin etmek, mümkün olan son derece fazla sayıda filogenetik ağaç göz önüne alındığında, anomali bölgesi dışındaki sınırlı sayıda takson için işe yarayacaktır.[11] Bununla birlikte, anormal gen ağaçlarının varlığı, aynı zamanda, gen ağaçlarını birleştirmek için basit yöntemlerin, çoğunluk kuralı genişletilmiş ("açgözlü") konsensüs yöntemi veya parsimony (MRP) süper ağacıyla matris gösterimi gibi basit yöntemler anlamına gelir.[20][21] yaklaşım olmayacak tutarlı ağaç türlerinin tahmin edicileri[22][23] (yani yanıltıcı olacaklar). Gen ağaçlarının en az% 50'sinde bulunan grupların tutulduğu gen ağaçları için basitçe çoğunluk kurallı konsensüs ağacının oluşturulması, yeterli sayıda gen ağacı kullanıldığı sürece yanıltıcı olmayacaktır.[22] Bununla birlikte, çoğunluk kuralı konsensüs ağacının, bir dizi gen ağacı için yanlış sınıflardan kaçınmak için bu yeteneği, çözülmemiş gruplara sahip olma pahasına gelir.
Simülasyonlar, tür ağaç parametre uzayının bazı kısımlarının olduğunu göstermiştir. maksimum olasılık soyoluş tahminleri, analiz edilen veri miktarı arttıkça olasılığı artan yanlış ağaçlardır.[24] Bu önemlidir, çünkü farklı lokuslardan gelen çoklu sekans hizalamalarının, daha sonra maksimum olasılık için kullanılan tek bir büyük süper matris hizalaması oluşturmak için birleştirildiği "birleştirme yaklaşımı" Bayesian MCMC ) analizi, hem uygulaması kolaydır hem de ampirik çalışmalarda yaygın olarak kullanılmaktadır. Birleştirme yaklaşımı dolaylı olarak tüm gen ağaçlarının aynı topolojiye sahip olduğunu varsaydığından, bu bir model yanlış tanımlama durumunu temsil eder.[25] Gerçekten de, birleştirilmiş verinin maksimum olasılık analizi kullanılarak çoklu türler altında oluşturulan verilerin analizlerinin, analiz için kullanılan lokus sayısı arttıkça gerçek tür ağacında yakınsama garantisinin olmadığı artık kanıtlanmıştır.[26][27][28] (yani, maksimum olasılık birleştirme istatistiksel olarak tutarsızdır).
Çok türlü birleşik kullanarak çıkarım için yazılım
Çok tür birleşik çerçevede filogenetik tahmin için iki temel yaklaşım vardır: 1) hem maksimum olasılık hem de Bayesci yöntemler dahil olmak üzere doğrudan çok odaklı dizi hizalamalarında çalışan tam olasılık veya tam veri yöntemleri ve 2) özet kullanan özet yöntemler Özet girdi olarak tahmini gen ağaçlarını kullanan iki adımlı yöntemler ve özet girdi olarak lokuslar üzerinden havuzlanan site örüntü sayılarını kullanan SVDQuartets dahil olmak üzere orijinal sekans verilerinin bir kısmı.
| Program | Açıklama | Yöntem | Referanslar |
|---|---|---|---|
| ASTRAL | ASTRAL (Doğru Türler TRee ALgorithm), dörtlü bir yöntem kullanarak bir dizi gen ağacını özetler, birleşik dal uzunlukları ve destek değerleri (yerel arka olasılıklar) ile tür ağacının bir tahminini üretir.[29]) | Özet | Mirarab vd. (2014);[30] Zhang vd. (2018)[31] |
| ASTRID | ASTRID (Doğru Tür TRees Internode Distances) NJst yönteminin bir uzantısıdır.[32] ASTRID / NJst, bir dizi giriş gen ağacından düğümler arası mesafeleri hesaplayan özet bir tür ağacı yöntemidir. Gibi bir mesafe yöntemi komşu katılıyor veya minimum evrim daha sonra bu mesafelerden ağaç türünü tahmin etmek için kullanılır. ASTRID / NJst'nin bir eksik veri modeli altında tutarlı olmadığını unutmayın[33] | Özet | Vachaspati ve Warnow (2015)[34] |
| BPP | Çok türlü birleştirme süreci altında popülasyonlar arasında soyoluş ve ıraksama zamanlarını çıkarmak için Bayesian MCMC yazılım paketi; türlerin sınırlandırılmasına yönelik yöntemi de içerir | Tam olasılık | Yang vd. (2015);[35] Flouri vd. (2018)[36] |
| * CANAVAR | Bayesian MCMC yazılım paketi, çok türlerin bir araya gelme süreci altında popülasyonlar arasında soyoluş ve ıraksama sürelerinin çıkarılması için. Bir parçası olarak uygulandı Canavar yazılım paketi (Star BEAST olarak okunur) | Tam olasılık | Heled ve Drummond (2010)[37] |
| MP-EST | Bir dizi gen ağacını girdi olarak kabul eder ve maksimum sözde olasılık ağaç türünün tahmini | Özet | Liu vd. (2010)[38] |
| SVDquartets (uygulandı PAUP * ) | PAUP *, birçok yöntemi uygulayan genel bir filogenetik tahmin paketidir. SVDquartets, çoklu türlerin bir araya gelmesiyle oluşturulan veriler için istatistiksel olarak tutarlı olduğu gösterilen bir yöntemdir. | Site kalıbı yöntemi | Chifman ve Kubatko (2014)[39] |
Referanslar
- ^ Degnan JH, Rosenberg NA (Haziran 2009). "Gen ağacı uyumsuzluğu, filogenetik çıkarım ve çok türler birleşiyor". Ekoloji ve Evrimdeki Eğilimler. 24 (6): 332–40. doi:10.1016 / j.tree.2009.01.009. PMID 19307040.
- ^ a b Maddison WP (1997-09-01). "Tür Ağaçlarındaki Gen Ağaçları". Sistematik Biyoloji. 46 (3): 523–536. doi:10.1093 / sysbio / 46.3.523. ISSN 1063-5157.
- ^ a b Edwards SV (Ocak 2009). "Yeni ve genel bir moleküler sistematiğin teorisi mi ortaya çıkıyor?" Evrim; Uluslararası Organik Evrim Dergisi. 63 (1): 1–19. doi:10.1111 / j.1558-5646.2008.00549.x. PMID 19146594.
- ^ a b c d Rannala B, Yang Z (Ağustos 2003). "Birden çok lokustan DNA dizilerini kullanarak türlerin farklılaşma sürelerinin ve atalara ait popülasyon boyutlarının Bayes tahmini". Genetik. 164 (4): 1645–56. PMC 1462670. PMID 12930768.
- ^ Avise JC, Robinson TJ (Haziran 2008). Kubatko L (ed.). "Hemiplazi: filogenetik sözlüğünde yeni bir terim". Sistematik Biyoloji. 57 (3): 503–7. doi:10.1080/10635150802164587. PMID 18570042.
- ^ Mendes FK, Hahn Y, Hahn MW (Aralık 2016). "Gen Ağacı Uyumsuzluğu Zaman İçinde Azalan Yakınsama Modelleri Oluşturabilir". Moleküler Biyoloji ve Evrim. 33 (12): 3299–3307. doi:10.1093 / molbev / msw197. PMID 27634870.
- ^ a b Jarvis ED, Mirarab S, Aberer AJ, Li B, Houde P, Li C, ve diğerleri. (Aralık 2014). "Bütün genom analizleri, modern kuşların hayat ağacındaki ilk dalları çözüyor". Bilim. 346 (6215): 1320–31. doi:10.1126 / science.1253451. PMC 4405904. PMID 25504713.
- ^ Houde P, Braun EL, Narula N, Minjares U, Mirarab S (2019-07-06). "Indellerin Filogenetik Sinyali ve Neoavian Radyasyon". Çeşitlilik. 11 (7): 108. doi:10.3390 / d11070108.
- ^ Robinson TJ, Ruiz-Herrera A, Avise JC (Eylül 2008). "Memelilerin karyotipik filojilerinde hemiplazi ve homoplazi". Amerika Birleşik Devletleri Ulusal Bilimler Akademisi Bildirileri. 105 (38): 14477–81. doi:10.1073 / pnas.0807433105. PMC 2567171. PMID 18787123.
- ^ Guerrero RF, Hahn MW (Aralık 2018). "Filogenetik çıkarımda hemiplazi riskini ölçmek". Amerika Birleşik Devletleri Ulusal Bilimler Akademisi Bildirileri. 115 (50): 12787–12792. doi:10.1073 / pnas.1811268115. PMC 6294915. PMID 30482861.
- ^ a b c Felsenstein J (Mart 1978). "Evrim Ağaçlarının Sayısı". Sistematik Zooloji. 27 (1): 27. doi:10.2307/2412810.
- ^ Hobolth A, Christensen OF, Mailund T, Schierup MH (Şubat 2007). "İnsan, şempanze ve gorilin genomik ilişkileri ve türleşme zamanları, birleşik gizli Markov modelinden çıkarılmıştır". PLoS Genetiği. 3 (2): e7. doi:10.1371 / dergi.pgen.0030007. PMC 1802818. PMID 17319744.
- ^ a b Pamilo P, Nei M (Eylül 1988). "Gen ağaçları ve tür ağaçları arasındaki ilişkiler". Moleküler Biyoloji ve Evrim. 5 (5): 568–83. doi:10.1093 / oxfordjournals.molbev.a040517. PMID 3193878.
- ^ Rosenberg NA (Mart 2002). "Gen ağaçları ve tür ağaçlarının topolojik uyum olasılığı". Teorik Popülasyon Biyolojisi. 61 (2): 225–47. doi:10.1006 / tpbi.2001.1568. PMID 11969392.
- ^ Suh A, Smeds L, Ellegren H (Ağustos 2015). Penny D (ed.). "Neoavian Kuşların Kadim Uyarlamalı Radyasyonunda Tamamlanmamış Soy Sıralamasının Dinamikleri". PLoS Biyolojisi. 13 (8): e1002224. doi:10.1371 / journal.pbio.1002224. PMC 4540587. PMID 26284513.
- ^ Felsenstein J (1981). "DNA dizilerinden evrim ağaçları: maksimum olasılık yaklaşımı". Moleküler Evrim Dergisi. 17 (6): 368–76. doi:10.1007 / BF01734359. PMID 7288891.
- ^ Xu B, Yang Z (Aralık 2016). "Çoklu Tür Birleştirme Modeli Kapsamında Tür Ağacı Tahminindeki Zorluklar". Genetik. 204 (4): 1353–1368. doi:10.1534 / genetik.116.190173. PMC 5161269. PMID 27927902.
- ^ Yang Z (2014). Moleküler evrim: istatistiksel bir yaklaşım (İlk baskı). Oxford: Oxford University Press. s. Bölüm 9. ISBN 9780199602605. OCLC 869346345.
- ^ Degnan JH, Rosenberg NA (Mayıs 2006). Wakeley J (ed.). "Tür ağaçlarının en olası gen ağaçlarıyla uyumsuzluğu". PLoS Genetiği. 2 (5): e68. doi:10.1371 / dergi.pgen.0020068. PMC 1464820. PMID 16733550.
- ^ Baum BR (Şubat 1992). "Filogenetik çıkarımlar için veri kümelerini birleştirmenin bir yolu olarak ağaçları birleştirmek ve gen ağaçlarını birleştirmenin istenirliği". TAKSON. 41 (1): 3–10. doi:10.2307/1222480. ISSN 0040-0262.
- ^ Ragan MA (Mart 1992). "Ağaçların matris gösterimine dayalı filogenetik çıkarım". Moleküler Filogenetik ve Evrim. 1 (1): 53–58. doi:10.1016 / 1055-7903 (92) 90035-F.
- ^ a b Degnan JH, DeGiorgio M, Bryant D, Rosenberg NA (Şubat 2009). "Gen ağaçlarından tür ağaçlarının çıkarılması için fikir birliği yöntemlerinin özellikleri". Sistematik Biyoloji. 58 (1): 35–54. doi:10.1093 / sysbio / syp008. PMC 2909780. PMID 20525567.
- ^ Wang Y, Degnan JH (2011-05-02). "Gen Ağaçlarından Türleri Çıkarmak için Parsimony ile Matris Gösteriminin Performansı". Genetik ve Moleküler Biyolojide İstatistiksel Uygulamalar. 10 (1). doi:10.2202/1544-6115.1611.
- ^ Kubatko LS, Degnan JH (Şubat 2007). Collins T (ed.). "Birleştirme altında birleştirilmiş verilerden filogenetik tahminlerin tutarsızlığı". Sistematik Biyoloji. 56 (1): 17–24. doi:10.1080/10635150601146041. PMID 17366134.
- ^ Warnow T (Mayıs 2015). "Tamamlanmamış Köken Sıralama Varlığında Birleştirme Analizleri". PLoS Akımları. 7. doi:10.1371 / currents.tol.8d41ac0f13d1abedf4c4a59f5d17b1f7. PMC 4450984. PMID 26064786.
- ^ Roch S, Steel M (Mart 2015). "Hizalanmış dizi veri kümelerinin bir araya getirilmesinde olasılığa dayalı ağaç yeniden yapılandırması istatistiksel olarak tutarsız olabilir". Teorik Popülasyon Biyolojisi. 100C: 56–62. doi:10.1016 / j.tpb.2014.12.005. PMID 25545843.
- ^ Mendes FK, Hahn MW (Ocak 2018). "Anormal Bölge Yakınında Birleştirme Neden Başarısız Oluyor". Sistematik Biyoloji. 67 (1): 158–169. doi:10.1093 / sysbio / syx063. PMID 28973673.
- ^ Roch S, Nute M, Warnow T (Mart 2019). Kubatko L (ed.). "Tür Ağacı Tahmininde Uzun Dallı Çekim: Bölünmüş Olabilirliğin Tutarsızlığı ve Topoloji Temelli Özet Yöntemleri". Sistematik Biyoloji. 68 (2): 281–297. doi:10.1093 / sysbio / syy061. PMID 30247732.
- ^ Sayyari E, Mirarab S (Temmuz 2016). "Dörtlü Frekanslardan Yerel Şube Desteğinin Hızlı Birleşim Tabanlı Hesaplaması". Moleküler Biyoloji ve Evrim. 33 (7): 1654–68. doi:10.1093 / molbev / msw079. PMC 4915361. PMID 27189547.
- ^ Mirarab S, Reaz R, Bayzid MS, Zimmermann T, Swenson MS, Warnow T (Eylül 2014). "ASTRAL: genom ölçeğinde birleşen türlere dayalı ağaç tahmini". Biyoinformatik. 30 (17): i541-8. doi:10.1093 / biyoinformatik / btu462. PMC 4147915. PMID 25161245.
- ^ Zhang C, Rabiee M, Sayyari E, Mirarab S (Mayıs 2018). "ASTRAL-III: kısmen çözülmüş gen ağaçlarından polinom zaman türlerinin ağaç rekonstrüksiyonu". BMC Biyoinformatik. 19 (Ek 6): 153. doi:10.1186 / s12859-018-2129-y. PMC 5998893. PMID 29745866.
- ^ Liu, Liang; Yu, Lili (2011-10-01). "Köklenmemiş Gen Ağaçlarından Tür Ağaçlarının Tahmin Edilmesi". Sistematik Biyoloji. 60 (5): 661–667. doi:10.1093 / sysbio / syr027. ISSN 1076-836X.
- ^ Rhodes JA, Nute MG, Warnow T. (Ocak 2020). "NJst ve ASTRID, rastgele bir kayıp veri modeli altında istatistiksel olarak tutarlı değildir". arXiv: 2001.07844 https://arxiv.org/abs/2001.07844
- ^ Vachaspati, Pranjal; Warnow, Tandy (Aralık 2015). "ASTRID: Internode Mesafelerinden Doğru Tür Ağaçları". BMC Genomics. 16 (S10): S3. doi:10.1186 / 1471-2164-16-S10-S3. ISSN 1471-2164. PMC 4602181. PMID 26449326.
- ^ Yang Z (2015-10-01). "Türlerin ağaç tahmini ve türlerin sınırlandırılması için BPP programı". Güncel Zooloji. 61 (5): 854–865. doi:10.1093 / czoolo / 61.5.854. ISSN 2396-9814.
- ^ Flouri T, Jiao X, Rannala B, Yang Z (Ekim 2018). Yoder AD (ed.). "Genomik Diziler ve Çok Türlü Birleştirmeyi Kullanan BPP ile Tür Ağacı Çıkarımı". Moleküler Biyoloji ve Evrim. 35 (10): 2585–2593. doi:10.1093 / molbev / msy147. PMC 6188564. PMID 30053098.
- ^ Heled, J .; Drummond, A.J. (2010-03-01). "Çok Odaklı Verilerden Tür Ağaçlarının Bayesci Çıkarımı". Moleküler Biyoloji ve Evrim. 27 (3): 570–580. doi:10.1093 / molbev / msp274. ISSN 0737-4038. PMC 2822290. PMID 19906793.
- ^ Liu L, Yu L, Edwards SV (Ekim 2010). "Birleştirici model altında ağaç türlerini tahmin etmek için maksimum sözde olasılık yaklaşımı". BMC Evrimsel Biyoloji. 10 (1): 302. doi:10.1186/1471-2148-10-302. PMC 2976751. PMID 20937096.
- ^ Chifman J, Kubatko L (Aralık 2014). "Birleşik model altında SNP verilerinden dörtlü çıkarım". Biyoinformatik. 30 (23): 3317–24. doi:10.1093 / biyoinformatik / btu530. PMC 4296144. PMID 25104814.









































![{ displaystyle exp {- { frac {n (n-1)} { theta}} [ tau - (t_ {m} + t_ {m-1} + ldots + t_ {n + 1} )]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/2f435c3e48b6ad522af7d79165c8713ab6a42446)







![{ displaystyle prod _ {j = n + 1} ^ {m} { Büyük [} { frac {2} { theta}} exp { Büyük {} - { frac {j (j- 1)} { theta}} t_ {j} { Büyük }} { Büyük]} exp { Büyük {} - { frac {n (n-1)} { theta}} ( tau - (t_ {m} + t_ {m + 1} + ldots + t_ {n + 1})) { Büyük }}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/a7937f1541db74c1d61321eb52a49d5a32f19407)
![{ displaystyle { begin {align} f (G_ {i} mid Theta) & = [2 / theta _ {H} exp {- 6t_ {3} ^ {(H)} / theta _ {H} } exp {- 2 ( tau _ {HC} -t_ {3} ^ {(H)}) / theta _ {H} }] & {} times [2 / theta _ {C} exp {- 2t_ {2} ^ {(C)} / theta _ {C} }] & {} times [2 / theta _ {HC} exp {-6t_ {3} ^ {HC} / theta _ {HC} }] times [2 / theta _ {HC} exp {- 2t_ {2} ^ {HC} / theta _ {HC } }] & {} times [ exp {- 2 ( tau _ {HCG} - tau _ {HG} - (t_ {3} ^ {HC} + t_ {2} ^ {HC })) / theta _ {HCG} }] & {} times [2 / theta _ {HCGO} exp {- 6t_ {3} ^ {HCGO} / theta _ {HCGO} }] times [2 / theta _ {HCGO} exp {- 2t_ {2} ^ {HCGO} / theta _ {HCGO} }] end {hizalı}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/2d396905de66e7b66c05298abe56ddc5be144a53)