Topluluk parmak izi - Community fingerprinting
Topluluk parmak izi bir dizi moleküler Biyoloji hızlı bir şekilde profil oluşturmak için kullanılabilecek teknikler çeşitlilik bir mikrobiyal topluluk. Bir çevresel örnekteki tek tek hücreleri doğrudan tanımlamak veya saymak yerine, bu teknikler, bir gen mevcut. Genel olarak, her farklı gen varyantının farklı bir mikrop türünü temsil ettiği varsayılır. Topluluk parmak izini kullanan mikrobiyologlar Biyolojik çeşitliliği ölçmek veya zaman içinde topluluk yapısındaki değişiklikleri izlemek için çeşitli mikrobiyal sistemleri (örneğin deniz, tatlı su, toprak ve insan mikrobiyal toplulukları) incelemek. Yöntem, çevresel örnekleri analiz ederek genomik DNA. Bu yaklaşım şuna bir alternatif sunar: mikrobiyal kültür bu önemlidir çünkü mikropların çoğu laboratuvarda kültürlenemez.[1] Topluluk parmak izi, bireysel mikrop türlerinin tanımlanmasıyla sonuçlanmaz; bunun yerine, bir mikrobiyal topluluğun genel bir resmini sunar. Bu yöntemler artık büyük ölçüde, hedeflenen gibi yüksek verimli sıralama ile değiştiriliyor. mikrobiyom analiz (örneğin, 16s rRNA dizileme) ve metagenomik.
Kullanım
Bir parmak izi analizi, toplam DNA'nın ekstrakte edildiği çevresel bir numuneyle (örneğin deniz suyu veya toprak) başlar. (Toplam DNA, numunede bulunan tüm mikroplardan elde edilen genetik materyalin bir karışımını içerir.) Daha sonra, her mikrop türünün farklı bir gen varyantına sahip olacağı varsayımı altında, analiz için hedef olarak belirli bir gen veya DNA bölgesi seçilir (aynı zamanda a "filotip "). Bir örnekte bulunan filotipleri görselleştirmek için farklı yöntemler (aşağıya bakın) kullanılabilir. Topluluk parmak izinin amacı topluluk yapısının genel bir anlayışını kazanmak olduğundan, analiz için özellikle yararlı bir tekniktir. Zaman serisi sahadan toplanan veriler. Örneğin, mikrobiyal paterni inceleyebilirsiniz. halefiyet bir habitatta veya bir mikrobiyal topluluğun, bir kirleticinin salınması gibi çevresel bir bozulmaya tepkisi incelenebilir. Hangi bilginin istendiğine bağlı olarak, farklı genler hedeflenebilir. En yaygın olanları küçük alt birimlerdir ribozomal RNA (rRNA) genleri, örneğin 16S rRNA. Bu genler sıklıkla mikrobiyal filogenetik analizler, çalışmaları için çok iyi kurulmuş teknikler var. İlgili diğer genler, çeşitli metabolik süreçlerde anahtar olan genler olabilir.[1]
Avantajlar ve dezavantajlar
Topluluk parmak izinin avantajları, hızlı ve nispeten ucuz bir şekilde gerçekleştirilebilmesi ve analizlerin aynı anda çok sayıda örneği barındırabilmesidir.[2][3] Bu özellikler, topluluk parmak izini özellikle mikrobiyal topluluklardaki zaman içindeki değişiklikleri izlemek için yararlı hale getirir. Ayrıca, parmak izi teknikleri, birinin sahip olmasını gerektirmez. Önsel Bir örnekteki organizmalar için dizi verileri: Topluluk parmak izinin bir dezavantajı, nicel değil, büyük ölçüde nitel verilerle sonuçlanmasıdır.[4] Niteliksel verileri kullanırken, farklı çalışmalarda veya farklı araştırmacılar arasında gözlemlenen modelleri karşılaştırmak zor olabilir. Ayrıca, topluluk parmak izi, çevresel bir örnekteki taksonları doğrudan tanımlamaz, ancak veriler belirli tekniklerden (örn. DGGE ) kimlik istenirse daha fazla analiz edilebilir. Bazı yazarlar, belirli parmak izi yöntemleri için sonuçların yeniden üretilebilirliğinin zayıf olduğuna işaret etmektedir.[3] diğer yazarlar, bolluk tahminlerinin yanlışlığını ve bazı tekniklerin nadir taksonların varlığını yakalayamamasını eleştirmişlerdir.[5] Örneğin, DGGE yönteminin bir bakteri topluluğunun% 0,5 - 1'inden daha azını oluşturan mikropları tespit etmesi zordur.[6]
Teknikler
Bu bölümde, topluluk parmak izi almanın üç yöntemi anlatılmaktadır.
Terminal kısıtlama parça uzunluğu polimorfizmi (T-RFLP)
Terminal kısıtlama parça uzunluğu polimorfizmi (T-RFLP) kullanan bir yöntemdir floresan - bir topluluk parmak izi oluşturmak için etiketlenmiş DNA parçaları.[2][7] Bu bölüm T-RFLP'nin kısa bir açıklamasını sunar belirli topluluk parmak izi bağlamında. Daha ayrıntılı bir açıklama için bkz. T-RFLP makale.
Prosedür
T-RFLP'yi gerçekleştirmek için (Şekil 1), amplifiye edilecek bir hedef gen (örneğin 16S rRNA geni) seçilmelidir. PCR. En az bir astar PCR reaksiyonunda kullanılan, floresan olarak etiketlenir. 5´ son. PCR amplifikasyonundan sonra, kopyalanan her DNA segmenti floresan etiketi taşır. Sonraki, Kısıtlama enzimleri amplifiye DNA'yı spesifik olarak kesmek için kullanılır tanıma siteleri. Altta yatan varsayım, numunedeki her bir mikropun hedef gende farklı bir diziye sahip olacağıdır, bu nedenle bir kısıtlama enzimi, her mikrobun DNA'sını farklı bir yerde kesecektir. (Her farklı kısıtlama sitesinin tek bir kısıtlama sitesini temsil ettiği kabul edilir. operasyonel taksonomik birim [OTU]). Böylece enzim, numunede bulunan her bir mikrop türü için bir parça uzunluğu üretecektir. Sindirimin sonucu, her biri bir uçta floresan olarak etiketlenmiş farklı uzunluklarda bir dizi kısıtlama fragmanıdır. Bunlar, PCR primerinin eklendiği uçta etiketlendikleri için "terminal fragmanlar" olarak bilinirler. (Etiketlenmemiş uçlar son analize kaydedilmez.) Daha sonra, parçalar boyuta göre ayrılır. jel veya kapiler Elektroforez. Lazer algılama, terminal parçalarının boyutunu ve floresan yoğunluğu modellerini yakalar. Bilinen boyut ve floresan DNA standartları, analize referans olarak dahil edilmiştir. Floresans için minimum bir eşik ayarlayarak, arka plan gürültüsü hariç tutulacaktır.[4]
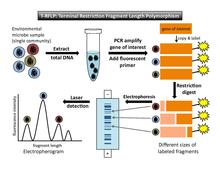
Veri çıkışı ve yorumlama
Lazer algılama adımının çıktısı bir elektroferogram yatay eksende parça uzunluğu ve dikey eksende floresan yoğunluğu ile bir dizi zirveyi gösterir (Şekil 1). İkinci bir çıktı[4] parçaların geçiş zamanını, boyutlarını listeleyen bir veri tablosudur. baz çiftleri Her bir tepenin yüksekliği ve her bir tepenin altındaki alan.
T-RFLP'nin teorik temeli, yatay eksen boyunca farklı konumlardaki piklerin farklı organizma türlerini (veya OTU'ları) temsil ettiğini varsayar. Her bir floresans yoğunluğu zirvesinin altındaki alan, topluluktaki her filotipin göreceli bolluğunun bir temsilcisidir. Ancak, bir takım uyarılar dikkate alınmalıdır. Farklı organizma türleri, ilgilenilen gende bir kısıtlama bölgesini paylaşabilir; bu durumda, bu organizmalar elektroferogramda farklı zirveler olarak ayırt edilmeyecektir. Ayrıca, bir tepe altındaki alan, mutlak bolluktan ziyade göreli bolluğu temsil eder ve bolluk ölçümü ve PCR amplifikasyonunda önyargılar vardır. Örneğin, orijinal toplam DNA örneğinde kıt olan organizmalar, son analizde tespit edilmek için yeterince amplifiye edilmeyecektir.[7] Bu, topluluk çeşitliliğinin hafife alınmasına yol açar. Liu et al.[7] "türler arasındaki gen kopya sayısındaki farklılıklar ve hücre lizizi, DNA ekstraksiyonu ve PCR amplifikasyonu sırasında ortaya çıkan önyargılar" da dahil olmak üzere sonuçları bozabilecek diğer olası faktörlerden bahsedin (s. 4521). Detaylı teknik bilgi arayanlar için Marsh[2] T-RFLP sürecinin her adımında kullanılabilecek olası önyargıların bir kataloğunu sağlar.
Avantajlar ve dezavantajlar
T-RFLP'nin en büyük avantajları, hızlı olması ve birçok örneği kolayca barındırabilmesidir.[2] Ayrıca, görsel çıktı, topluluk yapısı modellerinin farklı örnekler arasında karşılaştırılmasını kolaylaştırır. Bir dezavantaj, yukarıdaki uyarılar dikkate alınarak yorumlanması gereken veri çıktısının geniş, niteliksel doğasıdır. Ayrıca, bir numunedeki mikropların doğrudan tanımlanması T-RFLP ile mümkün değildir.
Başvurular
Disayathanoowat et al.[8] Tayland'daki iki bal arısı türünde mikrobiyal bağırsak topluluğunu veya mikrobiyomu değerlendirmek için T-RFLP kullandı. İki türün farklı mikrobiyal toplulukları barındırdığını ve mikrobiyomun arıların yaşamları boyunca değiştiğini buldular.
Joo et al.[9] T-RFLP'yi fitoplankton izleme yöntemi olarak test etti. Yazarlar, bir zaman serisinde rezervuarlardan çevresel su örnekleri topladılar. Örneklerin bilinen terminal kısıtlama fragmanları (kültürlerden oluşturulmuş bir veritabanından) ile karşılaştırılmasından sonra, T-RFLP'nin bir izleme tekniği olarak etkili bir şekilde kullanılabileceği sonucuna vardılar. değişiklikler fitoplankton topluluğunda. Bununla birlikte, çeşitlilik ve bolluk tahminleri, diğer yöntemlerle bulunanlardan daha az doğru bulundu.
Denatüre edici gradyan jel elektroforezi (DGGE)
Prosedür
Denatüre edici gradyan jel elektroforezi (DGGE) ayıran mikrobiyal bir parmak izi tekniğidir amplikonlar sıra özelliklerine göre kabaca aynı boyutta[1] (Şekil 2). Bu özellikler, DNA'nın denatüre olduğu eşiği belirler. DGGE jeli bir gradyan DNA denatürantı (bir üre ve formamid karışımı) veya bir doğrusal sıcaklık gradyanı kullanır.[10] Parça erime noktasına (yeterli denatürant eşiğine) ulaştığında hareket etmeyi durdurur. Bunun nedeni, kısmen erimiş çift sarmallı bir DNA'nın artık jelden geçememesidir.[11] Özel olarak bir GC kıskacı (yüksek GC içeriğine sahip yaklaşık 40 baz) kullanılır. astar demirlemek PCR bir kez bir araya gelen parçalar denatüre.[12]
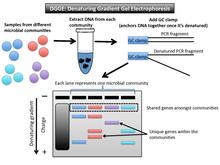
Veri çıkışı ve yorumlama
Jel üzerindeki her şerit, bir mikrobiyal topluluğu temsil eder. Örnekler arasında paylaşılan bantlar aynı boyuttadır ve jel üzerinde kabaca aynı pozisyondadır. Mikrobiyal topluluk örnekleri arasında paylaşılmayan gen varyantları, diğerleriyle yatay olarak eşleşmez. Örneğin, ilgilenilen gen, teknik ilk açıklandığı zaman olduğu gibi 16S rRNA ise, PCR ile büyütülmüş fragmanlar aynı dikey konumda olacaktır çünkü hepsi kabaca aynı boyuttadır.[13] Başka bir hedef gen, uzunluk açısından daha büyük varyasyona sahip olabilir, ancak denatürant gradyan, numuneler arasında daha fazla ayrım yapmak için ikinci bir eleman (erime noktasına sahip) kullanır. DGGE jeli aynı büyüklükteki genleri baz dizisine göre ayıracaktır.
Bu teknik, mikrobiyal toplulukların taksonomik bileşimde ne ölçüde aynı veya farklı olduğunu gösterir. Jel üzerinde farklı bir konumdaki her bant, farklı bir filotipi temsil eder (bir filogenetik işaretleyici genin benzersiz bir dizisi).[1] Mikrobiyal topluluklar için bu yöntem, birçok ayrı 16S rRNA dizisini profiller.[14] Farklı yatay konumlardaki bantların sayısı, o örnekteki biyolojik çeşitlilik düzeyini tahmin etmek ve filogenetik ilişkiyi çıkarmak için kullanılabilir.[10] Filogenetik ilişki hakkında daha fazla bilgi edinmek için, bu bantları jelden çıkarabilir ve ardından bunları sıralayabiliriz.
Avantajlar ve dezavantajlar
Denatüre edici profilin kullanımı, benzer boyutlardaki DNA fragmanlarını ayırmanın bir yolu olarak hizmet eder. 16S rRNA geninin, bakteriyel filumlar arasında boyut olarak çok fazla değişiklik göstermemesi nedeniyle bu, mikrobiyal çeşitliliğin değerlendirilmesinde faydalıdır. DGGE jeli, bir mikrobiyal numunedeki biyoçeşitliliğe hızlı bir bakış sağlar ve ilgilenilen bantların sıralanması seçeneğini engellemez. Bu yöntem, mikropların laboratuvarda kültürlenmesini gerektirmez ve hibridizasyon yöntemleri için problar tasarlamak için gerekli herhangi bir sekans verisi gerektirmez.[10] Ana dezavantaj, bunun biyolojik çeşitliliğin nitel bir değerlendirmesi olması ve filogenetik ilişki hakkında çıkarımlar yapmak için genlerin sıralanması gerektiğidir. Diğer bir dezavantaj, GC kelepçesinin her sentezlendiğinde değişken olabilmesidir. Bu, aynı 16S rRNA dizisi için farklı DGGE profilleri için potansiyele yol açar.[12]
Başvurular
Stephen et al.[15] topraktaki Proteobacteria'nın hızlı bir analizi için DGGE'yi kullandı. 36 yıl boyunca çeşitli pH değerlerinde tutulan topraktan çevresel örneklerinde mikrobiyal çeşitliliğin ilk değerlendirmesini yaptılar. Doğal popülasyonlar hakkında daha fazla ayrıntı elde etmek için DNA parçalarını araştırarak DGGE ve hibridizasyon tekniklerini birleştirdiler. Bu çalışmada, hepsi ototrofik β-proteobakteriyel amonyak oksitleyiciler olan, yakından ilişkili bir grup bakteri türüne bakıyorlardı. 16S rDNA örnekleri, bir jel üzerinde tükendiğinde belirsiz örtüşen bantlar vermiştir. Belirsiz örtüşen bantlar, numunelerdeki farklı genotiplerin göreli bolluğu hakkında bilgi veren, kümeye özgü radyo-etiketli problarla ayrıldı.
Koğuş et al.[14] Yellowstone kaplıcalarında, aerobik kemoorganotrofik popülasyonların 16S rRNA gen segmentlerinin DGGE analizini kullanarak siyanobakteriyel mat topluluklarını inceledi. DGGE, çeşitliliğe yeni bir bakış açısı kazandıran topluluğun profilini çıkarmalarına izin verdi. İlgilenilen bantları saflaştırarak ve sıralayarak karakterize ettiler ve daha önce mevcut olduğu bilinmeyen birçok soy tespit ettiler.
(Otomatik) ribozomal intergenik boşluk analizi (ARISA)
Prosedür
(Otomatik) ribozomal intergenik boşluk analizi veya (A) RISA (Şekil 3), prokaryotik DNA'nın iki yüksek düzeyde korunmuş gen, 16SrRNA geni ve 23SrRNA geni için kodlaması olgusundan yararlanır. Bunlar, rRNA operonundaki küçük ve büyük alt birim genleri kodlar. Bu iki gen arasında, dahili bir kopyalanmış boşluk (ITS) bölgesi vardır. Proteinleri kodlamadığından, oldukça değişken bir nükleotid dizisi ve uzunluğudur. DNA bir topluluktan izole edildikten sonra, PCR bu ara bölgeyi büyütür. Parçalar, bir jel (RISA) üzerinde çalıştırılabilir veya flüoresan primerler, bir üzerinde farklı fragman uzunluklarının bolluğu içinde piklere çevrilebilir. elektroferogram (ARISA).[16][17]
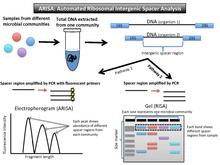
Veri çıkışı ve yorumlama
Değişken kodlamayan bölgeler nedeniyle, RISA için çıktı, farklı bantlama desenlerine sahip bir jeldir ve ARISA için çıktı, farklı tepelere sahip bir elektroferogramdır (T-RFLP'ye benzer).
Floresan etiketli primerlerin parlaklığı, bu bakteri türünün toplulukta ne kadar yaygın olduğu ile ilişkilidir. Jel üzerindeki bantlanma paterni, topluluğa özgü bir profil olarak yorumlanabilir. Her DNA bandı veya zirvesi, o organizmanın en az bir temsilcisini gösterir.[16] RISA'da, uzunluk bakımından eşleşmeyen jel üzerindeki bantlar, topluluktaki farklı organizmaları temsil eder çünkü bunlar, iki yüksek düzeyde korunmuş gen arasında farklı ara bölgelere sahiptir. Elektroferogram, numunedeki bu ara bölgenin göreceli bolluğuyla ilişkili zirveleri gösterir.
Avantajlar ve dezavantajlar
ARISA, mikrobiyal çeşitliliği tespit etmede daha yüksek çözünürlüğe sahip olabilir. T-RFLP.[18] Bu parmak izi yöntemi, mikrobiyal çeşitliliği tahmin etmek için hızlı ve hassas bir yöntemdir. Gözlenen uzunluk heterojeniteleri, kültürlenebilir organizmalarla örtüşme açısından veri tabanlarıyla karşılaştırılabilir.[16] Filogenetik gruplarla ilgili soruları yanıtlamak için filum düzeyinde oligonükleotid primerleri tasarlanabilir.[16] ARISA'nın bir dezavantajı, tek bir organizmanın topluluk profiline birden fazla zirveye katkıda bulunabilmesidir. İlişkili olmayan organizmalar da benzer boşluk uzunluklarına sahip olabilir, bu da topluluk çeşitliliğinin olduğundan az hesaplanmasına yol açar. Bu önyargılar nedeniyle, araştırmacılar ortalama bir değerlendirme elde etmek için genellikle bu yöntemi her topluluktan birden fazla örnek üzerinde kullanırlar.
Başvurular
Ranjard et al.[17] RISA'nın kullanılabileceği çeşitli çalışma türlerini tartışır. Bu tekniği antibiyotik tedavisi, cıva stresi ve ormansızlaşma gibi rahatsızlıkların ardından bakteri topluluklarının parmak izini almak için kullanan birkaç çalışmadan alıntı yapıyorlar. Ayrıca, mikrobiyal ekolojinin tam olarak araştırılmayı bekleyen bir yönü olan mantar topluluklarını karakterize etmek için ARISA'nın başarılı bir şekilde kullanıldığını da gösterdiler.
Schloss et al.[19] çevresel değişkenleri inceleyen ve bunları bir kompost yığınının mikrobiyal ekolojisindeki değişikliklerle ilişkilendiren bir çalışma yürüttü. Topluluk yapısının profilini çıkarmak ve kompostlama sürecindeki aşamalar boyunca mikrobiyal ardışıklığa bakmak için ARISA'yı kullandılar. Sürecin farklı aşamalarında topluluk üyelerini tanımlamak için DNA örnekleri aldılar ve 16S rRNA genini sıraladılar. Daha sonra topluluk çapındaki değişikliklere bakmak için ARISA'yı kullandılar ve daha sonra bu mikrobiyal topluluk üyelerini tanımlamak için 16S rRNA gen dizisini en yaygın ARISA parçalarıyla eşleştirdiler.
Referanslar
- ^ a b c d Madigan, M.T .; J.M. Martinko; P.V. Dunlap; D.P. Clark (2009). Brock Mikroorganizmaların Biyolojisi (12. baskı). San Francisco, CA: Pearson Education Inc.
- ^ a b c d Marsh, T.L. (2005). "Son kısıtlama parça uzunluğu polimorfizmi ile kültürden bağımsız mikrobiyal topluluk analizi". Enzimolojide Yöntemler. 397: 308–329. doi:10.1016 / s0076-6879 (05) 97018-3. ISBN 9780121828028. PMID 16260299.
- ^ a b Grant, A .; L.A. Ogilvie (2004). "Bu mikropu adlandırın: mikrobiyal topluluk yapısının parmak izlerindeki bireysel parçalardan sorumlu olan taksonların hızlı tanımlanması". Moleküler Ekoloji Notları. 4 (1): 133–136. doi:10.1111 / j.1471-8286.2004.00590.x.
- ^ a b c Osborn, A.M .; E.R.B. Moore; K.N. Timmis (2000). "Mikrobiyal topluluk yapısı ve dinamiklerinin incelenmesi için son sınırlama parça uzunluğu polimorfizminin (T-RFLP) analizi". Çevresel Mikrobiyoloji. 2 (1): 39–50. doi:10.1046 / j.1462-2920.2000.00081.x. PMID 11243261.
- ^ Bent, S.J .; J.D. Pierson; L.J. Forney (2007). "Tür zenginliğini mikrobiyal topluluk parmak izlerine göre ölçmek: İmparatorun kıyafeti yok". Uygulamalı ve Çevresel Mikrobiyoloji. 73 (7): 2399–2401. doi:10.1128 / aem.02383-06. PMC 1855686. PMID 17403942.
- ^ Kan, J .; K. Wang; F. Chen (2006). "Gradient jel elektroforezi (DGGE) denatüre ederek analiz edilen nehir ağzı bakterioplankton topluluğunun zamansal varyasyonu ve tespit sınırı". Sucul Mikrobiyal Ekoloji. 42: 7–18. doi:10.3354 / ame042007.
- ^ a b c Liu, W.-T .; T. L. Marsh; H. Cheng; L.J. Forney (1997). "16S rRNA'yı kodlayan genlerin terminal kısıtlama parça uzunluğu polimorfizmlerini belirleyerek mikrobiyal çeşitliliğin karakterizasyonu". Uygulamalı ve Çevresel Mikrobiyoloji. 63 (11): 4517–4522. doi:10.1128 / AEM.63.11.4516-4522.1997. PMC 168770. PMID 9361437.
- ^ Disayathanoowat, T .; J.P.W. Genç; T. Helgason; P. Chantawannakul (2012). "Bağırsaklardaki bakteri topluluklarının T-RFLP analizi Apis mellifera ve Apis cerana Tayland'daki bal arıları ". FEMS Mikrobiyoloji Ekolojisi. 79 (2): 273–281. doi:10.1111 / j.1574-6941.2011.01216.x. PMID 22092273.
- ^ Joo, S .; S.-R. Lee; S. Park (2010). "Uç kısıtlama parça uzunluğu polimorfizmi (T-RFLP) kullanılarak fitoplankton topluluk yapısının izlenmesi". Mikrobiyolojik Yöntemler Dergisi. 81 (1): 61–68. doi:10.1016 / j.mimet.2010.01.025. PMID 20138925.
- ^ a b c Muyzer, G. (1999). "DGGE / TGGE, doğal ekosistemlerden genlerin belirlenmesi için bir yöntem". Mikrobiyolojide Güncel Görüş. 2 (3): 317–322. doi:10.1016 / s1369-5274 (99) 80055-1. PMID 10383868.
- ^ Fischer, S.G .; L.S. Lerman (1983). "Tek baz çifti ikameleri ile farklılık gösteren DNA fragmanları, 16S rRN'yi kodlayan polimeraz zincir reaksiyonu ile büyütülmüş genlerin denatüre edici elektroforez analizinde ayrılır". PNAS. 80 (6): 1579–1583. doi:10.1073 / pnas.80.6.1579. PMC 393645. PMID 6220406.
- ^ a b Rettedal, E.A .; S. Clay; V. S. Brözel (2010). "GC-clamp primer grupları, denatüre edici gradyan jel elektroforez profillerini etkileyen değişken GC klempli 16S rRNA gen amplikon havuzları verir". FEMS Mikrobiyoloji Mektupları. 312 (1): 55–62. doi:10.1111 / j.1574-6968.2010.02097.x. PMID 20831594.
- ^ Muyzer, G .; E.C. De Waal; A.G. Uitterlinden (1993). "16S rRN için kodlayan polimeraz zincir reaksiyonu ile güçlendirilmiş genlerin denatüre edici gradyan jel elektroforez analizi ile karmaşık mikrobiyal popülasyonların profilinin çıkarılması". Uygulamalı ve Çevresel Mikrobiyoloji. 59 (3): 695–700. doi:10.1128 / AEM.59.3.695-700.1993.
- ^ a b Ward, D.M .; M. J. Ferris; S. C. Nold; M. M. Bateson (1998). "Kaplıca siyanobakteriyel mat toplulukları içindeki mikrobiyal biyoçeşitliliğin doğal görünümü". Mikrobiyoloji ve Moleküler Biyoloji İncelemeleri. 62 (4): 1353–1370. doi:10.1128 / MMBR.62.4.1353-1370.1998. PMC 98949. PMID 9841675.
- ^ Stephen, J.R .; G. A. Kowalchuk; M-A. V. Bruns; A. E. McCaig; C. J. Phillips; T. M. Embley; J. I. Prosse (1998). "Gradyen jel elektroforez analizi ve hiyerarşik filogenetik sondalama denatüre ederek topraktaki β-alt grubu Proteobakteriyel amonyak oksitleyici popülasyonlarının analizi". Uygulamalı ve Çevresel Mikrobiyoloji. 64 (8): 2958–2965. doi:10.1128 / AEM.64.8.2958-2965.1998. PMC 106799. PMID 9687457.
- ^ a b c d Fisher, M.M .; E.W. Triplett (1999). "Mikrobiyal çeşitliliğin ribozomal intergenik ayırıcı analizi için otomatik yaklaşım ve tatlı su bakteri topluluklarına uygulanması". Uygulamalı ve Çevresel Mikrobiyoloji. 65 (10): 4630–4636. doi:10.1128 / AEM.65.10.4630-4636.1999.
- ^ a b Ranjard, L .; F. Poly; J.-C. Lata; C. Mougel; J. Thioulouse; S. Nazaret (2001). "Otomatik ribozomal intergenik aralayıcı analiz parmak izleri ile bakteriyel ve fungal toprak topluluklarının karakterizasyonu: biyolojik ve metodolojik değişkenlik". Uygulamalı ve Çevresel Mikrobiyoloji. 67 (10): 4479–4487. doi:10.1128 / aem.67.10.4479-4487.2001. PMC 93193. PMID 11571146.
- ^ Danovaro, R .; G. M. Luna; A. Dell’Anno; B. Pietrangeli (2006). "Sucul ortamlarda bakteri çeşitliliğinin belirlenmesi için iki parmak izi tekniğinin karşılaştırılması, terminal kısıtlama parça uzunluğu polimorfizmi ve otomatik ribozomal intergenik aralayıcı analizi". Uygulamalı ve Çevresel Mikrobiyoloji. 72 (9): 5982–5989. doi:10.1128 / aem.01361-06. PMC 1563681. PMID 16957219.
- ^ Schloss, P.D .; A.G. Hay; D.B. Wilson; L.P. Walker (2003). "Kompostinin ilk aşamalarında bakteri topluluğu parmak izlerindeki zamansal değişiklikleri izleme". FEMS Mikrobiyoloji Ekolojisi. 46 (1): 1–9. doi:10.1016 / s0168-6496 (03) 00153-3. PMID 19719577.
