Haplogrup L0 (mtDNA) - Haplogroup L0 (mtDNA)
| Haplogrup L0 | |
|---|---|
| Muhtemel menşe zamanı | 130 ila 200 ka[1][2] |
| Olası menşe yeri | Güney Afrika veya Güney Doğu Afrika |
| Ata | L (Mitokondriyal Havva ) |
| Torunları | L0a'b'f'k, L0d |
| Mutasyonları tanımlama | 263 !, 1048, 3516A, 5442, 6185, 9042, 9347, 10589, 12007, 12720[3] |
Haplogrup L0 bir insan mitokondriyal DNA (mtDNA) haplogrubu.
Menşei

L0, aşağıdaki iki daldan biridir. en son ortak ata (MRCA) paylaşılan insan anne soyu için. Haplogrup beş ana daldan oluşur (L0a, L0b, L0d, L0f, L0k). Bunlardan dördü başlangıçta L1 alt kanatları, L1a, L1d, L1f ve L1k olarak sınıflandırıldı.
2014 yılında, 2.330 yaşındaki bir erkek avcının iskeletinin antik DNA analizi Güney Afrika örneğin L0d2c1c mtDNA alt sınıfına ait olduğunu buldu. Bu anne haplogrubu, bugün en çok yerli halkın bir alt grubu olan Ju ile ilişkilidir. San insanlar Bu da bölgedeki nüfus devamlılığına işaret ediyor.[4] 2016 yılında Geç Demir Çağı Kuzey Botsvana'daki Tuli bölgesinden kurutulmuş mumyanın da L0 haplogrubuna ait olduğu bulundu.[5]
| MRCA (mtDNA) |
| ||||||||||||||||||||||||||||||||||||
Dağıtım

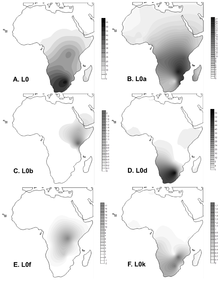
L0 en yaygın olarak şurada bulunur: Sahra-altı Afrika. En yüksek frekansına ulaşır. Khoisan ortalama% 73 olan insanlar.[6] En yüksek frekanslardan bazıları:[7] Namibya (! Xun ) 79%, Güney Afrika (Khwe /! Xun)% 83 ve Botsvana (! Kung ) 100%.
Haplogrup L0d Khoisan grupları arasında bulunur Güney Afrika (L0k'yi izleyen) daha Sanid olmakla birlikte, Khoid tarafına daha yakın, ancak büyük ölçüde bir bütün olarak Khoisan ile sınırlıdır.[7][8][9][10] L0d aynı zamanda Renkli Güney Afrika'nın nüfusu ve frekansları% 60 arasında değişiyor[11] % 71'e.[10] Bu, Khoisan halkının Güney Afrika'nın Renkli nüfusuna büyük anne katkısını göstermektedir.
Haplogruplar L0k (L0d'yi takiben) daha fazla Khoid olmakla birlikte, Khoisan gruplarında Sanid tarafına daha yakın olan ikinci en yaygın haplogruptur, ancak büyük ölçüde bir bütün olarak Khoisan ile sınırlıdır.[7][8][9][10] Khoisan ile ilişkili L0d haplogrubu, Güney Afrika'nın Renkli popülasyonunda yüksek frekanslarda bulunmasına rağmen, L0k, büyük Renkli birey gruplarını içeren iki çalışmada gözlenmedi.[10][11]
Haplogrup L0f nispeten küçük frekanslarda bulunur Tanzanya, Doğu Afrika arasında Sandawe insanlar Khoisan'dan daha yaşlı olan Tanzanya'nın.
Haplogrup L0a en çok Güneydoğu Afrika popülasyonlarında yaygındır (% 25 Mozambik ).[6]Arasında Gineliler % 1 ile% 5 arasında bir frekansa sahiptir. Balanta yaklaşık% 11 oranında artan sıklık gösteren grup. Haplogroup L0a, yaklaşık 33.000 yıllık bir Paleolitik zaman derinliğine sahiptir ve muhtemelen 10.000 ila 4.000 yıl önce Gine'ye ulaşmıştır. Aynı zamanda sıklıkla Mbuti ve Biaka Pigmeler. L0a, yaklaşık% 25'lik bir sıklıkta bulunur. Hadramawt (Yemen ).[12]
Haplogrup L0b Etiyopya.
İlaç ve hastalık etkileşimleri
İlaç verilen hastalarda Stavudin tedavi etmek HIV Haplogroup L0a2, daha yüksek olasılıkla ilişkilidir. periferik nöropati bir yan etki olarak.[13]
Alt kanat
Ağaç
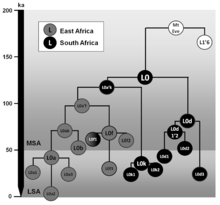
Haplogroup L0 alt kanatlarının bu filogenetik ağacı, Mannis van Oven ve Manfred Kayser'in yazısına dayanmaktadır. Küresel insan mitokondriyal DNA varyasyonunun kapsamlı filogenetik ağacı güncellendi[3] ve sonraki yayınlanmış araştırmalar.
- En Son Ortak Atalar (MRCA)
- L0
- L0d
- L0d3
- L0d1'2
- L0d1
- L0d1a
- L0d1b
- L0d1c
- L0d1c1
- L0d2
- L0d2a'b
- L0d2a
- L0d2a1
- L0d2b
- L0d2a
- L0d2c
- L0d2c1c[14]
- L0d2a'b
- L0d1
- L0a'b'f'k
- L0k
- L0k1
- L0k2
- L0a'b'f
- L0f
- L0f1
- L0f2
- L0f2a
- L0f2b
- L0a'b
- L0a
- L0a1
- L0a1a
- L0a1a2
- L0a1b
- L0a1b1
- L0a1b1a
- L0a1b2
- L0a1b1
- L0a1c
- L0a1d
- L0a1a
- L0a2
- L0a2a
- L0a2a1
- L0a2a1a
- L0a2a1a1
- L0a2a1a2
- L0a2a1a
- L0a2a2
- L0a2a2a
- L0a2a1
- L0a2b
- L0a2ba
- L0a2c
- L0a2d
- L0a2a
- L0a3
- L0a4
- L0a1
- L0b
- L0a
- L0f
- L0k
- L0d
- L0
Ayrıca bakınız
- Şecere DNA testi
- Genetik şecere
- İnsan mitokondriyal genetiği
- Popülasyon genetiği
- İnsan mitokondriyal DNA haplogrupları
Filogenetik ağaç insan mitokondriyal DNA (mtDNA) haplogrupları | |||||||||||||||||||||||||||||||||||||||
| Mitokondriyal Havva (L ) | |||||||||||||||||||||||||||||||||||||||
| L0 | L1–6 | ||||||||||||||||||||||||||||||||||||||
| L1 | L2 | L3 | L4 | L5 | L6 | ||||||||||||||||||||||||||||||||||
| M | N | ||||||||||||||||||||||||||||||||||||||
| CZ | D | E | G | Q | Ö | Bir | S | R | ben | W | X | Y | |||||||||||||||||||||||||||
| C | Z | B | F | R0 | JT öncesi | P | U | ||||||||||||||||||||||||||||||||
| HV | JT | K | |||||||||||||||||||||||||||||||||||||
| H | V | J | T | ||||||||||||||||||||||||||||||||||||
Referanslar
- ^ nokta tahmini 168.5 ka (136.3–201.1 ka % 95 CI ) göre Heinz, Tanja; et al. (2017). "Afrika insan mitokondriyal DNA ağacının güncellenmesi: Adli tıp ve popülasyon genetiğiyle alaka". Adli Bilimler Uluslararası: Genetik. 27: 156–159. doi:10.1016 / j.fsigen.2016.12.016. PMID 28086175. (tablo 2) .150 ka önerildi:Soares, Pedro; Ermini, Luca; Thomson, Noel; Mormina, Maru; Rito, Teresa; Röhl, Arne; Salas, Antonio; Oppenheimer, Stephen; MacAulay Vincent (2009). "Seçimi Arındırmak için Düzeltme: Geliştirilmiş İnsan Mitokondriyal Moleküler Saati". Amerikan İnsan Genetiği Dergisi. 84 (6): 740–59. doi:10.1016 / j.ajhg.2009.05.001. PMC 2694979. PMID 19500773..
- ^ Yaş tahminleri (ka, köşeli parantez içinde% 95 CI): ML tam-mtDNA yaş tahmini: 128.2 [% 95 CI: 107.9-148.9], ρ tam-mtDNA yaş tahmini: 121.3 [99.2; 143.7], ρ eşanlamlı yaş tahmini ( ka): 131.0 [97.8; 164.2]: Rito T, Richards MB, Fernandes V, Alshamali F, Cerny V, Pereira L, Soares P., "Afrika'daki ilk modern insan yayılımı", PLoS One 13 Kasım 2013; 8 (11): e80031. doi: 10.1371 / journal.pone.0080031.
- ^ a b Van Oven, Mannis; Kayser, Manfred (2009). "Küresel insan mitokondriyal DNA varyasyonunun kapsamlı filogenetik ağacı güncellendi". İnsan Mutasyonu. 30 (2): E386–94. doi:10.1002 / humu.20921. PMID 18853457. S2CID 27566749.
- ^ Alan G. Morris; Anja Heinze; Eva K.F. Chan; Andrew B. Smith; Vanessa M. Hayes (2014). "Pastoral Öncesi Bir Güney Afrikalı'dan İlk Antik Mitokondriyal İnsan Genomu". Genom Biyolojisi ve Evrim. 6 (10): 2647–53. doi:10.1093 / gbe / evu202. PMC 4224329. PMID 25212860. Alındı 17 Nisan 2016.
- ^ Frank J. Rühli; Maryna Steyn; Morongwa N. Mosothwane; Lena Öhrström; Molebogeng K. Bodiba; Abigail Bouwman (Ocak – Şubat 2016). "Botsvana, Tuli Bloğu'ndan Geç Demir Çağı mumyasının radyolojik ve genetik analizi" (PDF). Güney Afrika Bilim Dergisi. 112 (1/2). Arşivlenen orijinal (PDF) 21 Haziran 2016'da. Alındı 26 Nisan 2016.
- ^ a b Rosa, Alexandra; Brehm, Antonio; Kivisild, Toomas; Metspalu, Ene; Villems Richard (2004). "Batı Afrika Ginelilerinin MtDNA Profili: Senegambia Bölgesinin Daha İyi Anlaşılmasına Doğru". İnsan Genetiği Yıllıkları. 68 (4): 340–52. doi:10.1046 / j.1529-8817.2004.00100.x. PMID 15225159. S2CID 15391342.
- ^ a b c Tishkoff, S. A .; Gonder, M. K .; Henn, B. M .; Mortensen, H .; Knight, A .; Gignoux, C .; Fernandopulle, N .; Lema, G .; Nyambo, T.B. (2007). "MtDNA ve Y Kromozomu Genetik Varyasyonundan Çıkarılan Afrika'nın Tıklayarak Konuşan Popülasyonlarının Tarihi". Moleküler Biyoloji ve Evrim. 24 (10): 2180–95. doi:10.1093 / molbev / msm155. PMID 17656633.
- ^ a b Chen, Yu-Sheng; Olckers, Antonel; Schurr, Theodore G .; Kogelnik, Andreas M .; Huoponen, Kirsi; Wallace, Douglas C. (2000). "Güney Afrika Kung ve Khwe'deki MtDNA Varyasyonu - ve Bunların Diğer Afrika Popülasyonlarıyla Genetik İlişkileri". Amerikan İnsan Genetiği Dergisi. 66 (4): 1362–83. doi:10.1086/302848. PMC 1288201. PMID 10739760.
- ^ a b Knight, Alec; Underhill, Peter A .; Mortensen, Holly M .; Zhivotovsky, Lev A .; Lin, Alice A .; Henn, Brenna M .; Louis, Dorothy; Ruhlen, Merritt; Dağ, Joanna L. (2003). "Afrika Y Kromozomu ve mtDNA Ayrışması, Tıklama Dillerinin Tarihçesi Hakkında Bilgi Sağlıyor". Güncel Biyoloji. 13 (6): 464–73. doi:10.1016 / S0960-9822 (03) 00130-1. PMID 12646128. S2CID 52862939.
- ^ a b c d Schlebusch, Carina M .; Naidoo, Thijessen; Soodyall Himla (2009). "Afrika'da bulunan mitokondriyal makro haplogrupları çözmek için SNaPshot mini sıralaması". Elektroforez. 30 (21): 3657–64. doi:10.1002 / elps.200900197. PMID 19810027. S2CID 19515426.
- ^ a b Quintana-Murci, Lluis; Harmant, Christine; Quach, Hélène; Balanovsky, Oleg; Zaporozhchenko, Valery; Bormans, Connie; Van Helden, Paul D .; Hoal, Eileen G .; Behar, Doron M. (2010). "Güney Afrika Renkli Nüfusuna Güçlü Anne Khoisan Katkısı: Cinsiyete Bağlı Bir Karışım Örneği". Amerikan İnsan Genetiği Dergisi. 86 (4): 611–20. doi:10.1016 / j.ajhg.2010.02.014. PMC 2850426. PMID 20346436.
- ^ Rídl, Jakub; Edens, Christopher M .; Černý, Viktor (2009). "Yemen Nüfusunun Mitokondriyal DNA Yapısı: Bölgesel Farklılıklar ve Farklı Göç Katkılarının Etkileri". Arabistan'daki İnsan Popülasyonlarının Evrimi. Omurgalılar Paleobiyoloji ve Paleoantropoloji. s. 69–78. doi:10.1007/978-90-481-2719-1_5. ISBN 978-90-481-2718-4.
- ^ Kampira E, Kumwenda J, van Oosterhout JJ, Dandara C. Mitokondriyal DNA althaplogrupları L0a2 ve L2a, yüksek düzeyde aktif antiretroviral tedavi içeren stavudin kullanan malawi yetişkinlerde periferik nöropatiye duyarlılığı değiştirir., J Immune Defic Syndr'i Edin. 15 Ağu 2013; 63 (5): 647-52. doi: 10.1097 / QAI.0b013e3182968ea5
- ^ Pastoralist Öncesi Güney Afrika'dan İlk Antik Mitokondriyal İnsan Genomu
Dış bağlantılar
- Genel
- Ian Logan'ın Mitokondriyal DNA Sitesi
- Mannis van Oven's Filotree
- Haplogrup L0
- Rosa, Alexandra; Brehm, Antonio; Kivisild, Toomas; Metspalu, Ene; Villems Richard (2004). "Batı Afrika Ginelilerinin MtDNA Profili: Senegambia Bölgesinin Daha İyi Anlaşılmasına Doğru". İnsan Genetiği Yıllıkları. 68 (4): 340–52. doi:10.1046 / j.1529-8817.2004.00100.x. PMID 15225159. S2CID 15391342.
Afrika tam sekansları hakkındaki önceki bilgilere dayanarak L1, önceden tanımlanmış L1a ve L1d soylarını ve L1b ve L1c sınıflarını içeren L1 sınıfını yakalayan iki monofiletik birim L0'a bölünmüştür ...
- Pavesi, A. (2005). "JC polyomavirüsün tarih öncesi zamanlara dayanan insan göçlerinin izini sürmedeki faydası". Genel Viroloji Dergisi. 86 (5): 1315–26. doi:10.1099 / vir.0.80650-0. PMID 15831942.
Diğer yandan mtDNA'nın ilk ekseni, atadan kalma L0 haplogrubunu tek bir soy oluşturduğu için aşırı sola yerleştirir ...
- Mishmar, Dan; Ruiz-Pesini, Eduardo; Brandon, Martin; Wallace, Douglas C. (2004). "Çekirdekteki mitokondriyal DNA benzeri diziler (NUMT'ler): Afrika kökenlerimize ve yabancı DNA entegrasyon mekanizmasına ilişkin bilgiler". İnsan Mutasyonu. 23 (2): 125–33. doi:10.1002 / humu.10304. PMID 14722916. S2CID 25109836.
daha önce sonuçlandırdığımız haplogrup L0 mtDNA'lar, insan mtDNA'sının tabanında yatıyor. filogenetik analize dayalı ağaç…
- Knight, Alec; Underhill, Peter A .; Mortensen, Holly M .; Zhivotovsky, Lev A .; Lin, Alice A .; Henn, Brenna M .; Louis, Dorothy; Ruhlen, Merritt; Dağ, Joanna L. (2003). "Afrika Y Kromozomu ve mtDNA Ayrışması, Tıklama Dillerinin Tarihçesi Hakkında Bilgi Sağlıyor". Güncel Biyoloji. 13 (6): 464–73. doi:10.1016 / S0960-9822 (03) 00130-1. PMID 12646128. S2CID 52862939.
- Rosa, Alexandra; Brehm, Antonio; Kivisild, Toomas; Metspalu, Ene; Villems Richard (2004). "Batı Afrika Ginelilerinin MtDNA Profili: Senegambia Bölgesinin Daha İyi Anlaşılmasına Doğru". İnsan Genetiği Yıllıkları. 68 (4): 340–52. doi:10.1046 / j.1529-8817.2004.00100.x. PMID 15225159. S2CID 15391342.
