SNP genotipleme - SNP genotyping - Wikipedia
SNP genotipleme genetik varyasyonların ölçüsüdür tek nükleotid polimorfizmleri Bir türün üyeleri arasındaki (SNP'ler). Bu bir biçimdir genotipleme, daha genel genetik varyasyonun ölçüsüdür. SNP'ler, en yaygın genetik varyasyon türlerinden biridir. Bir SNP, belirli bir aralıktaki tek bir baz çifti mutasyonudur. mahal, genellikle ikiden oluşur aleller (nerede nadir alel frekansı >% 1). SNP'lerin, etiyoloji birçok insan hastalığının içinde ve özellikle ilgi çekici hale geliyor farmakogenetik. SNP'ler evrim sırasında korundukları için, kantitatif özellik lokuslarında kullanım için belirteçler olarak önerilmiştir (QTL ) analiz ve içinde ilişkilendirme çalışmaları yerine mikro uydular. SNP'lerin kullanımı, HapMap insan genomunu genotiplemek için gereken minimum SNP setini sağlamayı amaçlayan proje. SNP'ler ayrıca kimlik testinde kullanılmak üzere genetik bir parmak izi sağlayabilir.[1] SNP'lere olan ilginin artması, çok çeşitli SNP genotipleme yöntemlerinin şiddetli gelişimi tarafından yansıtılmıştır.
Hibridizasyon tabanlı yöntemler
Tamamlayıcı DNA problarını SNP sitesine hibridize ederek SNP'leri sorgulayan çeşitli uygulamalar geliştirilmiştir. Bu yaklaşımın zorluğu, alele özgü problar arasındaki çapraz hibridizasyonu azaltmaktır. Bu zorluğun üstesinden genellikle hibridizasyon sertlik koşullarının manipüle edilmesi ile gelinir.[1]
Dinamik alele özgü hibridizasyon

Dinamik alele özgü hibridizasyon (DASH) genotipleme, uyumsuz baz çiftlerinin kararsızlığından kaynaklanan DNA'daki erime sıcaklığındaki farklılıklardan yararlanır. Süreç büyük ölçüde otomatikleştirilebilir ve birkaç basit ilkeyi kapsar.
İlk adımda, bir genomik segment büyütülür ve bir boncuğa bir PCR reaksiyonu biyotinlenmiş bir primer ile. İkinci adımda, güçlendirilmiş ürün bir Streptavidin kolon ve biyotinile edilmemiş ipliği çıkarmak için NaOH ile yıkandı. Bir alele özgü oligonükleotid çift sarmallı DNA'ya bağlandığında floresan veren bir molekül varlığında eklenir. Yoğunluk daha sonra sıcaklık yükseldikçe ölçülür. erime sıcaklığı (Tm) Belirlenebilir. Bir SNP, beklenenden daha düşük Tm ile sonuçlanacaktır.[2]
DASH genotiplemesi, Tm'deki ölçülebilir bir değişikliği ölçtüğü için, sadece SNP'leri değil, her tür mutasyonu ölçebilir. DASH'ın diğer faydaları arasında etiketsiz problarla çalışma yeteneği ve basit tasarım ve performans koşulları yer alır.
Moleküler işaretçiler

SNP tespiti moleküler işaretçiler özel olarak tasarlanmış tek sarmallı bir oligonükleotid probu kullanır. Oligonükleotid, her bir uçta tamamlayıcı bölgeler ve aralarında yer alan bir sonda dizisi olacak şekilde tasarlanmıştır. Bu tasarım, probun bir saç tokası veya gövde halkası yapısını doğal, izole durumunda almasını sağlar. Probun bir ucuna bir florofor ve diğer ucuna bir flüoresan söndürücü eklenmiştir. Probun gövde-halka yapısı nedeniyle, florofor söndürücüye çok yakındır, dolayısıyla molekülün herhangi bir floresan yaymasını engeller. Molekül aynı zamanda, testte kullanılacak genomik DNA'ya yalnızca prob sekansı tamamlayıcı olacak şekilde tasarlanmıştır (Abravaya ve arkadaşları, 2003).
Deney sırasında moleküler işaretin prob dizisi hedef genomik DNA'sıyla karşılaşırsa, tavlanacak ve hibritlenecektir. Prob dizisinin uzunluğu nedeniyle, probun firkete segmenti, daha uzun, daha stabil bir prob-hedef hibrid oluşturma lehine denatüre edilecektir. Bu konformasyonel değişiklik, florofor ve söndürücünün, firkete birleşmesi nedeniyle sıkı yakınlıklarından kurtulmasına izin vererek molekülün floresan olmasına izin verir.
Öte yandan, sonda dizisi tamamlayıcı olmayan nükleotid kadar az bir hedef diziyle karşılaşırsa, moleküler işaret tercihen doğal saç tokası durumunda kalacaktır ve florofor söndürüldükçe hiçbir floresan gözlenmeyecektir.

Bu moleküler işaretçilerin benzersiz tasarımı, belirli bir konumdaki SNP'leri tanımlamak için basit bir teşhis testine izin verir. Bir moleküler işaret, vahşi tipte bir aleli eşleştirmek için ve diğeri alelin bir mutantına uyacak şekilde tasarlanırsa, ikisi bir bireyin genotipini tanımlamak için kullanılabilir. Tahlil sırasında sadece ilk probun florofor dalga boyu tespit edilirse, birey vahşi tipe homozigottur. Yalnızca ikinci probun dalga boyu tespit edilirse, birey mutant alele homozigottur. Son olarak, eğer her iki dalga boyu da tespit edilirse, o zaman her iki moleküler işaretçi, tamamlayıcılarına hibritlenmelidir ve bu nedenle, birey hem alelleri içermeli hem de heterozigot olmalıdır.
SNP mikro dizileri
İçinde yüksek yoğunluklu oligonükleotid SNP dizileri Küçük bir çip üzerine dizilmiş yüz binlerce sonda, birçok SNP'nin aynı anda sorgulanmasına izin veriyor.[1] SNP alelleri yalnızca bir nükleotidde farklılık gösterdiğinden ve dizideki tüm problar için optimum hibridizasyon koşullarının elde edilmesi zor olduğundan, hedef DNA, eşleşmeyen problara hibritleşme potansiyeline sahiptir. Bu, her SNP'yi sorgulamak için birkaç fazlalık sonda kullanılarak bir şekilde giderilir. Problar, SNP sahasının birkaç farklı konumda bulunmasının yanı sıra SNP aleline uyumsuzluklar içerecek şekilde tasarlanmıştır. Hedef DNA'nın farklı hibridizasyon miktarını bu fazlalık probların her biri ile karşılaştırarak, spesifik homozigot ve heterozigot allelleri belirlemek mümkündür.[1] Oligonükleotid mikrodizileri nispeten daha düşük bir özgüllük ve duyarlılığa sahip olmasına rağmen, sorgulanabilen SNP'lerin ölçeği büyük bir faydadır. Afimetriks İnsan SNP 5.0 GeneChip, 500.000'den fazla insan SNP'sini genotipleyebilen genom çapında bir test gerçekleştirir (Affymetrix 2007).
Enzim bazlı yöntemler
Dahil olmak üzere geniş bir enzim yelpazesi DNA ligaz, DNA polimeraz ve nükleazlar yüksek doğrulukta SNP genotipleme yöntemleri oluşturmak için kullanılmıştır.
Kısıtlama parçası uzunluğu polimorfizmi
Kısıtlama parçası uzunluğu polimorfizmi (RFLP), SNP'leri tespit etmek için en basit ve en eski yöntem olarak kabul edilir. SNP-RFLP, birçok farklı kısıtlama endonükleazını ve bunların benzersiz ve spesifik kısıtlama sitelerine yüksek afinitesini kullanır. Bir genomik numune üzerinde bir sindirim gerçekleştirerek ve bir jel deneyi ile parça uzunluklarını belirleyerek, enzimlerin beklenen kısıtlama bölgelerini kesip kesmediğini belirlemek mümkündür. Genomik numunenin kesilmesindeki bir başarısızlık, tanımlanabilir şekilde beklenenden daha büyük bir fragmana neden olur; bu da, kısıtlama bölgesinin noktasında nükleaz aktivitesinden koruma sağlayan bir mutasyon olduğunu gösterir.
Ne yazık ki, çoğu ökaryotik genomun yüksek karmaşıklığının birleşik faktörleri, spesifik endonükleazlar için gereklilik, kesin mutasyonun tek bir deneyde çözülemeyeceği gerçeği ve jel tahlillerinin yavaş doğası, RFLP'yi yüksek verim için zayıf bir seçim haline getirmektedir. analizi.
PCR tabanlı yöntemler
Tetra-primer amplifikasyon refrakter mutasyon sistemi PCR veya ARMS-PCR, bir PCR reaksiyonunda iki alleli amplifiye etmek için iki çift primer kullanır. Primerler, iki primer çifti bir SNP konumunda üst üste gelecek, ancak her biri olası SNP'lerden yalnızca biriyle mükemmel şekilde eşleşecek şekilde tasarlanmıştır. Buluşun temeli, beklenmedik bir şekilde, uyumsuz bir 3'-kalıntıya sahip oligonükleotidlerin, uygun koşullar altında PCR'de primerler olarak işlev görmemesidir.[3] Sonuç olarak, PCR reaksiyonunda belirli bir alel mevcutsa, bu alele özgü primer çifti ürün üretecek ancak farklı bir SNP'ye sahip alternatif aleli üretmeyecektir. İki primer çifti, aynı zamanda, PCR ürünlerinin, jel elektroforezi veya erime sıcaklığı analizi ile kolayca ayırt edilebilen bantlara izin veren önemli ölçüde farklı bir uzunlukta olacağı şekilde tasarlanmıştır.[4] Sonuçları incelerken, bir genomik numune homozigot ise, ortaya çıkan PCR ürünleri, SNP konumu ve dış zıt iplikli primer ile eşleşen primerden ve iki dış primerden olacaktır. Genomik örnek heterozigot ise, ürünler her alelin primerinden ve bunların ilgili dış primer benzerlerinden ve ayrıca dış primerlerden oluşacaktır.
Alternatif bir strateji, her aleli ayrı ayrı hedefleyen farklı primer setleriyle birden fazla qPCR reaksiyonu çalıştırmaktır. İyi tasarlanmış primerler, hedef SNP'lerini diğer SNP'lerden çok daha erken bir döngüde yükseltecektir. Bu, her SNP için ayrı bir qPCR reaksiyonu gerekmesine rağmen ikiden fazla alelin ayırt edilmesine izin verir. Yeterince yüksek özgüllük elde etmek için, primer dizisi, genellikle Taq-MAMA olarak bilinen bir yaklaşım olan 3'-ucunun yakınına yapay bir uyumsuzluğun yerleştirilmesini gerektirebilir.[5]
Flep endonükleaz
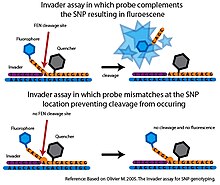
Flep endonükleaz (FEN), yapıya özgü bölünmeyi katalize eden bir endonükleazdır. Bu bölünme, uyumsuzluklara karşı oldukça hassastır ve SNP'leri yüksek derecede özgüllükle sorgulamak için kullanılabilir.[6]
Temelde İşgalci tahlil klevaz adı verilen bir FEN, hedef DNA ile birlikte klevaz tarafından tanınan üçlü bir yapı oluşturabilen iki spesifik oligonükleotid probu ile birleştirilir.[6] İlk araştırma İşgalci oligonükleotid, hedef DNA'nın 3 'ucuna tamamlayıcıdır. Son üssü İşgalci oligonükleotid, hedef DNA'daki SNP nükleotidiyle örtüşen eşleşmeyen bir bazdır. İkinci prob, hedef DNA'nın 5 'ucunu tamamlayan, ancak aynı zamanda SNP nükleotidinin 3' tarafını geçen allele özgü bir probdur. Alele özgü prob, SNP nükleotidine tamamlayıcı bir baz içerecektir. Hedef DNA istenen aleli içeriyorsa İstilacı ve alele özgü problar, üçlü yapıyı oluşturan hedef DNA'ya bağlanacaktır. Bu yapı, alele özgü probun 3 'ucunu yaracak ve serbest bırakacak olan klevaz tarafından tanınır. Hedef DNA'daki SNP nükleotidi, alele özgü proba tamamlayıcı değilse, doğru üçlü yapı oluşmaz ve bölünme meydana gelmez. İşgalci tahlil, bölünme olayını saptamak için genellikle floresan rezonans enerji transferi (FRET) sistemi ile birleştirilir. Bu kurulumda, alele özgü probun 3 'ucuna bir söndürücü molekülü ve 5' ucuna bir florofor eklenir. Bölünme meydana gelirse, florofor söndürücü molekülden ayrılacak ve tespit edilebilir bir sinyal üretecektir.[6]
Uyumsuz problarda yalnızca minimum bölünme meydana gelir. İşgalci test son derece spesifik. Bununla birlikte, orijinal formatında, reaksiyon numunesi başına yalnızca bir SNP aleli sorgulanabilirdi ve makul bir zaman çerçevesinde saptanabilir bir sinyal oluşturmak için büyük miktarda hedef DNA gerektirdi.[6] Çeşitli gelişmeler orijinali genişletti İşgalci tahlil. İkincil FEN bölünme reaksiyonlarını gerçekleştirerek, Seri İnvazif Sinyal Amplifikasyon Reaksiyonu (SISAR), her iki SNP alelinin tek bir reaksiyonda sorgulanmasına izin verir. SİSAR İşgalci tahlil ayrıca daha az hedef DNA gerektirir ve orijinalin duyarlılığını artırır İşgalci tahlil.[6] Tahlil, aynı zamanda, yüksek verimli bir formatta kullanılmak üzere çeşitli şekillerde uyarlanmıştır. Tek bir platformda, alele özgü problar mikrokürelere sabitlenir. FEN tarafından bölünme saptanabilir bir floresan sinyali oluşturduğunda, sinyal akış sitometrisi kullanılarak ölçülür. Akış sitometrisinin hassasiyeti, hedef DNA'nın PCR amplifikasyonu ihtiyacını ortadan kaldırır (Rao ve ark. 2003). Bu yüksek verimli platformlar ilke kanıtı aşamasının ötesine geçmedi ve şu ana kadar İşgalci sistemi herhangi bir büyük ölçekli SNP genotipleme projesinde kullanılmamıştır.[6]
Astar uzantısı
Primer uzantısı, ilk önce bir probun SNP nükleotidinin hemen yukarısındaki bazlara hibridizasyonunu ve ardından DNA polimerazın hibridize primeri tamamlayıcı bir baz ekleyerek genişlettiği bir 'mini sekanslama' reaksiyonunu içeren iki aşamalı bir süreçtir. SNP nükleotidi. Bu birleştirilen baz, SNP alelini tespit eder ve belirler (Goelet ve diğerleri 1999; Syvanen 2001). Primer uzantısı, yüksek doğrulukta DNA polimeraz enzimine dayandığından, yöntem genellikle çok güvenilirdir. Primer uzantısı, çok benzer reaksiyon koşulları altında çoğu SNP'yi genotipleyebilir ve bu da onu oldukça esnek hale getirir. Primer genişletme yöntemi, bir dizi tahlil formatında kullanılır. Bu formatlar, aşağıdakileri içeren çok çeşitli algılama teknikleri kullanır: MALDI-TOF Kütle spektrometresi (görmek Sequenom ) ve ELISA benzeri yöntemler.[1]
Genel olarak, flüoresan olarak etiketlenmiş her ikisinin birleşimini kullanan iki ana yaklaşım vardır. dideoksinükleotidler (ddNTP) veya floresan etiketli deoksinükleotidler (dNTP). DdNTP'ler ile, problar, SNP nükleotidinin hemen üst tarafındaki hedef DNA'ya hibritlenir ve SNP alleline tamamlayıcı olan tek bir ddNTP, probun 3 'ucuna eklenir (didiyoksinükleotiddeki eksik 3'-hidroksil, diğer nükleotidlerin eklenmesini önler ). Her bir ddNTP, aynı reaksiyondaki dört allelin tümünün saptanmasına izin veren farklı bir floresan sinyal ile etiketlenir. DNTP'ler ile, alele özgü problar, sorgulanan SNP allellerinin her birine tamamlayıcı olan 3 'bazlarına sahiptir. Hedef DNA, probun 3 'tabanına tamamlayıcı bir alel içeriyorsa, hedef DNA, proba tamamen hibridize olacak ve DNA polimerazın, probun 3' ucundan uzanmasına izin verecektir. Bu, floresan etiketli dNTP'lerin probun ucuna dahil edilmesiyle saptanır. Hedef DNA, probun 3 'tabanını tamamlayıcı bir alel içermiyorsa, hedef DNA, probun 3' ucunda bir uyumsuzluk üretecek ve DNA polimeraz, probun 3 'ucundan uzanamayacaktır. İkinci yaklaşımın yararı, birkaç etiketlenmiş dNTP'nin artan sinyale izin vererek büyüyen sarmala dahil edilebilmesidir. Bununla birlikte, bazı nadir durumlarda DNA polimeraz, yanlış bir pozitif sonuç veren uyumsuz 3 'problarından uzanabilir.[1]
Tarafından farklı bir yaklaşım kullanılır Sequenom 's iPLEX SNP genotipleme yöntemi, bir MassARRAY kütle spektrometresi kullanır. Uzatma probları, 40 farklı SNP testinin bir PCR kokteylinde amplifiye edilebileceği ve analiz edilebileceği şekilde tasarlanmıştır. Uzatma reaksiyonu, yukarıdaki gibi ddNTP'leri kullanır, ancak SNP alelinin saptanması, bir floresan moleküle değil, uzatma ürününün gerçek kütlesine bağlıdır. Bu yöntem, düşük ila orta yüksek verim içindir ve tüm genom taraması için tasarlanmamıştır.
Primer genişletmenin esnekliği ve özgüllüğü, onu yüksek verim analizine uygun hale getirir. Primer uzatma probları, birçok SNP'nin aynı anda genotiplenmesini sağlayan slaytlar üzerinde sıralanabilir. Genel olarak dizili primer uzantısı (APEX) olarak anılan bu teknolojinin, probların diferansiyel hibridizasyonuna dayalı yöntemlere göre çeşitli faydaları vardır. Nispeten, APEX yöntemleri, bu diferansiyel hibridizasyonu kullanan yöntemlerden daha fazla ayırt etme gücüne sahiptir, çünkü DNA mikrodizilerindeki binlerce prob için optimal hibridizasyon koşullarını elde etmek genellikle imkansızdır (genellikle bu, yüksek oranda fazlalık problara sahip olarak ele alınır). Bununla birlikte, APEX yöntemlerinde aynı prob yoğunluğu elde edilemez, bu da çalışma başına daha düşük çıktı anlamına gelir.[1]
Illumina Anonim Infinium tahlil, primer uzatma yöntemine dayanan bir tam genom genotipleme boru hattının bir örneğidir. Infinium testinde 100.000'den fazla SNP genotiplenebilir. Tahlil, bir primer uzatma reaksiyonunda hapten etiketli nükleotidler kullanır. Hapten etiketi, anti-cisimler tarafından tanınır ve bunlar da saptanabilir bir sinyale bağlanır (Gunderson ve diğerleri, 2006).
APEX-2 verimli homojen kullanarak paralel olarak yüzlerce SNP veya mutasyonu tanımlayabilen bir dizi primer uzantısı genotipleme yöntemidir. multipleks PCR (640-plex'e kadar) ve bir mikrodizide dört renkli tek tabanlı uzatma. Multipleks PCR, test edilen baz çiftini içeren amplikon üreten SNP / mutasyon başına iki oligonükleotid gerektirir. Bir mikrodizi üzerinde hareketsizleştirilmiş tek bazlı uzatma primerleri olarak aynı oligonükleotidler aşağıdaki adımda kullanılır (Krjutskov ve diğerleri 2008).
5’- nükleaz
Taq DNA polimerazın 5'-nükleaz aktivitesi, TaqMan SNP genotiplemesi için analiz. TaqMan tahlil, bir PCR reaksiyonu ile eş zamanlı olarak gerçekleştirilir ve sonuçlar, PCR reaksiyonu ilerledikçe gerçek zamanlı olarak okunabilir (McGuigan & Ralston 2002). Tahlil, SNP polimorfik bölgesini içeren bir bölgeyi büyütecek ileri ve geri PCR primerleri gerektirir. Allel ayrımcılığı kullanılarak elde edilir FRET SNP polimorfik sahasına hibridize olan bir veya iki alel spesifik prob ile kombine. Probların 5 'uçlarına bağlı bir florofor ve 3' uçlarına bağlı bir söndürücü molekülü olacaktır. Prob bozulmamışken söndürücü, floroforun yakınında kalacak ve floroforun sinyalini ortadan kaldıracaktır. PCR amplifikasyonu adımı sırasında, alele özgü prob SNP aleline mükemmel bir şekilde tamamlayıcıysa, hedef DNA zincirine bağlanacak ve ardından DNA'yı PCR'den genişletirken Taq polimerazın 5'-nükleaz aktivitesi ile bozunacaktır. astarlar. Probun bozulması, floroforun söndürücü molekülden ayrılmasıyla sonuçlanır ve tespit edilebilir bir sinyal oluşturur. Allele özgü prob tam olarak tamamlayıcı değilse, daha düşük erime sıcaklığına sahip olacak ve verimli bir şekilde bağlanmayacaktır. Bu, nükleazın sonda üzerinde hareket etmesini engeller (McGuigan & Ralston 2002).
Beri TaqMan test PCR'ye dayalıdır, uygulanması nispeten kolaydır. TaqMan tahlil, yedi adede kadar SNP'nin tespiti tek bir reaksiyonda birleştirilerek çoğaltılabilir. Bununla birlikte, her SNP ayrı bir araştırma gerektirdiğinden, TaqMan test, SNP'lerin ne kadar yakın konumlandırılabileceğiyle sınırlıdır. Mikrotitre plakalarında birçok eşzamanlı reaksiyon gerçekleştirilerek tahlilin ölçeği büyük ölçüde artırılabilir. Genel olarak, TaqMan Her SNP için optimal problar ve reaksiyon koşulları tasarlanması gerektiğinden, az sayıda SNP'nin sorgulanmasını içeren uygulamalarla sınırlıdır (Syvanen 2001).
Oligonükleotid Ligasyon Deneyi
DNA ligaz, bir DNA parçasının 3 'ucunun, doğrudan bitişik bir DNA parçasının 5' ucuna ligasyonunu katalize eder. Bu mekanizma, iki probu doğrudan SNP polimorfik bölgesi üzerinde hibridize ederek bir SNP'yi sorgulamak için kullanılabilir, böylece problar hedef DNA ile aynı ise ligasyon meydana gelebilir. Oligonükleotid ligaz tahlilinde iki prob tasarlanmıştır; Hedef DNA'ya hibridize olan alele özgü bir prob, 3 'tabanı doğrudan SNP nükleotidinin üzerine yerleştirilir ve SNP polimorfik sahasının yukarı akışını (tamamlayıcı sarmalda aşağı akış) hibridize eden ikinci bir prob 5' ucu sağlar ligasyon reaksiyonu için. Alele özgü prob, hedef DNA ile eşleşirse, hedef DNA'ya tam olarak hibridize olur ve ligasyon meydana gelebilir. Ligasyon genellikle uyumsuz bir 3 'baz varlığında meydana gelmez. Bağlanmış veya bağlanmamış ürünler, jel elektroforezi, MALDI-TOF kütle spektrometrisi veya büyük ölçekli uygulamalar için kapiler elektroforez ile tespit edilebilir.[1] Oligonükleotidler üzerindeki uygun diziler ve etiketlerle, bağlanmış ürünlerden ve belirlenen genotiplerden yüksek verimli dizi verileri oluşturulabilir (Curry ve diğerleri, 2012). Çok sayıda örnek indeksinin kullanılması, yüksek verimli bir sıralama çalışmasının küçük bir bölümünde binlerce örnekteki yüzlerce SNP'de yüksek verimli dizi verilerinin oluşturulmasına izin verir. Bu, sıralama teknolojisi (MGST) tarafından yapılan büyük bir genotiplemedir.
DNA'nın fiziksel özelliklerine dayalı diğer amplifikasyon sonrası yöntemler
Erime sıcaklığı ve tek sarmallı konformasyonun karakteristik DNA özellikleri, SNP alellerini ayırt etmek için çeşitli uygulamalarda kullanılmıştır. Bu yöntemler genellikle yüksek özgüllük sağlar, ancak mümkün olan en iyi sonuçları elde etmek için oldukça optimize edilmiş koşullar gerektirir.
Tek iplikli konformasyon polimorfizmi
Tek iplikli DNA (ssDNA), üçüncül bir yapıya katlanır. Konformasyon sekansa bağlıdır ve tek baz çifti mutasyonlarının çoğu yapının şeklini değiştirecektir. Bir jele uygulandığında, üçüncül şekil ssDNA'nın hareketliliğini belirleyecek ve SNP alelleri arasında ayrım yapmak için bir mekanizma sağlayacaktır. Bu yöntem ilk olarak hedef DNA'nın PCR amplifikasyonunu içerir. Çift sarmallı PCR ürünleri, ssDNA üretmek için ısı ve formaldehit kullanılarak denatüre edilir. SsDNA, denatüre edici olmayan bir elektroforez jeline uygulanır ve üçüncül bir yapı halinde katlanmasına izin verilir. DNA sekansındaki farklılıklar, üçüncül konformasyonu değiştirecek ve ssDNA ipliği hareketliliğindeki bir fark olarak tespit edilecektir (Costabile ve ark. 2006). Bu yöntem, teknik olarak basit, nispeten ucuz olması ve yaygın olarak bulunan ekipmanı kullanması nedeniyle yaygın olarak kullanılmaktadır. Bununla birlikte, diğer SNP genotipleme yöntemleriyle karşılaştırıldığında, bu testin duyarlılığı daha düşüktür. SsDNA konformasyonunun büyük ölçüde sıcaklığa bağlı olduğu ve ideal sıcaklığın ne olduğu genellikle açık olmadığı bulunmuştur. Çoğu zaman deney birkaç farklı sıcaklık kullanılarak gerçekleştirilecektir. 400 bp'den daha uzun sekanslar kullanıldığında hassasiyet düştüğü için fragmanın uzunluğunda da bir kısıtlama vardır (Costabile ve ark. 2006).
Sıcaklık gradyanı jel elektroforezi
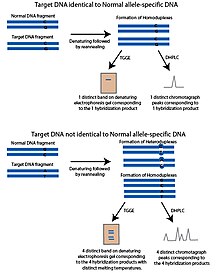
sıcaklık gradyanı jel elektroforezi (TGGE) veya sıcaklık gradyanlı kapiler elektroforez (TGCE) yöntemi, kısmen denatüre DNA'nın daha kısıtlı olduğu ve jel gibi gözenekli bir malzemede daha yavaş hareket ettiği ilkesine dayanmaktadır. Bu özellik, DNA'nın erime sıcaklığı ile ayrılmasına izin verir. Bu yöntemleri SNP tespitine uyarlamak için iki parça kullanılır; sorgulanan SNP polimorfik bölgesini içeren hedef DNA ve normal DNA parçası olarak anılan alele özgü bir DNA dizisi. Normal parça, potansiyel olarak hedef DNA'da bilinmeyen SNP polimorfik sahasında olması dışında hedef DNA ile aynıdır. Parçalar denatüre edilir ve ardından yeniden tavlanır. Hedef DNA, normal fragman ile aynı alele sahipse, aynı erime sıcaklığına sahip olacak homodubleksler oluşacaktır. Sıcaklık gradyanı ile jel üzerinde çalıştırıldığında, yalnızca bir bant görünecektir. Hedef DNA'nın farklı bir aleli varsa, yeniden tavlama aşamasını takiben dört ürün oluşacaktır; Hedef DNA'dan oluşan homodubleksler, normal DNA'dan oluşan homodubleksler ve normal DNA ipliği ile hibritlenmiş hedef DNA'nın her zincirinin iki heterdubleksini içerir. Bu dört ürün farklı erime sıcaklıklarına sahip olacak ve denatüre edici jelde dört bant olarak görünecektir.[1]
Denatüre edici yüksek performanslı sıvı kromatografisi
Denatüre edici yüksek performanslı sıvı kromatografisi (DHPLC) ters faz kullanır HPLC SNP'leri sorgulamak için. DHPLC'nin anahtarı, tek ve çift sarmallı DNA için farklı afiniteye sahip katı fazdır. DHPLC'de, DNA fragmanları ısıtılarak denatüre edilir ve sonra yeniden tavlanmaya bırakılır. Yeniden tavlanan DNA fragmanlarının erime sıcaklığı, bunların kolon içinde tutulma süresinin uzunluğunu belirler.[7] PCR kullanılarak iki parça üretilir; SNP polimorfik bölgesini ve alele özgü bir DNA sekansını içeren, normal DNA parçası olarak adlandırılan hedef DNA. Bu normal parça, potansiyel olarak hedef DNA'da bilinmeyen SNP polimorfik sahasında olması haricinde hedef DNA ile aynıdır. Parçalar denatüre edilir ve ardından kademeli olarak yeniden tavlanmaya bırakılır. Yeniden kanalize edilen ürünler DHPLC sütununa eklenir. Hedef DNA'daki SNP aleli normal DNA fragmanıyla eşleşirse, yeniden tavlama adımı sırasında sadece aynı homodubleksler oluşacaktır. Hedef DNA, normal DNA fragmanından farklı bir SNP aleli içeriyorsa, homodublekslere ek olarak hedef DNA'nın heterodubleksleri ve uyumsuz bir polimorfik alan içeren normal DNA oluşacaktır. Uyumsuz heterodubleksler, homodublekslerden farklı bir erime sıcaklığına sahip olacak ve kolon içinde uzun süre tutulmayacaktır. Bu, hedef DNA fragmanı ve normal DNA fragmanları aynıysa üretilecek modelden farklı bir kromatograf modeli oluşturur. Ayrıştırılan DNA, UV absorpsiyonu ile tespit edilir.[7]
DNA fragmanlarının etiketlenmesine veya saflaştırılmasına gerek olmadığından DHPLC kolayca otomatikleştirilir. Yöntem ayrıca nispeten hızlıdır ve yüksek bir özgüllüğe sahiptir. DHPLC'nin önemli bir dezavantajı, doğru denatürasyon derecesini elde etmek için kolon sıcaklığının her hedef için optimize edilmesi gerekliliğidir.[1]
Tüm amplikonun yüksek çözünürlüklü erimesi
Yüksek Çözünürlüklü Erime analizi anlaşılması en basit PCR tabanlı yöntemdir. Temel olarak, jel tekniklerinin çalışmasına izin veren aynı termodinamik özellikler burada ve gerçek zamanlı olarak geçerlidir. Bir florimetre, tüm dsDNA amplikonunun PCR sonrası denatürasyonunu izler. Yükseltmek istediğiniz siteye özgü primerler yaparsınız. Amplikonu, PCR karışımına dahil olan çift iplikli özel bir boya ile "boyarsınız". Ds'ye özgü boya kendisini PCR ürününe entegre eder. Temelde, tüm amplikon bir sonda haline gelir. Bu keşif için yeni olanaklar açar. Ya primerleri söz konusu SNP'nin her iki tarafına çok yakın yerleştirirsiniz (küçük amplikon genotipleme, Liew, 2004) ya da tarama amacıyla daha büyük bir bölgeyi (uzunluk olarak 100-400bp) büyütün. Bir SNP'nin basit genotiplemesi için, bir SNP'yi diğeriyle hata yapma olasılığınızı en aza indirmek için amplikonu küçültmek daha kolaydır. Tüm amplikonun erime sıcaklığı (Tm) belirlenir ve çoğu homozigot, Tm'den genotipe yeterince farklıdır (daha iyi cihazlarda). Heterozigotları ayırt etmek daha da kolaydır çünkü bunlar, eriyik geçişini genişleten ve genellikle iki fark edilebilir tepe veren heterodublekslere sahiptir (jel bazlı açıklamalara bakın). Floresan etiketli bir primer kullanılarak amplikon eritme açıklanmıştır (Gundry ve diğerleri, 2003), ancak florojenik primerin maliyeti nedeniyle ds'ye özgü boyalar kullanmaktan daha az pratiktir.
Daha büyük amplikonların taranması, yukarıda özetlenen aynı prensiplere dayanmaktadır. Bununla birlikte, erime sıcaklığı ve erime eğrisinin genel şekli bilgi verici hale gelir. Amplikonlar> c.150bp için genellikle> 2 erime tepe noktası vardır ve bunların her biri DNA şablon bileşimine bağlı olarak değişebilir. Çok sayıda araştırmacı, eriyik tabanlı tarama yoluyla dizilişlerinin çoğunu başarılı bir şekilde ortadan kaldırmayı başardı, bu da çok sayıda bireyin doğru lokusa dayalı genotiplemesine izin verdi.[8] Birçok araştırmacı, tüm genleri incelemek için uygun ve pratik bir yol olarak yüksek çözünürlüklü eritmeyi kullanarak mutasyon taraması buldu.
DNA uyuşmazlığı bağlayıcı proteinlerin kullanımı
DNA uyuşmazlığı bağlayıcı proteinler, tek nükleotid uyumsuzluklarını ayırt edebilir ve böylece SNP'lerin farklı analizini kolaylaştırabilir. Örneğin, MutS proteini Thermus aquaticus farklı tek nükleotid uyumsuzluklarını farklı afinitelerle bağlar ve altı takım uyumsuzluğu ayırt etmek için kapiler elektroforezde kullanılabilir (Drabovich & Krylov 2006).
SNPlex
SNPlex, tarafından satılan tescilli bir genotipleme platformudur Uygulamalı Biyosistemler.
Surveyor nükleaz testi
Surveyor nükleaz, tüm baz ikamelerini ve küçük insersiyonları / silmeleri (indelleri) tanıyan ve her iki DNA ipliğindeki uyumsuz bölgelerin 3 ′ tarafını ayıran uyumsuz bir endonükleaz enzimidir.
Sıralama
Yeni nesil sıralama gibi teknolojiler Pyrosequencing bir okumada 250 bazdan daha az dizi, tüm genomları sıralama yeteneklerini sınırlar. Bununla birlikte, sonuçları gerçek zamanlı olarak üretme yetenekleri ve büyük ölçüde büyütme potansiyelleri, onları SNP genotiplemesi gerçekleştirmek için küçük bölgeleri sıralamak için uygun bir seçenek haline getirir. Diğer SNP genotipleme yöntemleriyle karşılaştırıldığında, dizileme özellikle yüksek polimorfik gibi küçük bir bölgedeki birden çok SNP'yi tanımlamak için uygundur. Başlıca Histo-uyumluluk Kompleksi genom bölgesi.[1]
Referanslar
Bu makale genel bir liste içerir Referanslar, ancak büyük ölçüde doğrulanmamış kalır çünkü yeterli karşılık gelmiyor satır içi alıntılar. (Temmuz 2012) (Bu şablon mesajını nasıl ve ne zaman kaldıracağınızı öğrenin) |
- ^ a b c d e f g h ben j k Harbron S; Rapley R (2004). Moleküler analiz ve genom keşfi. Londra: John Wiley & Sons Ltd. ISBN 978-0-471-49919-0.
- ^ Howell WM; İşler M; Gyllensten U; Brookes AJ (Ocak 1999). "Dinamik alele özgü hibridizasyon. Tek nükleotid polimorfizmlerini puanlamak için yeni bir yöntem". Nat. Biyoteknol. 17 (1): 87–8. doi:10.1038/5270. PMID 9920276.
- ^ Newton CR, Graham A, Heptinstall LE, Powell SJ, Summers C, Kalsheker N, Smith JC, Markham AF (1989). "DNA'daki herhangi bir nokta mutasyonunun analizi. Amplifikasyona dirençli mutasyon sistemi (ARMS)". Nükleik Asit Araştırması. 17 (7): 2503–2516. doi:10.1093 / nar / 17.7.2503. PMC 317639. PMID 2785681.CS1 Maint: birden çok isim: yazarlar listesi (bağlantı)
- ^ Birdsell D.N., Pearson T., Price E. P., Hornstra H.M., Nera R. D., Stone N .; et al. (2012). "Uyumsuzluk Amplifikasyon Mutasyon Testlerinin (Eriyik-MAMA) Eriyik Analizi: Bakteriyel Modellerde Maliyet Etkili SNP Genotipleme Testinin Fonksiyonel Bir Çalışması". PLOS ONE. 7 (3): e32866. doi:10.1371 / journal.pone.0032866.s002.CS1 Maint: birden çok isim: yazarlar listesi (bağlantı)
- ^ Li B., Kadura I., Fu D.-J., Watson D. E. (2004). "TaqMAMA ile genotipleme". Genomik. 83 (2): 311–320. doi:10.1016 / j.ygeno.2003.08.005. PMID 14706460.CS1 Maint: birden çok isim: yazarlar listesi (bağlantı)
- ^ a b c d e f Olivier M (Haziran 2005). "SNP genotiplemesi için Invader analizi". Mutat. Res. 573 (1–2): 103–10. doi:10.1016 / j.mrfmmm.2004.08.016. PMC 2771639. PMID 15829241.
- ^ a b Oefner PJ; Underhill PA (1995). "Yüksek performanslı sıvı kromatografisinin (DHPLC) denatüre edilmesiyle karşılaştırmalı DNA dizilemesi". Am J Hum Genet. 57: 103–10.
- ^ (Dang ve diğerleri, 2012)
daha fazla okuma
- Abravaya K; Huff J; Marshall R; Satıcı B; et al. (Nisan 2003). "Teşhis araçları olarak moleküler işaretler: teknoloji ve uygulamalar". Clin. Chem. Lab. Orta. 41 (4): 468–74. doi:10.1515 / CCLM.2003.070. PMID 12747588.
- Costabile M; Quach A; Ferrante A (Aralık 2006). "Birincil immün yetmezlik hastalıklarının tanısında moleküler yaklaşımlar". Hum. Mutat. 27 (12): 1163–73. doi:10.1002 / humu.20412. PMC 7165860. PMID 16960849.
- Drabovich AP; Krylov SN (Mart 2006). "Tek nükleotid polimorfizmlerindeki baz çiftlerinin MutS proteini aracılı kapiler elektroforez ile tanımlanması". Anal. Kimya. 78 (6): 2035–8. doi:10.1021 / ac0520386. PMID 16536443.
- Gundry CN; Vandersteen JG; Reed GH; Pryor RJ; et al. (Mart 2003). "Etiketli primerlerle amplikon erime analizi: homozigotları ve heterozigotları ayırt etmek için kapalı tüp yöntemi". Clin. Kimya. 49 (3): 396–406. doi:10.1373/49.3.396. PMID 12600951.
- Gunderson KL; Steemers FJ; Ren H; Ng P; et al. (2006). Tüm genom genotipleme. Meth. Enzimol. Enzimolojide Yöntemler. 410. s. 359–76. doi:10.1016 / S0076-6879 (06) 10017-8. ISBN 9780121828158. PMID 16938560.
- Liew M; Pryor R; Palais R; Çayırlar C; et al. (Temmuz 2004). "Küçük amplikonların yüksek çözünürlüklü eritilmesiyle tek nükleotid polimorfizmlerinin genotiplenmesi". Clin. Kimya. 50 (7): 1156–64. doi:10.1373 / Clinchem.2004.032136. PMID 15229148.
- McGuigan FE; Ralston SH (Eylül 2002). "Tek nükleotid polimorfizm tespiti: TaqMan kullanarak alelik ayrım". Psychiatr. Genet. 12 (3): 133–6. doi:10.1097/00041444-200209000-00003. PMID 12218656.
- Syvänen AC (Aralık 2001). "Genetik varyasyona erişim: genotipleme tek nükleotid polimorfizmleri". Nat. Rev. Genet. 2 (12): 930–42. doi:10.1038/35103535. PMID 11733746.
Dış bağlantılar
- Uluslararası HapMap Projesi
- Delaware Valley Kişiselleştirilmiş Tıp Projesi Tıbbı kişiselleştirmeye yardımcı olmak için SNP'leri kullanır
- Oligonükleotid Ligasyon Deneyi (OLA) ile genotipleme
- SNP farelerinizi neden test ediyor?
