MRNA gözetimi - MRNA surveillance
mRNA gözetim mekanizmaları vardır yollar organizmalar tarafından sadakat ve kaliteyi sağlamak için kullanılır haberci RNA (mRNA) molekülleri. Hücrelerde bir dizi gözetim mekanizması mevcuttur. Bu mekanizmalar mRNA'nın çeşitli adımlarında çalışır biyogenez uygun şekilde işlenmemiş transkriptleri tespit etme ve bozma yolu.
Genel Bakış
tercüme nın-nin haberci RNA proteinlere transkriptler, moleküler biyolojinin temel dogması. Bununla birlikte, mRNA molekülleri, mRNA'nın kaliteye çevrilmesinde hatalara neden olabilecek bir dizi aslına uygunluk hatalarına eğilimlidir. proteinler.[1] RNA gözetim mekanizmaları, hücrelerin mRNA moleküllerinin kalitesini ve aslına uygunluğunu sağlamak için kullandığı yöntemlerdir.[2] Bu genellikle anormal mRNA molekülünü işaretleyerek elde edilir. bozulma çeşitli endojen nükleazlar.[3]
mRNA sürveyansı, bakteri ve Maya. İçinde ökaryotlar, bu mekanizmaların hem çekirdek ve sitoplazma.[4] Çekirdekteki mRNA moleküllerinin doğruluk kontrolleri, sitoplazmaya aktarılmadan önce yanlış işlenmiş transkriptlerin bozunmasına neden olur. Transkriptler, sitoplazmada bir kez daha fazla gözetime tabidir. Sitoplazmik gözetim mekanizmaları mRNA'yı değerlendirir transkriptler erken durdurma kodonlarının yokluğu veya varlığı için.[3][4]
Şu anda üç gözetim mekanizmasının işlediği bilinmektedir. hücreler: anlamsız aracılı mRNA bozunma yolu (NMD); kesintisiz aracılı mRNA bozunma yolları (NSD); ve gitmeyen aracılı mRNA bozunma yolu (NGD).
Anlamsız aracılı mRNA bozunması
Genel Bakış
Anlamsız aracılı bozunma, erken sonlandırma kodonları (PTC'ler) içeren mRNA transkriptlerinin saptanması ve bozulmasında rol oynar. PTC'ler, çeşitli mekanizmalar yoluyla hücrelerde ortaya çıkabilir: germ hattı DNA'daki mutasyonlar; somatik DNA'daki mutasyonlar; hatalar transkripsiyon; veya transkripsiyon sonrası mRNA işlemedeki hatalar.[5][6] Bu mRNA transkriptlerinin tanınmaması ve bozulmaması, organizma için zararlı olabilecek kesik proteinlerin üretimine neden olabilir. C-terminalde bozulmaya neden olarak kesilmiş polipeptitler, NMD mekanizması hücreleri zararlı maddelere karşı koruyabilir baskın -negatif ve işlev kazancı Etkileri.[7] PTC'ler, hepsinin yaklaşık% 30'unda yer almıştır. miras hastalıklar; Bu nedenle, NMD yolu, bir organizmanın genel hayatta kalmasını ve uygunluğunu sağlamada hayati bir rol oynar.[8][9]
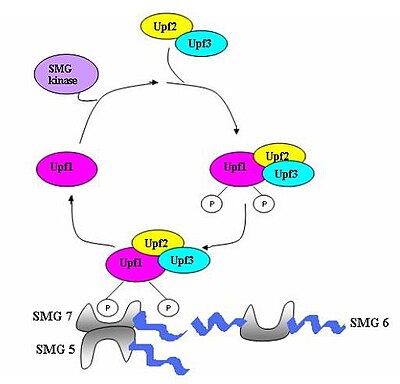
Çeşitli proteinlerden (eRF1, eRF3, Upf1, Upf2 ve Upf3) oluşan bir gözetim kompleksi birleştirilir ve erken durdurma kodonları için mRNA'yı tarar.[5] Bu kompleksin montajı erken çeviri sonlandırmasıyla tetiklenir. Prematüre bir durdurma kodonu tespit edilirse, mRNA transkripti degradasyon için sinyallenir - deteksiyonun bozunma ile birleşmesi meydana gelir.[3][10][11]
Yedi smg geni (smg1-7) ve üç UPF geni (Upf1-3), Saccharomyces cerevisiae ve Caenorhabditis elegans NMD aktivitesine katkıda bulunan temel trans-etkili faktörler olarak.[12][13] Tüm bu genler korunmuş içinde Drosophila melanogaster ve ayrıca NMD'de kritik roller oynadıkları diğer memeliler. Ökaryotlar boyunca NMD sürecinde korunan üç bileşen vardır.[14] Bunlar Upf1 / SMG-2, Upf2 / SMG-3 ve Upf3 / SMG-4 kompleksleridir. Upf1 / SMG-2, çok hücreli organizmalarda bir fosfoproteindir ve fosforilasyon aktivitesi aracılığıyla NMD'ye katkıda bulunduğu düşünülmektedir. Bununla birlikte, proteinlerin tam etkileşimleri ve NMD'deki rolleri şu anda tartışmalıdır.[11][12][14][15][16]
Memelilerde mekanizma
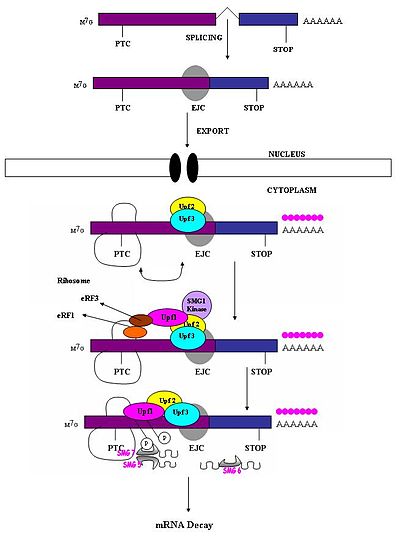
Bir erken durdurma kodonu, normal bir durdurma kodonundan farklı olarak tanınmalıdır, böylece sadece eski bir NMD yanıtını tetikler. Anlamsız bir kodonun mRNA degradasyonuna neden olma kabiliyetinin, aşağı akış sekans elemanına ve ilişkili proteinlere göreceli konumuna bağlı olduğu gözlemlenmiştir.[1] Çalışmalar göstermiştir ki nükleotidler Son ekson-ekson bileşkesinin yukarı akış yönündeki 50-54 nükleotitten fazlası, çürüme için mRNA'yı hedefleyebilir.[1][4][5][6][7][17] Bu bölgeden aşağı havzada olanlar bunu yapamaz. Bu nedenle, anlamsız kodonlar, sonuncudan 50-54 nükleotidden daha yukarı akışta bulunur. ekson sınır oysa doğal durdurma kodonları terminal eksonlar içinde bulunur.[18] Ekson bağlantı kompleksleri (EJC'ler) ekson-ekson sınırlarını işaretler. EJC'ler, ekleme bağlantısının yaklaşık 20-24 nükleotid yukarısındaki bir pozisyonda ekleme sırasında birleşen multiplrotein kompleksleridir.[19] Prematüre durdurma kodonlarını doğal durdurma kodonlarından ayırmak için gereken konum bilgisini sağlayan bu EJC'dir. PTC'lerin tanınması, ekson-ekson bağlantılarının tanımlarına bağlı görünmektedir. Bu, spliceozomun memeli NMD'sine dahil olduğunu gösterir.[17][20] Araştırma, memeli NMD'sinde spliceozom tutulumu olasılığını araştırdı ve bunun olası bir olasılık olduğunu belirledi.[18] Ayrıca, NMD mekanizmalarının, doğal olarak intron içermeyen genlerden (yani Histon H4, Hsp70, melanokortin-4-reseptörü) üretilen anlamsız transkriptler tarafından aktive edilmediği gözlemlenmiştir.[7]
Ne zaman ribozom bir PTC'ye ulaşır, çeviri faktörleri eRF1 ve eRF3, bir multiprotein köprüsü vasıtasıyla tutulan EJC kompleksleri ile etkileşime girer.[21] Etkileşimleri UPF1 sonlandırma kompleksi ile ve UPF2 / Tutulan EJC'lerin UPF3'ü kritiktir. Bu etkileşimler hızlı bir şekilde mRNA'yı hedef alır. çürüme endojen nükleazlarla[18][21]

Omurgasızlarda mekanizma
Gibi organizmaları içeren çalışmalar S. cerevisiae, D. melanogaster ve C. elegans PTC tanımanın şunları içerdiğini göstermiştir: omurgasız organizmalar ekson-ekson sınırları içermez.[20] Bu çalışmalar, omurgasız NMD'nin eklemeden bağımsız olarak gerçekleştiğini göstermektedir. Sonuç olarak, omurgasız NMD'de ekson-ekson sınırlarının işaretlenmesinden sorumlu olan EJC'ler gerekli değildir.[3] Omurgasızlarda PTC'lerin normal durdurma kodonlarından nasıl ayırt edildiğini açıklamak için birkaç model önerilmiştir. Bunlardan biri, memelilerdeki ekson bağlantılarına benzer şekilde işlev gören bir aşağı akış sekans elemanı olabileceğini düşündürmektedir.[11] İkinci bir model, bir 3 'poli-A kuyruğu gibi, mRNA'da yaygın olarak bulunan bir özelliğin, tanıma için gereken konum bilgilerini sağlayabileceğini önermektedir.[22] "Sahte 3'UTR modeli" olarak adlandırılan başka bir model, erken çeviri sonlandırmasının, uygun olmayan bir ortamda varlığını tanımasına izin verebilecek içsel özellikler nedeniyle normal sonlandırmadan ayırt edilebileceğini öne sürer.[3] Bununla birlikte, bu mekanizmalar henüz kesin olarak gösterilmemiştir.
Bitkilerde mekanizma
Bitkilerde iki PTC tanıma mekanizması vardır: EJC'den uzaklığına göre (omurgalılarda olduğu gibi) veya poli-A kuyruğundan. Bitkilerdeki NMD mekanizması, 300 nt'den daha uzun 3'NTR içeren mRNA'ların bozulmasına neden olur, bu nedenle bitkilerde daha uzun 3'NTR'lere sahip mRNA'ların oranı omurgalılara göre çok daha düşüktür.[23][24]
NMD'den kaçınma
Anlamsız mutasyonlara sahip mRNA'ların genellikle NMD yolları aracılığıyla bozunmaya hedeflendiği düşünülmektedir. Bu erken durdurma kodonunun ekson bağlantı noktasına yaklaşık 50-54 nts 5 'varlığı, hızlı bozulma için tetikleyici gibi görünmektedir; ancak, erken durdurma kodonuna sahip bazı mRNA moleküllerinin saptama ve bozunmayı önleyebildiği gözlenmiştir.[17][25] Genel olarak, bu mRNA molekülleri okuma çerçevesinde çok erken durdurma kodonuna sahiptir (yani PTC, AUG-proksimaldir). Bu pozisyon, ekson-ekson bağlantısının önemli ölçüde 5 'olduğu için NMD'nin mevcut kabul edilen modeline bir çelişki gibi görünmektedir.[26]
Bu, β-globulin'de gösterilmiştir. Genin ilk eksonunun başlarında anlamsız bir mutasyon içeren β-globulin mRNA'lar, NMD'ye duyarlı mRNA moleküllerinden daha stabildir. Algılamadan kaçınmanın kesin mekanizması şu anda bilinmemektedir. Poli-A bağlayıcı proteinin (PABP) bu stabilitede bir rol oynadığı öne sürülmüştür.[27] AUG-proksimal PTC'lerin yakınında bu proteinin varlığının, NMD'ye duyarlı bu mRNA'ların stabilitesini arttırdığı başka çalışmalarda da gösterilmiştir. Bu koruyucu etkinin sadece p-globulin destekleyici ile sınırlı olmadığı gözlenmiştir.[25] Bu, bu NMD'den kaçınma mekanizmasının çeşitli genler için diğer doku tiplerinde yaygın olabileceğini düşündürmektedir. Mevcut NMD modelinin daha ileri çalışmalarla yeniden gözden geçirilmesi gerekebilir.
Kesintisiz aracılı mRNA bozunması
Genel Bakış
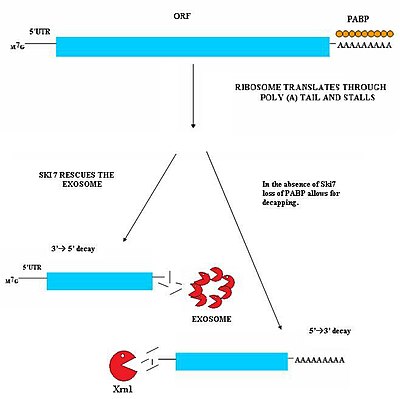
Kesintisiz aracılı bozunma (NSD), bir durdurma kodonu olmayan mRNA transkriptlerinin saptanması ve bozulmasıyla ilgilidir.[29][30] Bu mRNA transkriptleri, bir genin kodlama bölgesi içindeki erken 3 'adenilasyon veya kriptik poliadenilasyon sinyalleri gibi birçok farklı mekanizmadan kaynaklanabilir.[31] Bir durdurma kodonunun olmaması, hücreler için önemli bir soruna neden olur. MRNA'yı çeviren ribozomlar, en sonunda, transkriptlerin ve durakların 3'poly-A kuyruk bölgesine çevrilir. Sonuç olarak, mRNA'yı çıkaramaz.[32] Dolayısıyla ribozomlar, kesintisiz mRNA ile bağlantılı sekestrasyona uğrayabilir ve diğer mRNA moleküllerini proteinlere çevirmek için mevcut olmayacaktır. Kesintisiz aracılıklı bozunma, hem durmuş ribozomları serbest bırakarak hem de kesintisiz mRNA'yı hücrede nükleazlar tarafından parçalanması için işaretleyerek bu sorunu çözer. Kesintisiz aracılı bozunma, muhtemelen kesintisiz mRNA'yı bozmak için birlikte hareket eden iki farklı yoldan oluşur.[29][30]
Ski7 yolu
Bu yol, Ski7 proteini hücrede mevcut olduğunda aktiftir. Ski7 proteininin ribozomun boş A bölgesine bağlandığı düşünülmektedir. Bu bağlanma, ribozomun sıkışmış kesintisiz mRNA molekülünü dışarı atmasına izin verir - bu, ribozomu serbest bırakır ve diğer transkriptleri çevirmesine izin verir. Ski7 artık kesintisiz mRNA ile ilişkilendirilmiştir ve sitosolik tarafından tanınmak üzere kesintisiz mRNA'yı hedefleyen bu ilişkidir. ekzozom. Ski7-eksozom kompleksi, eksozomun transkripti 3 'ila 5' şeklinde bozmasına izin veren mRNA molekülünü hızla öldürür.[29][30]
Non-Ski7 patika
Mayada ikinci bir NSD türü gözlenmiştir. Bu mekanizmada, Ski7'nin yokluğu, translasyon ribozomunun etkisiyle poli-A kuyruğuna bağlanan PABP proteinlerinin kaybına neden olur. Bu PABP proteinlerinin uzaklaştırılması, daha sonra koruyucu maddenin kaybına neden olur. 5'm7G kapak. Başlığın kaybı, XrnI gibi bir endojen 5'-3 'eksonükleaz tarafından transkriptin hızlı degradasyonu ile sonuçlanır.[30]
Gitmesiz bozunma

No-Go bozunma (NGD), en son keşfedilen gözetim mekanizmasıdır.[33] Bu nedenle, şu anda iyi anlaşılmamıştır. NGD'nin otantik hedefleri tam olarak anlaşılmamış olsa da, büyük ölçüde çeviri sırasında ribozomların üzerinde durduğu mRNA transkriptlerinden oluşuyor gibi görünmektedir. Bu durma, güçlü de dahil olmak üzere çeşitli faktörlerden kaynaklanabilir. ikincil yapılar, çeviri mekanizmasının transkripte aşağı hareket etmesini fiziksel olarak engelleyebilir.[33] Dom34 / Hbs1 muhtemelen durmuş ribozomların bulunduğu A bölgesinin yakınında bağlanır ve komplekslerin geri dönüşümünü kolaylaştırabilir.[34] Bazı durumlarda, transkript, durak bölgesinin yakınında endonükleolitik bir şekilde de bölünür; ancak sorumlu endonükleazın kimliği tartışmalı kalır. parçalanmış mRNA molekülleri daha sonra eksozom tarafından 3 'ila 5' şeklinde ve Xrn1 tarafından 5 'ila 3' şeklinde tamamen parçalanır.[33]Bu sürecin mRNA'yı ribozomlardan nasıl serbest bıraktığı şu anda bilinmemektedir, ancak Hbs1, Ski7 aracılı NSD'de ribozom salınımında açık bir rol oynayan Ski7 proteini ile yakından ilişkilidir. Hbs1'in NGD'de benzer bir rol oynayabileceği varsayılmaktadır.[5][35]
Evrim
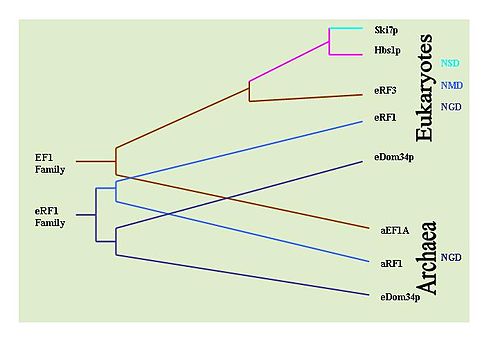
Belirlemek mümkündür evrimsel her mekanizmada yer alan anahtar proteinlerin korunmasını gözlemleyerek bu mekanizmaların geçmişi. Örneğin: Dom34 / Hbs1, NGD ile ilişkilidir;[33] Ski7, NSD ile ilişkilidir;[29] ve eRF proteinleri NMD ile ilişkilidir.[6] Bu amaçla kapsamlı ÜFLEME Proteinlerin çeşitli organizma türlerindeki prevalansını belirlemek için araştırmalar yapılmıştır. NGD Hbs1 ve NMD eRF3'ün sadece ökaryotlarda bulunduğu belirlendi. Bununla birlikte, NGD Dom34 ökaryotlarda evrenseldir ve Archaea. Bu, NGD'nin ilk evrimleşmiş mRNA gözetim mekanizması olduğunu göstermektedir. NSD Ski7 proteini, NSD'nin en son geliştirilen gözetim mekanizması olduğunu düşündüren, kesinlikle maya türleriyle sınırlı görünmektedir. Bu, varsayılan olarak NMD'yi ikinci gelişmiş gözetim mekanizması olarak bırakır.[36]
Referanslar
- ^ a b c Amrani N, Sachs MS, Jacobson A (Haziran 2006). "Erken saçmalık: mRNA bozunması bir çeviri problemini çözer". Doğa Yorumları. Moleküler Hücre Biyolojisi. 7 (6): 415–25. doi:10.1038 / nrm1942. PMID 16723977.
- ^ Moore MJ (Eylül 2005). "Doğumdan ölüme: ökaryotik mRNA'ların karmaşık yaşamları". Bilim. 309 (5740): 1514–8. Bibcode:2005Sci ... 309.1514M. doi:10.1126 / science.1111443. PMID 16141059.
- ^ a b c d e Amrani N, Ganesan R, Kervestin S, Mangus DA, Ghosh S, Jacobson A (Kasım 2004). "Sahte 3'-UTR, anormal sonlandırmayı teşvik eder ve anlamsız aracılı mRNA bozulmasını tetikler". Doğa. 432 (7013): 112–8. Bibcode:2004Natur.432..112A. doi:10.1038 / nature03060. PMID 15525991.
- ^ a b c Fasken MB, Corbett AH (Haziran 2005). "Süreç veya yok olma: mRNA biyogenezinde kalite kontrolü". Doğa Yapısal ve Moleküler Biyoloji. 12 (6): 482–8. doi:10.1038 / nsmb945. PMID 15933735.
- ^ a b c d e f g Chang YF, İmam JS, Wilkinson MF (2007). "Saçma-aracılı bozunma RNA gözetim yolu". Biyokimyanın Yıllık Değerlendirmesi. 76: 51–74. doi:10.1146 / annurev.biochem.76.050106.093909. PMID 17352659.
- ^ a b c Rehwinkel J, Raes J, Izaurralde E (Kasım 2006). "Anlamsız aracılı mRNA bozunması: Hedef genler ve efektörlerin fonksiyonel çeşitliliği". Biyokimyasal Bilimlerdeki Eğilimler. 31 (11): 639–46. doi:10.1016 / j.tibs.2006.09.005. PMID 17010613.
- ^ a b c Maquat LE (Şubat 2004). "Anlamsız aracılı mRNA bozunması: ekleme, çeviri ve mRNP dinamikleri". Doğa Yorumları. Moleküler Hücre Biyolojisi. 5 (2): 89–99. doi:10.1038 / nrm1310. PMID 15040442.
- ^ Holbrook JA, Neu-Yilik G, Hentze MW, Kulozik AE (Ağustos 2004). "Saçma kaynaklı çürüme kliniğe yaklaşıyor". Doğa Genetiği. 36 (8): 801–8. doi:10.1038 / ng1403. PMID 15284851.
- ^ Mendell JT, Sharifi NA, Meyers JL, Martinez-Murillo F, Dietz HC (Ekim 2004). "Anlamsız gözetim, memeli transkriptlerinin çeşitli sınıflarının ifadesini düzenler ve genomik gürültüyü susturur". Doğa Genetiği. 36 (10): 1073–8. doi:10.1038 / ng1429. PMID 15448691.
- ^ Lejeune F, Maquat LE (Haziran 2005). "Anlamsız aracılı mRNA bozunması ve memeli hücrelerinde pre-mRNA ekleme arasındaki mekanik bağlantılar". Hücre Biyolojisinde Güncel Görüş. 17 (3): 309–15. doi:10.1016 / j.ceb.2005.03.002. PMID 15901502.
- ^ a b c Conti E, Izaurralde E (Haziran 2005). "Anlamsız aracılı mRNA bozunması: türler arasında moleküler anlayış ve mekanik varyasyonlar". Hücre Biyolojisinde Güncel Görüş. 17 (3): 316–25. doi:10.1016 / j.ceb.2005.04.005. PMID 15901503.
- ^ a b Cali BM, Kuchma SL, Latham J, Anderson P (Şubat 1999). "Smg-7, Caenorhabditis elegans'ta mRNA sürveyansı için gereklidir". Genetik. 151 (2): 605–16. PMC 1460488. PMID 9927455.
- ^ Yamashita A, Kashima I, Ohno S (Aralık 2005). "SMG-1'in anlamsız aracılı mRNA bozulmasındaki rolü". Biochimica et Biophysica Açta (BBA) - Proteinler ve Proteomikler. 1754 (1–2): 305–15. doi:10.1016 / j.bbapap.2005.10.002. PMID 16289965.
- ^ a b Kim YK, Furic L, Desgroseillers L, Maquat LE (Ocak 2005). "Memeli Staufen1, mRNA bozulmasını ortaya çıkarmak için Upf1'i belirli mRNA 3'UTR'lere toplar". Hücre. 120 (2): 195–208. doi:10.1016 / j.cell.2004.11.050. PMID 15680326.
- ^ Longman D, Plasterk RH, Johnstone IL, Cáceres JF (Mayıs 2007). "C. elegans NMD yolundaki iki yeni faktörün mekanik anlayışları ve tanımlanması". Genler ve Gelişim. 21 (9): 1075–85. doi:10.1101 / gad.417707. PMC 1855233. PMID 17437990.
- ^ Gatfield D, Unterholzner L, Ciccarelli FD, Bork P, Izaurralde E (Ağustos 2003). "Drosophila'da anlamsız aracılı mRNA bozunması: maya ve memeli yollarının kesişme noktasında". EMBO Dergisi. 22 (15): 3960–70. doi:10.1093 / emboj / cdg371. PMC 169044. PMID 12881430.
- ^ a b c Nagy E, Maquat LE (Haziran 1998). "İntron içeren genler içinde sonlandırma-kodon pozisyonu için bir kural: saçmalık RNA bolluğunu etkilediğinde". Biyokimyasal Bilimlerdeki Eğilimler. 23 (6): 198–9. doi:10.1016 / S0968-0004 (98) 01208-0. PMID 9644970.
- ^ a b c Oyuncu TJ, Mills DJ, Horton AA (Haziran 1979). "Mikrozomal fraksiyonun lipid peroksidasyonu ve DAB kaynaklı hepatomlardan ekstrakte edilmiş mikrozomal lipidler". İngiliz Kanser Dergisi. 39 (6): 773–8. doi:10.1128 / mcb.18.9.5272. PMC 109113. PMID 9710612.
- ^ Neu-Yilik G, Gehring NH, Thermann R, Frede U, Hentze MW, Kulozik AE (Şubat 2001). "Anlamsız aracılı bozunma-yetkin insan beta-globin mRNPlerinin tanımında ekleme ve 3 'uç oluşumu". EMBO Dergisi. 20 (3): 532–40. doi:10.1093 / emboj / 20.3.532. PMC 133467. PMID 11157759.
- ^ a b Behm-Ansmant I, Gatfield D, Rehwinkel J, Hilgers V, Izaurralde E (Mart 2007). "Anlamsız aracılı mRNA bozulmasında sitoplazmik poli (A) -bağlayıcı protein 1 (PABPC1) için korunan bir rol". EMBO Dergisi. 26 (6): 1591–601. doi:10.1038 / sj.emboj.7601588. PMC 1829367. PMID 17318186.
- ^ a b Kashima I, Yamashita A, Izumi N, Kataoka N, Morishita R, Hoshino S, Ohno M, Dreyfuss G, Ohno S (Şubat 2006). "Yeni bir SMG-1-Upf1-eRF1-eRF3 kompleksinin (SURF) ekson bağlantı kompleksine bağlanması, Upf1 fosforilasyonunu ve anlamsız aracılı mRNA bozulmasını tetikler". Genler ve Gelişim. 20 (3): 355–67. doi:10.1101 / gad.1389006. PMC 1361706. PMID 16452507.
- ^ Palaniswamy V, Moraes KC, Wilusz CJ, Wilusz J (Mayıs 2006). "Nükleofosmin, poliadenilasyon sırasında seçici olarak mRNA üzerinde biriktirilir". Doğa Yapısal ve Moleküler Biyoloji. 13 (5): 429–35. doi:10.1038 / nsmb1080. PMC 2811576. PMID 16604083.
- ^ Schwartz AM, Komarova TV, Skulachev MV, Zvereva AS, Dorokhov I, Atabekov JG (Aralık 2006). "Bitki mRNA'larının stabilitesi, 3'-çevrilmemiş bölgenin uzunluğuna bağlıdır". Biyokimya. Biokhimiia. 71 (12): 1377–84. doi:10.1134 / s0006297906120145. PMID 17223792.
- ^ Nyikó T, Kerényi F, Szabadkai L, Benkovics AH, Major P, Sonkoly B, Mérai Z, Barta E, Niemiec E, Kufel J, Silhavy D (Temmuz 2013). "Bitki anlamsız aracılı mRNA bozunması, farklı otoregülasyon devreleri tarafından kontrol edilir ve EJC benzeri bir kompleks tarafından indüklenebilir". Nükleik Asit Araştırması. 41 (13): 6715–28. doi:10.1093 / nar / gkt366. PMC 3711448. PMID 23666629.
- ^ a b Inácio A, Silva AL, Pinto J, Ji X, Morgado A, Almeida F, Faustino P, Lavinha J, Liebhaber SA, Romão L (Temmuz 2004). "Başlatma kodonunun yakınındaki anlamsız mutasyonlar, tam anlamsız aracılı mRNA bozunmasını tetikleyemiyor". Biyolojik Kimya Dergisi. 279 (31): 32170–80. doi:10.1074 / jbc.m405024200. PMID 15161914.
- ^ Silva AL, Pereira FJ, Morgado A, Kong J, Martins R, Faustino P, Liebhaber SA, Romão L (Aralık 2006). "Kanonik UPF1 bağımlı anlamsız aracılı mRNA bozunması, sekans bağlamından bağımsız kısa bir açık okuma çerçevesi taşıyan transkriptlerde inhibe edilir". RNA. 12 (12): 2160–70. doi:10.1261 / rna.201406. PMC 1664719. PMID 17077274.
- ^ Silva AL, Ribeiro P, Inácio A, Liebhaber SA, Romão L (Mart 2008). "Poli (A) -bağlayıcı proteinin bir erken sonlandırma kodonuna yakınlığı, memelilerde anlamsız aracılı mRNA bozunmasını engeller". RNA. 14 (3): 563–76. doi:10.1261 / rna.815108. PMC 2248256. PMID 18230761.
- ^ Garneau NL, Wilusz J, Wilusz CJ (Şubat 2007). "MRNA çürümesinin otoyolları ve yan yolları". Doğa Yorumları. Moleküler Hücre Biyolojisi. 8 (2): 113–26. doi:10.1038 / nrm2104. PMID 17245413.
- ^ a b c d van Hoof A, Frischmeyer PA, Dietz HC, Parker R (Mart 2002). "Bir sonlandırma kodonu olmayan mRNA'ların eksozom aracılı tanınması ve bozunması". Bilim. 295 (5563): 2262–4. doi:10.1126 / bilim.1067272. PMID 11910110.
- ^ a b c d Frischmeyer PA, van Hoof A, O'Donnell K, Guerrerio AL, Parker R, Dietz HC (Mart 2002). "Sonlandırma kodonları olmayan transkriptleri ortadan kaldıran bir mRNA gözetim mekanizması". Bilim. 295 (5563): 2258–61. Bibcode:2002Sci ... 295.2258F. doi:10.1126 / science.1067338. PMID 11910109.
- ^ Temperley RJ, Seneca SH, Tonska K, Bartnik E, Bindoff LA, Lightowlers RN, Chrzanowska-Lightowlers ZM (Eylül 2003). "Patojenik bir mtDNA mikrodelesyonunun araştırılması, insan mitokondrilerinde translasyona bağlı bir deadenilasyon bozunma yolunu ortaya koymaktadır". İnsan Moleküler Genetiği. 12 (18): 2341–8. doi:10.1093 / hmg / ddg238. PMID 12915481.
- ^ Karzai AW, Roche ED, Sauer RT (Haziran 2000). "Protein etiketleme, yönlendirilmiş bozunma ve ribozom kurtarma için SsrA-SmpB sistemi". Doğa Yapısal Biyoloji. 7 (6): 449–55. doi:10.1038/75843. PMID 10881189.
- ^ a b c d Doma MK, Parker R (Mart 2006). "Ökaryotik mRNA'ların translasyon uzamasında stall'larla birlikte Endonükleolitik bölünmesi". Doğa. 440 (7083): 561–4. Bibcode:2006Natur.440..561D. doi:10.1038 / nature04530. PMC 1839849. PMID 16554824.
- ^ Kobayashi K, Kikuno I, Kuroha K, Saito K, Ito K, Ishitani R, Inada T, Nureki O (Ekim 2010). "Archaeal Pelota ve GTP'ye bağlı EF1α kompleksi tarafından mRNA gözetiminin yapısal temeli". Amerika Birleşik Devletleri Ulusal Bilimler Akademisi Bildirileri. 107 (41): 17575–9. Bibcode:2010PNAS..10717575K. doi:10.1073 / pnas.1009598107. PMC 2955123. PMID 20876129.
- ^ Graille M, Chaillet M, van Tilbeurgh H (Mart 2008). "Dom34 maya yapısı: çeviri sonlandırma faktörü Erf1 ile ilişkili ve No-Go bozunmasına dahil olan bir protein". Biyolojik Kimya Dergisi. 283 (11): 7145–54. doi:10.1074 / jbc.M708224200. PMID 18180287.
- ^ a b Atkinson GC, Baldauf SL, Hauryliuk V (Ekim 2008). "Kesintisiz, gitmeyen ve anlamsız aracılı mRNA bozunmasının evrimi ve bunların sonlandırma faktöründen türetilen bileşenleri". BMC Evrimsel Biyoloji. 8: 290. doi:10.1186/1471-2148-8-290. PMC 2613156. PMID 18947425.