Tıbbi görüntü hesaplama - Medical image computing
Tıbbi görüntü hesaplama (MIC) disiplinlerarası bir alandır kesiştiği noktada bilgisayar Bilimi, Bilgi Mühendisliği, elektrik Mühendisliği, fizik, matematik ve ilaç. Bu alan, tıbbi görüntülere ve bunların biyomedikal araştırma ve klinik bakım için kullanımına ilişkin problemleri çözmek için hesaplamalı ve matematiksel yöntemler geliştirir.
MIC'in temel amacı, klinik olarak ilgili bilgileri veya bilgileri tıbbi görüntülerden çıkarmaktır. Alanı ile yakından ilgili olsa da tıbbi Görüntüleme MIC, görüntülerin elde edilmesine değil hesaplamalı analizine odaklanır. Yöntemler birkaç geniş kategoriye ayrılabilir: Resim parçalama, Görüntü kaydı, görüntü tabanlı fizyolojik modelleme, ve diğerleri.
Veri formları
Tıbbi görüntü hesaplama, tipik olarak, düzenli x-y-z uzamsal aralıklarla (2D'de görüntüler ve 3D'de, genel olarak görüntüler olarak anılır) tek tip örneklenmiş veriler üzerinde çalışır. Her örnek noktada, veriler genellikle şu şekilde temsil edilir: integral işaretsiz ve işaretsiz kısa (16-bit) gibi formlar, ancak işaretsiz karakterden (8-bit) 32-bit float'a kadar olan formlar nadir değildir. Örnek noktadaki verilerin özel anlamı modaliteye bağlıdır: örneğin a CT edinimi radyodensite değerlerini toplar, bir MRI edinimi toplayabilir T1 veya T2 ağırlıklı görüntüler. Boylamsal, zamanla değişen çekimler, normal zaman adımlarıyla görüntüler alabilir veya almayabilir. Gibi modaliteler nedeniyle hayran benzeri görüntüler kavisli dizi ultrason ayrıca yaygındır ve işlemek için farklı temsil ve algoritmik teknikler gerektirir. Diğer veri formları aşağıdakilerden dolayı kesilmiş görüntüleri içerir: portal eğimi edinim sırasında; ve yapılandırılmamış ağlar, ileri düzeylerde kullanılan altı yüzlü ve dört yüzlü formlar gibi biyomekanik analiz (örneğin, doku deformasyonu, vasküler taşıma, kemik implantları).
Segmentasyon
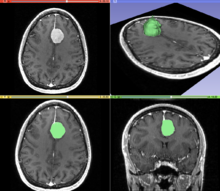
Segmentasyon, bir görüntüyü farklı anlamlı bölümlere ayırma işlemidir. Tıbbi görüntülemede, bu segmentler genellikle farklı doku sınıflarına karşılık gelir, organlar, patolojiler veya biyolojik olarak ilgili diğer yapılar.[1] Medikal görüntü segmentasyonu, düşük kontrast, gürültü ve diğer görüntüleme belirsizlikleri nedeniyle zorlaştırılır. Çok olmasına rağmen görüntü bölütleme için bilgisayarla görme teknikleri bazıları tıbbi görüntü hesaplama için özel olarak uyarlanmıştır. Aşağıda bu alandaki tekniklerden bir örnek verilmiştir; uygulama, klinisyenlerin sağlayabileceği uzmanlığa dayanır.
- Atlas Tabanlı Segmentasyon: Birçok uygulama için, bir klinik uzmanı birkaç görüntüyü manuel olarak etiketleyebilir; Görünmeyen görüntüleri bölümlere ayırmak, bu manuel olarak etiketlenmiş eğitim görüntülerinden çıkarım yapma meselesidir. Bu tarzın yöntemleri tipik olarak atlas bazlı bölümleme yöntemleri olarak adlandırılır. Parametrik atlas yöntemleri tipik olarak bu eğitim görüntülerini tek bir atlas görüntüsünde birleştirir,[2] parametrik olmayan atlas yöntemleri tipik olarak tüm eğitim görüntülerini ayrı ayrı kullanır.[3] Atlas tabanlı yöntemler genellikle şunları gerektirir: Görüntü kaydı atlas görüntüsünü veya görüntüleri yeni, görünmeyen bir görüntüye hizalamak için.
- Şekil Bazlı Segmentasyon: Çoğu yöntem, belirli bir yapı için bir şablon şeklini parametrize eder ve genellikle sınır boyunca kontrol noktalarına dayanır. Tüm şekil daha sonra yeni bir görüntüyle eşleşecek şekilde deforme edilir. En yaygın şekil tabanlı tekniklerden ikisi Aktif Şekil Modelleridir [4] ve Aktif Görünüm Modelleri.[5] Bu yöntemler çok etkili olmuş ve benzer modellere yol açmıştır.[6]
- Görüntü Tabanlı segmentasyon: Bazı yöntemler bir şablonu başlatır ve şeklini görüntü verilerine göre rafine ederken, tümleşik hata önlemlerini en aza indirirken, örneğin Aktif kontur modeli ve çeşitleri.[7]
- Etkileşimli Segmentasyon: Etkileşimli yöntemler, klinisyenler bir tohum bölgesi veya bölümlere ayrılacak bölgenin kaba taslağı gibi bazı bilgiler sağlayabildiğinde yararlıdır. Daha sonra bir algoritma, klinisyenin rehberliği olsun veya olmasın, böyle bir segmentasyonu yinelemeli olarak iyileştirebilir. Her pikselin doku sınıfını açık bir şekilde tanımlamak için boya fırçası gibi araçlar kullanan manuel bölümleme, birçok görüntüleme uygulaması için altın standart olmaya devam ediyor. Son zamanlarda, geribildirimden alınan ilkeler kontrol teorisi kullanıcıya çok daha fazla esneklik sağlayan ve hataların otomatik olarak düzeltilmesine olanak tanıyan segmentasyona dahil edilmiştir.[8]
- Öznel yüzey Segmentasyonu: Bu yöntem, bir adveksiyon-difüzyon modeli tarafından yönetilen bölümleme fonksiyonunun evrimi fikrine dayanmaktadır.[9] Bir nesneyi bölümlere ayırmak için, bir bölümleme çekirdeğine ihtiyaç vardır (bu, nesnenin görüntüdeki yaklaşık konumunu belirleyen başlangıç noktasıdır). Sonuç olarak, bir başlangıç bölümleme işlevi oluşturulur. Öznel yüzey yönteminin arkasındaki fikir [10][11][12] tohumun pozisyonunun, bu segmentasyon fonksiyonunun şeklini belirleyen ana faktör olmasıdır.
Bununla birlikte, yukarıdaki kategorilere benzer başka görüntü bölümleme yöntemleri sınıflandırması da vardır. Ayrıca yöntemlerin kombinasyonuna dayanan başka bir grubu "Hibrit" olarak sınıflandırabiliriz.[13]
Kayıt
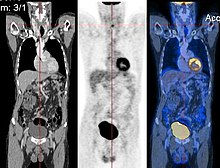
Görüntü kaydı görüntülerin doğru hizalanmasını arayan bir işlemdir.[14][15][16][17] En basit durumda, iki görüntü hizalanır. Tipik olarak, bir görüntü hedef görüntü olarak, diğeri ise kaynak görüntü olarak değerlendirilir; kaynak görüntü, hedef görüntüyle eşleşecek şekilde dönüştürülür. optimizasyon prosedürü hizalamanın mevcut kalitesini değerlendiren benzerlik değerine dayalı olarak kaynak görüntünün dönüşümünü günceller. Bu yinelemeli prosedür, (yerel) bir optimum bulunana kadar tekrar edilir. Bir örnek CT'nin kaydıdır ve EVCİL HAYVAN yapısal ve metabolik bilgileri birleştiren görüntüler (şekle bakın).
Görüntü kaydı çeşitli tıbbi uygulamalarda kullanılır:
- Zamansal değişiklikleri incelemek. Boylamsal çalışmalar hastalık ilerlemesi gibi uzun vadeli süreçleri incelemek için birkaç ay veya yıl boyunca görüntüler elde edin. Zaman serisi aynı oturumda elde edilen görüntülere karşılık gelir (saniye veya dakika). Bilişsel süreçleri, kalp deformasyonlarını ve solunumu incelemek için kullanılabilirler.
- Farklı kaynaklardan gelen tamamlayıcı bilgileri birleştirmek görüntüleme yöntemleri. Bir örnek, anatomik ve fonksiyonel bilgilerin kaynaşmasıdır. Yapıların boyutu ve şekli modalitelere göre değişiklik gösterdiğinden, hizalama kalitesini değerlendirmek daha zordur. Bu, kullanımına yol açtı benzerlik ölçüleri gibi karşılıklı bilgi.[18]
- Bir denek popülasyonunu karakterize etmek. Konu içi kaydın aksine, ilgilenilen organın yapısal değişkenliğine bağlı olarak konular arasında bire bir haritalama olmayabilir. Atlas inşası için konular arası kayıt gereklidir. hesaplamalı anatomi.[19] Burada amaç, denekler arasında organların anatomisini istatistiksel olarak modellemektir.
- Bilgisayar destekli cerrahi. Bilgisayar destekli ameliyatta CT veya MRI gibi ameliyat öncesi görüntüler, görüntü rehberliğini veya navigasyonu kolaylaştırmak için ameliyat içi görüntülere veya izleme sistemlerine kaydedilir.
Görüntü kaydı yapılırken birkaç önemli husus vardır:
- dönüşüm modeli. Ortak seçenekler katı, afin, ve deforme olabilir dönüşüm modelleri. B-spline ve ince levha eğri modeller genellikle parametreleştirilmiş dönüşüm alanları için kullanılır. Parametrik olmayan veya yoğun deformasyon alanları, her ızgara konumunda bir yer değiştirme vektörü taşır; bu ek gerektirir düzenleme kısıtlamalar. Belirli bir deformasyon alanı sınıfı diffeomorfizmler, pürüzsüz bir tersi olan tersinir dönüşümler.
- Benzerlik ölçüsü. Kayıt kalitesini ölçmek için bir mesafe veya benzerlik işlevi kullanılır. Bu benzerlik, orijinal görüntülerde veya görüntülerden çıkarılan özelliklerde hesaplanabilir. Ortak benzerlik ölçüleri, kare mesafelerin toplamıdır (SSD), korelasyon katsayısı, ve karşılıklı bilgi. Benzerlik ölçüsü seçimi, görüntülerin aynı modaliteden olup olmadığına bağlıdır; edinim gürültüsü de bu kararda rol oynayabilir. Örneğin, SSD, aynı modaliteye sahip görüntüler için optimum benzerlik ölçüsüdür. Gauss gürültüsü.[20] Bununla birlikte, ultrasondaki görüntü istatistikleri, Gauss gürültüsünden önemli ölçüde farklıdır ve bu da ultrasona özgü benzerlik önlemlerinin kullanılmasına yol açar.[21] Çok modlu kayıt, daha karmaşık bir benzerlik ölçüsü gerektirir; alternatif olarak, yapısal temsiller gibi farklı bir görüntü temsili kullanılabilir[22] veya bitişik anatomiyi kaydetme.[23][24]
- optimizasyon prosedürü. Ya sürekli veya ayrık optimizasyon gerçekleştirilir. Sürekli optimizasyon için, gradyan tabanlı optimizasyon yakınsama hızını artırmak için teknikler uygulanır.
Görselleştirme
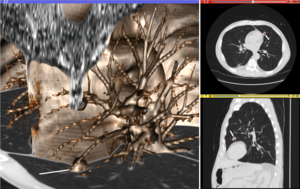
Görselleştirme, Tıbbi Görüntü Hesaplamada birkaç önemli rol oynar. Yöntemler bilimsel görselleştirme doğası gereği uzaysal-zamansal olan tıbbi görüntüleri anlamak ve bunlar hakkında iletişim kurmak için kullanılır. Veri goruntuleme ve veri analizi kullanılır yapılandırılmamış veriler formlar, örneğin algoritmik işleme sırasında elde edilen istatistiksel ölçümleri değerlendirirken. Doğrudan etkileşim görselleştirme sürecinin önemli bir özelliği olan verilerle birlikte, veriler hakkında görsel sorgular gerçekleştirmek, görüntülere açıklama eklemek, bölümleme ve kayıt işlemlerini yönlendirmek ve verilerin görsel temsilini kontrol etmek (aydınlatma oluşturma özelliklerini ve görüntüleme parametrelerini kontrol ederek) kullanılır. Görselleştirme, hem ilk keşif hem de analizlerin ara ve nihai sonuçlarını iletmek için kullanılır.
"Tıbbi Görüntülemenin Görselleştirilmesi" şekli, çeşitli görselleştirme türlerini gösterir: 1. Kesitlerin gri ölçekli görüntüler olarak görüntülenmesi; 2. Gri ölçekli görüntülerin yeniden biçimlendirilmiş görünümleri (bu örnekteki sagital görünüm, görüntü edinmenin orijinal yönünden farklı bir yönlendirmeye sahiptir; ve 3. A 3B hacim oluşturma aynı verilerin. Nodüler lezyon, farklı sunumlarda açıkça görülebilir ve beyaz bir çizgi ile açıklanmıştır.
Atlaslar
Tıbbi görüntüler, farklı şekil ve boyutlarda organlara sahip kişilerden dolayı bireyler arasında önemli ölçüde değişebilir. Bu nedenle, bu değişkenliği açıklamak için tıbbi görüntüleri temsil etmek çok önemlidir. Tıbbi görüntüleri temsil etmeye yönelik popüler bir yaklaşım, bir veya daha fazla atlasın kullanılmasıdır. Burada, bir atlas, bir eğitim veri kümesinden öğrenilen parametrelere sahip bir görüntü popülasyonu için belirli bir modeli ifade eder.[25][26]
Bir atlasın en basit örneği, genellikle şablon olarak adlandırılan ortalama yoğunluklu bir görüntüdür. Bununla birlikte, bir atlas, yerel görüntü istatistikleri ve belirli bir uzamsal konumun belirli bir etikete sahip olma olasılığı gibi daha zengin bilgileri de içerebilir. Eğitim sırasında kullanılmayan yeni tıbbi görüntüler, aşağıdaki gibi belirli bir uygulamaya uyarlanmış bir atlasla eşleştirilebilir. segmentasyon ve grup analizi. Bir görüntüyü bir atlasla eşleştirmek genellikle şunları içerir: kayıt görüntü ve atlas. Bu deformasyon, tıbbi görüntülerde değişkenliği ele almak için kullanılabilir.
Tek şablon
En basit yaklaşım, tıbbi görüntüleri tek bir şablon görüntünün deforme olmuş versiyonları olarak modellemektir. Örneğin, anatomik MRI beyin taramaları genellikle MNI şablonuna eşlenir [27] tüm beyin taramalarını ortak koordinatlarda temsil edecek şekilde. Tek şablon yaklaşımının ana dezavantajı, şablon ile belirli bir test görüntüsü arasında önemli farklılıklar varsa, birini diğerine eşlemek için iyi bir yol olmayabileceğidir. Örneğin, şiddetli beyin anormallikleri (yani, bir tümör veya cerrahi prosedür) olan bir hastanın anatomik MRI beyin taraması, MNI şablonuna kolayca eşlenemeyebilir.
Birden çok şablon
Tek bir şablona güvenmek yerine, birden çok şablon kullanılabilir. Buradaki fikir, bir görüntüyü şablonlardan birinin deforme olmuş bir versiyonu olarak temsil etmektir. Örneğin, sağlıklı bir popülasyon için bir şablon ve hastalıklı bir popülasyon için bir şablon olabilir. Ancak birçok uygulamada kaç şablona ihtiyaç duyulduğu net değildir. Bununla baş etmenin basit ama hesaplama açısından pahalı bir yolu, bir eğitim veri kümesindeki her görüntünün bir şablon görüntüsü olması ve böylece karşılaşılan her yeni görüntünün eğitim veri kümesindeki her görüntü ile karşılaştırılmasıdır. Daha yeni bir yaklaşım, gereken şablon sayısını otomatik olarak bulur.[28]
istatistiksel analiz
İstatistiksel yöntemler, tıbbi görüntüleme alanını modern ile birleştirir Bilgisayar görüşü, Makine öğrenme ve Desen tanıma. Son on yılda, kısmen çeşitli enstitüler ve araştırma merkezleri arasındaki işbirliği nedeniyle, birkaç büyük veri kümesi kamuya açık hale getirildi (örneğin ADNI, 1000 fonksiyonel Connectomes Projesi). Veri boyutundaki bu artış, klinik soruları ele almak için görüntülerdeki ince değişiklikleri araştırıp tespit edebilen yeni algoritmalar gerektirir. Bu tür klinik sorular çok çeşitlidir ve grup analizi, görüntüleme biyobelirteçleri, hastalık fenotiplemesi ve uzunlamasına çalışmaları içerir.
Grup analizi
Grup Analizinde amaç, iki veya daha fazla kohortun görüntülerini karşılaştırarak bir hastalığın neden olduğu anormallikleri tespit etmek ve nicelendirmektir. Genellikle bu kohortlardan biri normal (kontrol) deneklerden, diğeri ise anormal hastalardan oluşur. Hastalığın neden olduğu varyasyon, anormal anatomi deformasyonu olarak kendini gösterebilir (bkz. Voksel tabanlı morfometri ). Örneğin, subkortikal dokuların küçülmesi gibi Hipokamp beyinde bağlantılı olabilir Alzheimer hastalığı. Ek olarak, biyokimyasal (fonksiyonel) aktivitede değişiklikler, aşağıdaki gibi görüntüleme yöntemleri kullanılarak gözlemlenebilir. Pozitron emisyon tomografi.
Gruplar arasındaki karşılaştırma genellikle voksel seviyesi. Bu nedenle, en popüler ön işleme hattı, özellikle nöro-görüntüleme, bir veri kümesindeki tüm görüntüleri ortak bir koordinat çerçevesine (Tıbbi Görüntü Kaydı ) vokseller arasındaki yazışmayı sürdürmek için. Bu voksel bazlı yazışma göz önüne alındığında, en yaygın olanı Sık görüşen yöntem, her voksel için bir istatistik çıkarmaktır (örneğin, her grup için ortalama voksel yoğunluğu) ve istatistiksel hipotez testi boş bir hipotezin desteklenip desteklenmediğini değerlendirmek için. Boş hipotez tipik olarak iki kohortun aynı dağılımdan çekildiğini ve dolayısıyla aynı istatistiksel özelliklere sahip olması gerektiğini varsayar (örneğin, iki grubun ortalama değerleri belirli bir voksel için eşittir). Tıbbi görüntüler çok sayıda voksel içerdiğinden, çoklu karşılaştırma ele alınması gerekiyor.[29][30] Ayrıca orada Bayes grup analizi probleminin üstesinden gelmek için yaklaşımlar.[31]
Sınıflandırma
Grup analizi bir patolojinin bir anatomi ve işlev üzerindeki genel etkilerini ölçebilmesine rağmen, konu düzeyinde ölçümler sağlamaz ve bu nedenle tanı için biyobelirteçler olarak kullanılamaz (bkz. Görüntüleme Biyobelirteçleri). Öte yandan klinisyenler genellikle patolojinin erken teşhisi ile ilgilenirler (yani sınıflandırma,[32][33]) ve bir hastalığın ilerlemesini öğrenirken (yani gerileme [34]). Metodolojik bakış açısından, mevcut teknikler, standart makine öğrenimi algoritmalarının uygulanmasından tıbbi görüntüleme veri setlerine (ör. Destek Vektör Makinesi [35]), alanın ihtiyaçlarına göre uyarlanmış yeni yaklaşımlar geliştirmek.[36] Ana zorluklar aşağıdaki gibidir:
- Küçük örnek boyutu (Boyutluluk Laneti ): büyük bir tıbbi görüntüleme veri seti, yüz ila binlerce görüntü içerirken, tipik bir hacimsel görüntüdeki voksel sayısı kolayca milyonları aşabilir. Bu sorunun çaresi, bilgilendirici anlamda özelliklerin sayısını azaltmaktır (bkz. Boyutsal küçülme ). Birkaç denetimsiz ve yarı / denetimli,[36][37][38][39] bu sorunu çözmek için yaklaşımlar önerilmiştir.
- Yorumlanabilirlik: İyi bir genelleme doğruluğu her zaman birincil hedef değildir, çünkü klinisyenler anatominin hangi bölümlerinin hastalıktan etkilendiğini anlamak ister. Bu nedenle sonuçların yorumlanabilirliği çok önemlidir; görüntü yapısını göz ardı eden yöntemler tercih edilmez. Aşağıdakilere dayalı alternatif yöntemler Öznitelik Seçimi teklif edilmiştir.[37][38][39][40]
Kümeleme
Görüntü tabanlı model sınıflandırma yöntemleri tipik olarak bir hastalığın nörolojik etkilerinin farklı ve iyi tanımlanmış olduğunu varsayar. Bu her zaman böyle olmayabilir. Bir dizi tıbbi durum için, hasta popülasyonları oldukça heterojendir ve alt koşullara göre daha fazla sınıflandırma yapılmamıştır. Ek olarak, bazı hastalıklar (ör. Otizm spektrum bozukluğu (ASD), şizofreni, hafif bilişsel bozukluk (MCI)), hafif bilişsel bozukluktan çok belirgin patolojik değişikliklere kadar sürekli veya neredeyse sürekli bir spektrumla karakterize edilebilir. Heterojen bozuklukların görüntüye dayalı analizini kolaylaştırmak için, örüntü sınıflandırmasına yönelik metodolojik alternatifler geliştirilmiştir. Bu teknikler, yüksek boyutlu kümelemeden fikir alır [41] ve belirli bir popülasyonu homojen alt popülasyonlar halinde kümelemek için yüksek boyutlu model regresyonu. Amaç, her bir alt popülasyondaki hastalığın nicel olarak daha iyi anlaşılmasını sağlamaktır.
Şekil analizi
Şekil Analizi Tıbbi Görüntü Hesaplama alanıdır. geometrik farklı kaynaklardan elde edilen yapıların özellikleri görüntüleme yöntemleri. Şekil analizi, kesin olarak konumlandırma potansiyeli nedeniyle son zamanlarda tıp camiasında artan bir ilgi haline geldi morfolojik farklı yapı popülasyonları arasındaki değişiklikler, yani sağlıklı ve patolojik, kadın-erkek, genç-yaşlı. Şekil Analizi iki ana adımı içerir: şekil uyuşması ve istatistiksel analiz.
- Şekil yazışması, üçgen ağlar, konturlar, nokta kümeleri veya hacimsel görüntülerle temsil edilen geometrik şekiller arasındaki karşılık gelen konumları hesaplayan metodolojidir. Açıktır ki, yazışmanın tanımı doğrudan analizi etkileyecektir. Karşılıklı çerçeveler için farklı seçenekler arasında şunları bulabiliriz: Anatomik karşılık, manüel işaretler, işlevsel karşılık (yani aynı nöronal işlevsellikten sorumlu beyin morfometrisi lokusunda), geometri karşılığı, (görüntü hacimleri için) yoğunluk benzerliği, vb. Bazı yaklaşımlar, örn. spektral şekil analizi, yazışma gerektirmez, ancak şekil tanımlayıcılarını doğrudan karşılaştırır.
- İstatistiksel analiz, muhabir yerlerdeki yapısal değişimin ölçümlerini sağlayacaktır.
Boylamsal çalışmalar
Boylamsal çalışmalarda aynı kişi defalarca görüntülenmektedir. Bu bilgiler her ikisine de dahil edilebilir. görüntü analizi yanı sıra istatistiksel modelleme.
- Boylamsal görüntü işlemede, bireysel zaman noktalarının segmentasyon ve analiz yöntemleri, genellikle bir konu içi şablondan alınan ortak bilgilerle bilgilendirilir ve düzenlenir. Bu düzenleme, ölçüm gürültüsünü azaltmak için tasarlanmıştır ve böylece duyarlılığı ve istatistiksel gücü artırmaya yardımcı olur. Aynı zamanda, aşırı regülasyondan da kaçınılmalıdır, böylece efekt büyüklükleri sabit kalır. Örneğin yoğun bir düzenleme, mükemmel test-tekrar test güvenilirliğine yol açabilir, ancak gruplar arasında gerçek değişiklikleri ve farklılıkları algılama yeteneğini sınırlar. Çoğunlukla, sınırlı etki boyutu kaybı pahasına gürültü azaltmayı optimize eden bir değiş tokuşun hedeflenmesi gerekir. Boylamsal görüntü işlemede diğer bir yaygın zorluk, genellikle kasıtsız olarak işleme önyargısının getirilmesidir. Örneğin, takip görüntüleri kaydedildiğinde ve temel görüntüye yeniden örneklendiğinde, ara değerleme yapıları taban çizgisine değil, yalnızca takip görüntülerine dahil edilir. Bu artefakt, sahte etkilere neden olabilir (genellikle uzunlamasına değişimi abartmaya ve dolayısıyla gerekli örnek boyutunu küçümsemeye yönelik bir önyargı). Bu nedenle, herhangi bir işleme önyargısından kaçınmak için tüm zaman noktalarının tamamen aynı şekilde ele alınması önemlidir.
- Boylamsal verilerin sonradan işlenmesi ve istatistiksel analizi, genellikle tekrarlanan ölçüm ANOVA veya daha güçlü doğrusal karma efekt modelleri gibi özel istatistiksel araçlar gerektirir. Ek olarak, sinyalin uzamsal dağılımını dikkate almak avantajlıdır. Örneğin, kortikal kalınlık ölçümleri zaman içinde özne içinde ve ayrıca kortikal yüzeydeki bir mahalle içinde bir korelasyon gösterecektir - istatistiksel gücü artırmak için kullanılabilecek bir gerçek. Ayrıca, uzunlamasına verileri analiz etmek ve önemli öngörücüleri belirlemek için olaya kadar geçen zaman (aka hayatta kalma) analizi sıklıkla kullanılır.
Görüntü tabanlı fizyolojik modelleme
Geleneksel olarak, tıbbi görüntü hesaplama, görüntü edinme noktasında ve zamanında mevcut olan yapısal veya işlevsel bilgilerin nicelendirilmesi ve birleştirilmesini ele alır. Bu bağlamda, altta yatan anatomik, fiziksel veya fizyolojik süreçlerin nicel olarak algılanması olarak görülebilir. Bununla birlikte, son birkaç yılda, hastalık veya terapi seyrinin öngörücü değerlendirmesine artan bir ilgi var. Biyomekanik veya fizyolojik nitelikte olsun, görüntüye dayalı modelleme, bu nedenle görüntü hesaplama olanaklarını tanımlayıcıdan öngörücü bir açıya doğru genişletebilir.
STEP araştırma yol haritasına göre,[42][43] Sanal Fizyolojik İnsan (VPH), bir kez oluşturulduktan sonra insan vücudunun tek bir karmaşık sistem olarak incelenmesini sağlayacak metodolojik ve teknolojik bir çerçevedir. VPH konseptinin altında yatan Uluslararası Fizyolojik Bilimler Birliği (IUPS), IUPS Physiome Projesi on yıldan fazla bir süredir.[44][45] Bu, insan fizyolojisini anlamak için hesaplamalı bir çerçeve sağlamak için dünya çapında kamuya açık bir çabadır. Gen düzenleyici ağlar, protein yolları, bütünleştirici hücre işlevleri ve doku ve tüm organ yapı / işlev ilişkileri aracılığıyla genlerden tüm organizmalara kadar biyolojik organizasyonun tüm seviyelerinde bütünleştirici modeller geliştirmeyi amaçlamaktadır. Böyle bir yaklaşım, tıptaki mevcut uygulamayı dönüştürmeyi amaçlamaktadır ve yeni bir hesaplamalı tıp çağının temelini oluşturmaktadır.[46]
Bu bağlamda, tıbbi görüntüleme ve görüntü hesaplama, insan hakkında hem yapısal hem de işlevsel bilgiyi in vivo olarak görüntülemek, ölçmek ve birleştirmek için sistemler ve yöntemler sağladıkları için giderek daha önemli bir rol oynamaktadır. Bu iki geniş araştırma alanı, genel hesaplama modellerinin belirli konuları temsil edecek şekilde dönüştürülmesini içerir ve böylece kişiselleştirilmiş hesaplama modellerinin yolunu açar.[47] Görüntüleme yoluyla genel hesaplama modellerinin bireyselleştirilmesi üç tamamlayıcı yönde gerçekleştirilebilir:
- konuya özgü hesaplama alanının (anatomi) ve ilgili alt alanların (doku türleri) tanımı;
- (dinamik ve / veya fonksiyonel) görüntülemeden sınır ve başlangıç koşullarının tanımı; ve
- yapısal ve fonksiyonel doku özelliklerinin karakterizasyonu.
Ek olarak, görüntüleme aynı zamanda bu tür modellerin hem insanlarda hem de hayvan modellerinde değerlendirilmesi ve onaylanmasında ve modellerin hem tanısal hem de terapötik uygulamalarla klinik ortama çevrilmesinde önemli bir rol oynar. Bu spesifik bağlamda, moleküler, biyolojik ve klinik öncesi görüntüleme, uygun olduğunda insan fizyolojisine aktarılabilen moleküller, hücreler, dokular ve hayvan modellerinde temel yapı ve işlev hakkında ek veriler ve anlayış sağlar.
Görüntü tabanlı VPH / Physiome modellerinin temel ve klinik alanlarda uygulamaları çok geniştir. Genel olarak, yeni olma sözü veriyorlar sanal görüntüleme teknikleri. Etkili bir şekilde daha fazla, genellikle gözlemlenemeyen parametreler görüntülenecek silikoda gözlemlenebilir ancak bazen seyrek ve tutarsız multimodal görüntülerin ve fizyolojik ölçümlerin entegrasyonuna dayanır. Hesaplamalı modeller, araştırılan fizyolojik veya patofizyolojik süreçlerin temelindeki biyofiziksel, biyokimyasal veya biyolojik yasalarla uyumlu bir şekilde ölçümlerin yorumlanmasına hizmet edecektir. Nihayetinde, bu tür araştırma araçları ve sistemleri, hastalık süreçlerini, hastalığın doğal seyrini ve farmakolojik ve / veya girişimsel terapötik prosedürlerin bir hastalığın seyri üzerindeki etkisini anlamamıza yardımcı olacaktır.
Görüntüleme ve modelleme arasındaki çapraz döllenme, fizyolojiyle tutarlı bir şekilde ölçümlerin yorumlanmasının ötesine geçer. Tıbbi cihaz modelleri ve farmakolojik tedavilerle birlikte görüntü tabanlı hastaya özel modelleme, bu tür müdahaleleri anlayabilecek, planlayabilecek ve optimize edebilecek tahmini görüntülemeye giden yolu açar. silikoda.
Tıbbi görüntülemede matematiksel yöntemler
Bir dizi sofistike matematiksel yöntem tıbbi görüntülemeye girmiş ve çeşitli yazılım paketlerinde zaten uygulanmıştır. Bunlar aşağıdakilere dayalı yaklaşımları içerir: kısmi diferansiyel denklemler (PDE'ler) ve iyileştirme, bölümleme ve kayıt için eğriliğe dayalı akışlar. PDE'leri kullandıklarından, yöntemler GPGPU'larda paralelleştirme ve uygulamaya uygundur. Bu tekniklerin birçoğu, optimal kontrol. Buna göre, çok yakın zamanda, kontrolden gelen fikirler son zamanlarda etkileşimli yöntemlere, özellikle de segmentasyona girdi. Dahası, gürültü ve görüntüleri daha dinamik bir şekilde değiştirmek için istatistiksel tahmin tekniklerine duyulan ihtiyaç nedeniyle, Kalman filtresi [48] ve partikül filtresi kullanıma girmiştir. Kapsamlı bir referans listesi ile bu yöntemlerin bir araştırması bulunabilir.[49]
Modaliteye özgü bilgi işlem
Bazı görüntüleme yöntemleri çok özel bilgiler sağlar. Ortaya çıkan görüntüler normal skaler görüntüler olarak ele alınamaz ve Tıbbi Görüntü Hesaplamanın yeni alt alanlarına yol açar. Örnekler şunları içerir: difüzyon MR,fonksiyonel MR ve diğerleri.
Difüzyon MR
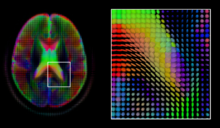
Difüzyon MR yapısal manyetik rezonans görüntüleme moleküllerin difüzyon sürecinin ölçülmesini sağlayan modalite. Difüzyon, belirli bir yön boyunca bir manyetik alana bir gradyan darbesi uygulanarak ölçülür. Tipik bir edinimde, bir dizi difüzyon ağırlıklı hacim oluşturmak için tekdüze dağıtılmış bir dizi gradyan yönü kullanılır. Ek olarak, bir gradyan darbesi uygulanmadan aynı manyetik alan altında ağırlıksız bir hacim elde edilir. Her edinme birden fazla hacimle ilişkilendirildiğinden, difüzyon MR tıbbi görüntü hesaplamada çeşitli benzersiz zorluklar yarattı.
Tıpta, iki ana hesaplama hedefi vardır. difüzyon MR:
- Difüzivite gibi lokal doku özelliklerinin tahmini;
- Yerel yönlerin ve küresel difüzyon yollarının tahmini.
difüzyon tensörü,[50] 3 × 3 simetrik pozitif tanımlı matris, bu hedeflerin her ikisine de basit bir çözüm sunar. Normal olarak dağıtılmış bir yerel difüzyon profilinin kovaryans matrisi ile orantılıdır ve bu nedenle, bu matrisin baskın özvektörü, yerel difüzyonun ana yönüdür. Bu modelin basitliğinden dolayı, difüzyon tensörünün maksimum olasılık tahmini, her konumda bağımsız olarak bir doğrusal denklem sistemi çözülerek bulunabilir. Bununla birlikte, hacmin bitişik doku lifleri içerdiği varsayıldığından, tensörlerin altta yatan alanına düzenlilik koşulları uygulayarak bütünüyle difüzyon tensörlerinin hacminin tahmin edilmesi tercih edilebilir.[51] Skaler değerler, difüzyon tensöründen çıkarılabilir, örneğin fraksiyonel anizotropi, ortalama, eksenel ve radyal yayınımlar, dolaylı olarak aksonal liflerin dismiyelinasyonu gibi doku özelliklerini ölçmek [52] veya ödem varlığı.[53] Kayıt ve bölümleme gibi standart skaler görüntü hesaplama yöntemleri, bu tür skaler değerlerin hacimlerine doğrudan uygulanabilir. Bununla birlikte, difüzyon tensöründeki bilgilerden tam olarak yararlanmak için bu yöntemler, kayıt yapılırken tensör değerli hacimleri hesaba katacak şekilde uyarlanmıştır. [54][55] ve segmentasyon.[56][57]
Hacimdeki her konumdaki temel difüzyon yönü göz önüne alındığında, küresel difüzyon yollarını şu şekilde bilinen bir süreçle tahmin etmek mümkündür: traktografi.[58] Ancak, nispeten düşük çözünürlük nedeniyle difüzyon MR Bu yolların çoğu tek bir yerde kesişebilir, öpebilir veya yelpaze olabilir. Bu durumda, tek ana yönü difüzyon tensörü yerel difüzyon dağılımı için uygun bir model değildir. Bu sorunun en yaygın çözümü, daha karmaşık modeller kullanarak yerel difüzyonun çoklu yönlerini tahmin etmektir. Bunlar, difüzyon tensörlerinin karışımlarını içerir,[59] Q-ball görüntüleme,[60] difüzyon spektrum görüntüleme [61] ve elyaf oryantasyon dağıtım fonksiyonları,[62][63] tipik olarak gerektiren HARDI çok sayıda gradyan yönü ile edinim. Difüzyon tensöründe olduğu gibi, bu karmaşık modellerle değerlenen hacimler, görüntü hesaplama yöntemlerini uygularken özel işlem gerektirir. kayıt [64][65][66] ve segmentasyon.[67]
Fonksiyonel MR
Fonksiyonel manyetik rezonans görüntüleme (fMRI) yerel bölgeyi gözlemleyerek sinirsel aktiviteyi dolaylı olarak ölçen tıbbi bir görüntüleme yöntemidir. hemodinamik veya kan oksijen seviyesine bağlı sinyal (BOLD). fMRI verileri bir dizi bilgi sunar ve kabaca iki kategoriye ayrılabilir:
- Görevle ilgili fMRI denek bir dizi zamanlanmış deneysel koşul gerçekleştirirken edinilir. Blok tasarım deneylerinde, koşullar kısa süreler için (örneğin, 10 saniye) mevcuttur ve dinlenme süreleri ile değiştirilir. Olayla ilgili deneyler, rastgele bir uyarı dizisine dayanır ve her koşulu belirtmek için tek bir zaman noktası kullanır. Görevle ilgili fMRI'yi analiz etmek için standart yaklaşım, genel doğrusal model (GLM) [68]
- Dinlenme durumu fMRI herhangi bir deneysel görevin yokluğunda elde edilir. Tipik olarak amaç, beynin içsel ağ yapısını incelemektir. Dinlenme sırasında yapılan gözlemler, kodlama veya yansıtma gibi belirli bilişsel süreçlerle de ilişkilendirilmiştir. Dinlenme durumu fMRI ile ilgili çoğu çalışma, fMRI sinyalinin (LF-BOLD) düşük frekanslı dalgalanmalarına odaklanır. Seminal keşifler şunları içerir: varsayılan ağ,[69] kapsamlı bir kortikal parselasyon,[70] ve ağ özelliklerinin davranışsal parametrelere bağlanması.
Fonksiyonel nörogörüntüleme verilerini analiz etmek için kullanılan zengin bir metodoloji seti vardır ve genellikle bu konuda fikir birliği yoktur. en iyi yöntem. Bunun yerine, araştırmacılar her probleme bağımsız olarak yaklaşır ve uygun bir model / algoritma seçerler. Bu bağlamda, aralarında nispeten aktif bir alışveriş var sinirbilim, hesaplamalı biyoloji, İstatistik, ve makine öğrenme topluluklar. Öne çıkan yaklaşımlar arasında
- Büyük tek değişkenli yaklaşımlar görüntüleme verilerindeki bireysel vokselleri deney koşuluyla bir ilişki için araştıran. Temel yaklaşım, genel doğrusal model (GLM) [68]
- Çok değişkenli ve sınıflandırıcı tabanlı yaklaşımlar, genellikle çoklu voksel desen analizi veya çok değişkenli desen analizi olarak adlandırılan, deneysel bir duruma genel ve potansiyel olarak dağıtılmış yanıtlar için verileri araştırır. Kullanılan erken yaklaşımlar destek vektör makineleri (SVM) görsel uyaranlara verilen tepkileri incelemek.[71] Son zamanlarda, alternatif örüntü tanıma algoritmaları araştırılmıştır, örneğin rastgele orman tabanlı gini kontrastı [72] veya seyrek regresyon ve sözlük öğrenme [73]
- Fonksiyonel bağlantı analizi Bölgeler arasındaki etkileşimler dahil olmak üzere beynin içsel ağ yapısını inceler. Bu tür çalışmaların çoğu, beyni bölümlere ayırmak için dinlenme durumu verilerine odaklanır. [70] veya davranışsal ölçülerle ilişkili bulmak için.[74] Göreve özgü veriler, beyin bölgeleri arasındaki nedensel ilişkileri incelemek için kullanılabilir (ör. Dinamik nedensel haritalama (DCM)) [75]).
Geniş konu grupları ile çalışırken, bireysel konuların ortak bir referans çerçevesine normalleştirilmesi (kayıt edilmesi) çok önemlidir. Anatomiye dayalı normalleştirme gerçekleştirmek için bir dizi çalışma ve araç mevcuttur (FSL, FreeSurfer, SPM ). Konular arasındaki mekansal değişkenliği hesaba katan uyum, daha yeni bir çalışma alanıdır. Örnekler, korteksin fMRI sinyal korelasyonuna göre hizalanmasıdır.[76] hem görev hem de dinlenme durumu verilerinde global fonksiyonel bağlantı yapısına dayalı hizalama,[77] ve bireysel voksellerin uyarıcıya özgü aktivasyon profillerine dayalı hizalama.[78]
Yazılım
Tıbbi görüntü hesaplama yazılımı, IO, görselleştirme ve etkileşim, kullanıcı arabirimi, veri yönetimi ve hesaplama sağlayan karmaşık bir sistem kombinasyonudur. Tipik olarak sistem mimarileri, algoritma geliştiricilerine, uygulama geliştiricilerine ve kullanıcılara hizmet verecek şekilde katmanlanır. The bottom layers are often libraries and/or toolkits which provide base computational capabilities; while the top layers are specialized applications which address specific medical problems, diseases, or body systems.
Ek Notlar
Medical Image Computing is also related to the field of Bilgisayar görüşü. An international society, MICCAI Topluluğu represents the field and organizes an annual conference and associated workshops. Proceedings for this conference are published by Springer in the Lecture Notes in Computer Science series.[79] In 2000, N. Ayache and J. Duncan reviewed the state of the field.[80]
Ayrıca bakınız
Referanslar
- ^ Forghani, M.; Forouzanfar, M.; Teshnehlab, M. (2010). "Parameter optimization of improved fuzzy c-means clustering algorithm for brain MR image segmentation". Engineering Applications of Artificial Intelligence. 23 (2): 160–168. doi:10.1016/j.engappai.2009.10.002.
- ^ J Gee; M Reivich; R Bajcsy (1993). "Elastically Deforming a Three-Dimensional Atlas to Match Anatomical Brain Images". Bilgisayar Destekli Tomografi Dergisi. 17 (1): 225–236. doi:10.1097/00004728-199303000-00011. PMID 8454749.
- ^ MR Sabuncu; BT Yeo; K Van Leemput; B Fischl; P Golland (June 2010). "A Generative Model for Image Segmentation Based on Label Fusion". Tıbbi Görüntülemede IEEE İşlemleri. 29 (10): 1714–1729. doi:10.1109/TMI.2010.2050897. PMC 3268159. PMID 20562040.
- ^ Cootes TF, Taylor CJ, Cooper DH, Graham J (1995). "Active shape models-their training and application". Bilgisayarla Görme ve Görüntü Anlama. 61 (1): 38–59. doi:10.1006/cviu.1995.1004.
- ^ Cootes, T.F.; Edwards, G.J.; Taylor, C.J. (2001). "Active appearance models". Örüntü Analizi ve Makine Zekası Üzerine IEEE İşlemleri. 23 (6): 681–685. CiteSeerX 10.1.1.128.4967. doi:10.1109/34.927467.
- ^ G. Zheng; S. Li; G. Szekely (2017). Statistical Shape and Deformation Analysis. Akademik Basın. ISBN 9780128104941.
- ^ R. Goldenberg, R. Kimmel, E. Rivlin, and M. Rudzsky (2001). "Fast geodesic active contours" (PDF). Görüntü İşlemede IEEE İşlemleri. 10 (10): 1467–1475. Bibcode:2001ITIP...10.1467G. CiteSeerX 10.1.1.35.1977. doi:10.1109/83.951533. PMID 18255491.CS1 bakım: birden çok isim: yazarlar listesi (bağlantı)
- ^ Karasev, P.; Kolesov I.; Chudy, K.; Vela, P.; Tannenbaum, A. (2011). Interactive MRI segmentation with controlled active vision. Proceedings of the Conference on Decision and Control. pp. 2293–2298. doi:10.1109/CDC.2011.6161453. ISBN 978-1-61284-801-3. PMC 3935399. PMID 24584213.
- ^ K. Mikula, N. Peyriéras, M. Remešíková, A.Sarti: 3D embryogenesis image segmentation by the generalized subjective surface method using the finite volume technique. Proceedings of FVCA5 – 5th International Symposium on Finite Volumes for ComplexApplications, Hermes Publ., Paris 2008.
- ^ A. Sarti, G. Citti: Subjective surfaces and Riemannian mean curvature flow graphs.Acta Math. Üniv. Comenian. (N.S.) 70 (2000), 85–103.
- ^ A. Sarti, R. Malladi, J.A. Sethian: Subjective Surfaces: A Method for Completing Missing Boundaries. Proc. Nat. Acad. Sci. mi 12, No. 97 (2000), 6258–6263.
- ^ A. Sarti, R. Malladi, J.A. Sethian: Subjective Surfaces: A Geometric Model for Boundary Completion, International Journal of Computer Vision, mi 46, No. 3 (2002), 201–221.
- ^ Ehsani Rad, Abdolvahab; Mohd Rahim Mohd Shafry; Rehman Amjad; Altameem Ayman; Saba Tanzila (May 2013). "Evaluation of Current Dental Radiographs Segmentation Approaches in Computer-aided Applications". IETE Teknik İncelemesi. 30 (3): 210. doi:10.4103/0256-4602.113498.
- ^ Lisa Gottesfeld Brown (1992). "Görüntü kayıt tekniklerinin incelenmesi". ACM Hesaplama Anketleri. 24 (4): 325–376. CiteSeerX 10.1.1.35.2732. doi:10.1145/146370.146374.
- ^ J. Maintz; M. Viergever (1998). "A survey of medical image registration". Tıbbi Görüntü Analizi. 2 (1): 1–36. CiteSeerX 10.1.1.46.4959. doi:10.1016/S1361-8415(01)80026-8. PMID 10638851.
- ^ J. Hajnal; D. Hawkes; D. Hill (2001). Medical Image Registration. Baton Rouge, Florida: CRC Press.
- ^ Barbara Zitová; Jan Flusser (2003). "Image registration methods: a survey". Image Vision Comput. 21 (11): 977–1000. doi:10.1016/S0262-8856(03)00137-9. hdl:10338.dmlcz/141595.
- ^ J. P. W. Pluim; J. B. A. Maintz; M. A. Viergever (2003). "Mutual information based registration of medical images: A survey". IEEE Trans. Med. Görüntüleme. 22 (8): 986–1004. CiteSeerX 10.1.1.197.6513. doi:10.1109/TMI.2003.815867. PMID 12906253.
- ^ Grenander, Ulf; Miller, Michael I. (1998). "Computational anatomy: an emerging discipline". Q. Appl. Matematik. LVI (4): 617–694. doi:10.1090 / qam / 1668732.
- ^ P. A. Viola (1995). Alignment by Maximization of Mutual Information (Tez). Massachusetts Teknoloji Enstitüsü.
- ^ C. Wachinger; T. Klein; N. Navab (2011). "Locally adaptive Nakagami-based ultrasound similarity measures". Ultrasonik. 52 (4): 547–554. doi:10.1016/j.ultras.2011.11.009. PMID 22197152.
- ^ C. Wachinger; N. Navab (2012). "Entropy and Laplacian images: structural representations for multi-modal registration". Tıbbi Görüntü Analizi. 16 (1): 1–17. doi:10.1016/j.media.2011.03.001. PMID 21632274.
- ^ Hill, Derek LG; Hawkes, David J (1994-04-01). "Medical image registration using knowledge of adjacency of anatomical structures". Görüntü ve Görüntü Hesaplama. 12 (3): 173–178. CiteSeerX 10.1.1.421.5162. doi:10.1016/0262-8856(94)90069-8. ISSN 0262-8856.
- ^ Toth, Daniel; Panayiotou, Maria; Brost, Alexander; Behar, Jonathan M.; Rinaldi, Christopher A.; Rhode, Kawal S.; Mountney, Peter (2016-10-17). Registration with Adjacent Anatomical Structures for Cardiac Resynchronization Therapy Guidance. Statistical Atlases and Computational Models of the Heart. Imaging and Modelling Challenges (Gönderilen makale). Bilgisayar Bilimi Ders Notları. pp. 127–134. doi:10.1007/978-3-319-52718-5_14. ISBN 9783319527178.
- ^ M. De Craene; A. B. d Aische; B. Macq; S. K. Warfield (2004). "Multi-subject registration for unbiased statistical atlas construction" (PDF). Proceedings of Medical Image Computing and Computer-Assisted Intervention 2004. Bilgisayar Bilimi Ders Notları. 3216: 655–662. doi:10.1007/978-3-540-30135-6_80. ISBN 978-3-540-22976-6.
- ^ C. J. Twining; T. Cootes; S. Marsland; V. Petrovic; R. Schestowitz; C. Taylor (2005). "A unified information-theoretic approach to groupwise non-rigid registration and model building". Proceedings of Information Processing in Medical Imaging 2005. Bilgisayar Bilimi Ders Notları. 19: 1–14. doi:10.1007/11505730_1. ISBN 978-3-540-26545-0. PMID 17354680.
- ^ "The MNI brain and the Talairach atlas".
- ^ M. Sabuncu; S. K. Balci; M. E. Shenton; P. Golland (2009). "Image-driven Population Analysis through Mixture Modeling". Tıbbi Görüntülemede IEEE İşlemleri. 28 (9): 1473–1487. CiteSeerX 10.1.1.158.3690. doi:10.1109/TMI.2009.2017942. PMC 2832589. PMID 19336293.
- ^ J. Ashburner; K.J. Friston (2000). "Voxel-Based Morphometry – The Methods". NeuroImage. 11 (6): 805–821. CiteSeerX 10.1.1.114.9512. doi:10.1006 / nimg.2000.0582. PMID 10860804.
- ^ C. Davatzikos (2004). "Why voxel-based morphometric analysis should be used with great caution when characterizing group differences". NeuroImage. 23 (1): 17–20. doi:10.1016/j.neuroimage.2004.05.010. PMID 15325347.
- ^ K.J. Friston; W.D. Penny; C. Phillips; S.J. Kiebel; G. Hinton; J. Ashburner (2002). "Classical and Bayesian Inference in Neuroimaging: Theory". NeuroImage. 16 (2): 465–483. CiteSeerX 10.1.1.128.8333. doi:10.1006/nimg.2002.1090. PMID 12030832.
- ^ Yong Fan; Nematollah Batmanghelich; Chris M. Clark; Christos Davatzikos (2008). "Spatial patterns of brain atrophy in MCI patients, identified via high-dimensional pattern classification, predict subsequent cognitive decline". NeuroImage. 39 (4): 1731–1743. doi:10.1016/j.neuroimage.2007.10.031. PMC 2861339. PMID 18053747.
- ^ Rémi Cuingnet; Emilie Gerardin; Jérôme Tessieras; Guillaume Auzias; Stéphane Lehéricy; Marie-Odile Habert; Marie Chupin; Habib Benali; Olivier Colliot (2011). "The Alzheimer's Disease Neuroimaging Initiative, Automatic classification of patients with Alzheimer's disease from structural MRI: A comparison of ten methods using the ADNI database" (PDF). NeuroImage. 56 (2): 766–781. doi:10.1016/j.neuroimage.2010.06.013. PMID 20542124.
- ^ Y. Wang; Y. Fan; P. Bhatt P; C. Davatzikos (2010). "High-dimensional pattern regression using machine learning: from medical images to continuous clinical variables". NeuroImage. 50 (4): 1519–35. doi:10.1016/j.neuroimage.2009.12.092. PMC 2839056. PMID 20056158.
- ^ Benoît Magnin; Lilia Mesrob; Serge Kinkingnéhun; Mélanie Pélégrini-Issac; Olivier Colliot; Marie Sarazin; Bruno Dubois; Stéphane Lehéricy; Habib Benali (2009). "Support vector machine-based classification of Alzheimer's disease from whole-brain anatomical MRI". Nöroradyoloji. 51 (2): 73–83. doi:10.1007/s00234-008-0463-x. PMID 18846369.
- ^ a b N.K. Batmanghelich; B. Taskar; C. Davatzikos (2012). "Generative-discriminative basis learning for medical imaging". IEEE Trans Med Görüntüleme. 31 (1): 51–69. doi:10.1109/TMI.2011.2162961. PMC 3402718. PMID 21791408.
- ^ a b Glenn Fung; Jonathan Stoeckel (2007). "SVM feature selection for classification of SPECT images of Alzheimer's disease using spatial information". Bilgi ve Bilgi Sistemleri. 11 (2): 243–258. CiteSeerX 10.1.1.62.6245. doi:10.1007/s10115-006-0043-5.
- ^ a b R. Chaves; J. Ramírez; J.M. Górriz; M. López; D. Salas-Gonzalez; I. Álvarez; F. Segovia (2009). "SVM-based computer-aided diagnosis of the Alzheimer's disease using t-test NMSE feature selection with feature correlation weighting". Sinirbilim Mektupları. 461 (3): 293–297. doi:10.1016/j.neulet.2009.06.052. PMID 19549559.
- ^ a b Yanxi Liu; Leonid Teverovskiy; Owen Carmichael; Ron Kikinis; Martha Shenton; Cameron S. Carter; V. Andrew Stenger; Simon Davis; Howard Aizenstein; James T. Becker (2004). "discriminative mr image feature analysis for automatic schizophrenia and alzheimer's disease classification" (PDF). Medical Image Computing and Computer-assisted Intervention – Miccai. Bilgisayar Bilimi Ders Notları. 3216: 393–401. doi:10.1007/978-3-540-30135-6_48. ISBN 978-3-540-22976-6.
- ^ Savio A.; Graña M. (2013). "Deformation based feature selection for Computer Aided Diagnosis of Alzheimer's Disease". Uygulamalarla uzmanlık sistmeleri. 40 (5): 1619–1628. doi:10.1016/j.eswa.2012.09.009. ISSN 0957-4174.
- ^ R. Filipovych; S. M. Resnick; C. Davatzikos (2011). "Semi-supervised cluster analysis of imaging data". NeuroImage. 54 (3): 2185–2197. doi:10.1016/j.neuroimage.2010.09.074. PMC 3008313. PMID 20933091.
- ^ STEP research roadmap Arşivlendi 2008-08-28 Wayback Makinesi. europhysiome.org
- ^ J. W. Fenner; B. Brook; G. Clapworthy; P. V. Coveney; V. Feipel; H. Gregersen; D. R. Hose; P. Kohl; P. Lawford; K. M. McCormack; D. Pinney; S. R. Thomas; S. Van Sint Jan; S. Waters; M. Viceconti (2008). "The EuroPhysiome, STEP and a roadmap for the virtual physiological human" (PDF). Kraliyet Derneği'nin Felsefi İşlemleri A. 366 (1878): 2979–2999. Bibcode:2008RSPTA.366.2979F. doi:10.1098/rsta.2008.0089. PMID 18559316.
- ^ J. B. Bassingthwaighte (2000). "Strategies for the Physiome Project". Ann. Biomed. Eng. 28 (8): 1043–1058. doi:10.1114/1.1313771. PMC 3425440. PMID 11144666.
- ^ P. J. Hunter; T. K. Borg (2003). "Integration from proteins to organs: The Physiome Project". Nat. Rev. Mol. Hücre Biol. 4 (3): 237–243. doi:10.1038/nrm1054. PMID 12612642.
- ^ R. L.Winslow; N. Trayanova; D. Geman; M. I. Miller (2012). "Computational medicine: Translating models to clinical care". Sci. Trans. Orta. 4 (158): 158rv11. doi:10.1126/scitranslmed.3003528. PMC 3618897. PMID 23115356.
- ^ N. Ayache, J.-P. Boissel, S. Brunak, G. Clapworthy, G. Lonsdale, J. Fingberg, A. F. Frangi, G.Deco, P. J. Hunter, P.Nielsen, M.Halstead, D. R. Hose, I. Magnin, F. Martin-Sanchez, P. Sloot, J. Kaandorp, A. Hoekstra, S. Van Sint Jan, and M. Viceconti (2005) "Towards virtual physiological human: Multilevel modelling and simulation of the human anatomy and physiology". Directorate General INFSO & Directorate General JRC, White paper
- ^ Boulfelfel D.; Rangayyan R.M.; Hahn L.J.; Kloiber R.; Kuduvalli G.R. (1994). "Restoration of single photon emission computed tomography images by the Kalman filter". Tıbbi Görüntülemede IEEE İşlemleri. 13 (1): 102–109. doi:10.1109/42.276148. PMID 18218487.
- ^ Angenent, S.; Pichon, E.; Tannenbaum, A. (2006). "Mathematical methods in medical image processing". AMS Bülteni. 43 (3): 365–396. doi:10.1090/S0273-0979-06-01104-9. PMC 3640423. PMID 23645963.
- ^ P Basser; J Mattiello; D LeBihan (January 1994). "MR diffusion tensor spectroscopy, imaging". Biyofizik Dergisi. 66 (1): 259–267. Bibcode:1994BpJ....66..259B. doi:10.1016/S0006-3495(94)80775-1. PMC 1275686. PMID 8130344.
- ^ P Fillard; X Pennec; V Arsigny; N Ayache (2007). "Clinical DT-MRI estimation, smoothing,, fiber tracking with log-Euclidean metrics". Tıbbi Görüntülemede IEEE İşlemleri. 26 (11): 1472–1482. CiteSeerX 10.1.1.218.6380. doi:10.1109/TMI.2007.899173. PMID 18041263.
- ^ S-K Song; S-W Sun; M Ramsbottom; C Cheng; J Russell; A Cross (November 2002). "Dysmyelination Revealed through MRI as Increased Radial (but Unchanged Axial) Diffusion of Water". NeuroImage. 13 (3): 1429–1436. doi:10.1006/nimg.2002.1267. PMID 12414282.
- ^ P Barzo; A Marmarou; P Fatouros; K Hayasaki; F Corwin (December 1997). "Contribution of vasogenic and cellular edema to traumatic brain swelling measured by diffusion-weighted imaging". Nöroşirurji Dergisi. 87 (6): 900–907. doi:10.3171/jns.1997.87.6.0900. PMID 9384402.
- ^ D Alexander; C Pierpaoli; P Basser (January 2001). "Spatial transformation of diffusion tensor magnetic resonance images" (PDF). Tıbbi Görüntülemede IEEE İşlemleri. 20 (11): 1131–1139. doi:10.1109/42.963816. PMID 11700739.
- ^ Y Cao; M Miller; S Mori; R Winslow; L Younes (June 2006). "Difüzyon Tensör Görüntülerinin Diffeomorfik Eşleşmesi". Proceedings of IEEE Computer Society Conference on Computer Vision, Pattern Recognition (CVPR), Workshop on Mathematical Methods in Biomedical Image Analysis (MMBIA 2006). New York. s. 67. doi:10.1109 / CVPRW.2006.65. PMC 2920614.
- ^ Z Wang; B Vemuri (October 2005). "DTI segmentation using an information theoretic tensor dissimilarity measure". Tıbbi Görüntülemede IEEE İşlemleri. 24 (10): 1267–1277. CiteSeerX 10.1.1.464.9059. doi:10.1109/TMI.2005.854516. PMID 16229414.
- ^ Melonakos, J.; Pichon, E.; Angenent, S.; Tannenbaum, A. (2008). "Finsler active contours". IEEE Trans. PAMI. 30 (3): 412–423. doi:10.1109/TPAMI.2007.70713. PMC 2796633. PMID 18195436.
- ^ S Mori; B Crain; V Chacko; P van Zijl (February 1999). "Three-dimensional tracking of axonal projections in the brain by magnetic resonance imaging". Nöroloji Yıllıkları. 45 (2): 265–269. doi:10.1002/1531-8249(199902)45:2<265::AID-ANA21>3.0.CO;2-3. PMID 9989633.
- ^ D Tuch; T Reese; M Wiegell; N Makris; J Belliveau; V Wedeen (October 2002). "High angular resolution diffusion imaging reveals intravoxel white matter fiber heterogeneity". Tıpta Manyetik Rezonans. 48 (4): 577–582. doi:10.1002/mrm.10268. PMID 12353272.
- ^ D Tuch (December 2004). "Q-ball imaging". Tıpta Manyetik Rezonans. 52 (6): 1358–1372. doi:10.1002/mrm.20279. PMID 15562495.
- ^ V Wedeen; P Hagmann; W-Y Tseng; T Reese (December 2005). "Mapping complex tissue architecture with diffusion spectrum magnetic resonance imaging". Tıpta Manyetik Rezonans. 54 (6): 1377–1386. doi:10.1002/mrm.20642. PMID 16247738.
- ^ K Jansons; D Alexander (July 2003). "Persistent angular structure: new insights from diffusion magnetic resonance imaging data". Proceedings of Information Processing in Medical Imaging (IPMI) 2003, LNCS 2732. pp. 672–683. doi:10.1007/978-3-540-45087-0_56.
- ^ J-D Tournier; F Calamante; D Gadian; A Connelly (2007). "Direct estimation of the fiber orientation density function from diffusion-weighted MRI data using spherical deconvolution". NeuroImage. 23 (3): 1176–1185. doi:10.1016/j.neuroimage.2004.07.037. PMID 15528117.
- ^ X Geng; T Ross; W Zhan; H Gu; Y-P Chao; C-P Lin; G Christensen; N Schuff; Y Yang (July 2009). "Diffusion MRI Registration Using Orientation Distribution Functions". Proceedings of Information Processing in Medical Imaging (IPMI) 2009, LNCS 5636. 21. sayfa 626–637. doi:10.1007/978-3-642-02498-6_52. PMC 3860746.
- ^ P-T Yap; Y Chen; H An; Y Yang; J Gilmore; W Lin; D Shen (2011). "SPHERE: SPherical Harmonic Elastic REgistration of HARDI data". NeuroImage. 55 (2): 545–556. doi:10.1016/j.neuroimage.2010.12.015. PMC 3035740. PMID 21147231.
- ^ P Zhang; M Niethammer; D Shen; P-T Yap (2012). "Large Deformation Diffeomorphic Registration of Diffusion-Weighted Images" (PDF). Proceedings of Medical Image Computing and Computer-Assisted Intervention (MICCAI). doi:10.1007/978-3-642-33418-4_22.
- ^ M Descoteaux; R Deriche (September 2007). "Segmentation of Q-Ball Images Using Statistical Surface Evolution". Proceedings of Medical Image Computing and Computer-Assisted Intervention (MICCAI) 2007, LNCS 4792. pp. 769–776. doi:10.1007/978-3-540-75759-7_93.
- ^ a b Friston, K.; Holmes, A .; Worsley, K .; Poline, J.; Frith, C.; Frackowiak, R.; et al. (1995). "Statistical parametric maps in functional imaging: a general linear approach". Hum Beyin Haritası. 2 (4): 189–210. doi:10.1002/hbm.460020402.
- ^ Buckner, R. L.; Andrews-Hanna, J. R.; Schacter, D. L. (2008). "The brain's default network: anatomy, function, and relevance to disease". New York Bilimler Akademisi Yıllıkları. 1124 (1): 1–38. Bibcode:2008NYASA1124....1B. CiteSeerX 10.1.1.689.6903. doi:10.1196/annals.1440.011. PMID 18400922.
- ^ a b Yeo, B. T. T.; Krienen, F. M.; Sepulcre, J.; Sabuncu, M. R.; Lashkari, D.; Hollinshead, M.; Roffman, J. L.; Smoller, J. W.; Zöllei, L.; Polimeni, J. R.; Fischl, B.; Liu, H .; Buckner, R. L. (2011). "The organization of the human cerebral cortex estimated by intrinsic functional connectivity". J Neurophysiol. 106 (3): 1125–65. doi:10.1152/jn.00338.2011. PMC 3174820. PMID 21653723.
- ^ J. V. Haxby; M. I. Gobbini; M. L. Furey; A. Ishai; J. L. Schouten; P. Pietrini (2001). "Distributed and overlapping representations of faces and objects in ventral temporal cortex". Bilim. 293 (5539): 2425–30. Bibcode:2001Sci...293.2425H. CiteSeerX 10.1.1.381.2660. doi:10.1126/science.1063736. PMID 11577229.
- ^ Langs, G.; Menze, B. H.; Lashkari, D.; Golland, P. (2011). "Detecting stable distributed patterns of brain activation using Gini contrast". NeuroImage. 56 (2): 497–507. doi:10.1016/j.neuroimage.2010.07.074. PMC 3960973. PMID 20709176.
- ^ Varoquaux, G.; Gramfort, A.; Pedregosa, F.; Michel, V.; Thirion, B. (2011). "Multi-subject dictionary learning to segment an atlas of brain spontaneous activity". Inf Process Med Imaging. 22. pp. 562–73.
- ^ van den Heuvel, M. P.; Stam, C. J.; Kahn, R. S.; Hulshoff Pol, H. E. (2009). "Efficiency of functional brain networks and intellectual performance". J Neurosci. 29 (23): 7619–24. doi:10.1523/JNEUROSCI.1443-09.2009. PMC 6665421. PMID 19515930.
- ^ Friston, K. (2003). "Dynamic causal modelling". NeuroImage. 19 (4): 1273–1302. doi:10.1016/S1053-8119(03)00202-7. PMID 12948688.
- ^ Sabuncu, M. R.; Singer, B. D.; Conroy, B.; Bryan, R. E.; Ramadge, P. J.; Haxby, J. V. (2010). "Function-based Intersubject Alignment of Human Cortical Anatomy". Beyin zarı. 20 (1): 130–140. doi:10.1093/cercor/bhp085. PMC 2792192. PMID 19420007.
- ^ Langs, G.; Lashkari, D.; Sweet, A.; Tie, Y.; Rigolo, L.; Golby, A. J.; Golland, P. (2011). "Learning an atlas of a cognitive process in its functional geometry". Inf Process Med Imaging. 22. s. 135–46.
- ^ Haxby, J. V.; Guntupalli, J. S.; Connolly, A. C.; Halchenko, Y. O.; Conroy, B. R.; Gobbini, M. I.; Hanke, M.; Ramadge, P. J. (2011). "A common, high-dimensional model of the representational space in human ventral temporal cortex". Nöron. 72 (2): 404–416. doi:10.1016/j.neuron.2011.08.026. PMC 3201764. PMID 22017997.
- ^ Wells, William M; Colchester, Alan; Delp, Scott (1998). Bilgisayar Bilimlerinde Ders Notları (Gönderilen makale). Bilgisayar Bilimi Ders Notları. 1496. doi:10.1007/BFb0056181. ISBN 978-3-540-65136-9.
- ^ JS Duncan; N Ayache (2000). "Medical image analysis: Progress over two decades and the challenges ahead". Örüntü Analizi ve Makine Zekası Üzerine IEEE İşlemleri. 22: 85–106. CiteSeerX 10.1.1.410.8744. doi:10.1109/34.824822.
Journals on medical image computing
- Medical Image Analysis (MedIA) ; also the official journal of MICCAI Topluluğu düzenleyen Yıllık MICCAI Konferansı a premier conference for medical image computing
- IEEE Transactions on Medical Imaging (IEEE TMI)
- Tıp fiziği
- Journal of Digital Imaging (JDI) ; resmi gazetesi Society of Imaging Informatics
- Bilgisayarlı Tıbbi Görüntüleme ve Grafik
- Journal of Computer Aided Radiology and Surgery
- BMC Medical Imaging
In addition the following journals occasionally publish articles describing methods and specific clinical applications of medical image computing or modality specific medical image computing
