Deniz virüsleri - Marine viruses
| Bir dizi genel bakışın parçası |
| Deniz yaşamı |
|---|
| Bir dizinin parçası |
| Plankton |
|---|
 |
Deniz virüsleri habitatları tarafından şöyle tanımlanır: virüsler bulunanlar deniz ortamları yani tuzlu su denizlerin veya okyanusların veya acı kıyı suyu haliçler. Virüsler küçüktür bulaşıcı ajanlar bu sadece tekrarlamak yaşamın içinde hücreler bir ev sahibi organizma, çünkü bunu yapmak için ana bilgisayarın çoğaltma mekanizmasına ihtiyaçları vardır.[4] Her türden bulaşabilir yaşam formları, şuradan hayvanlar ve bitkiler -e mikroorganizmalar, dahil olmak üzere bakteri ve Archaea.[5]
Bir hücrenin içinde olmadığında veya bir hücreyi enfekte etme sürecinde olmadığında, virüsler adı verilen bağımsız parçacıklar şeklinde bulunur. Virionlar. Bir virion, bir genetik şifre (uzun moleküller şeklinde genetik bilgi taşıyan DNA veya RNA ) ile çevrili kapsid (bir protein genetik materyali koruyan kaplama). Bu virüs parçacıklarının şekilleri basitten farklı helezoni ve ikosahedral bazı virüs türleri için, diğerleri için daha karmaşık yapılar oluşturur. Virüs türlerinin çoğunda, bir optik mikroskop. Ortalama virion, ortalamanın doğrusal boyutunun yaklaşık yüzde biri kadardır. bakteri.
Bir çay kaşığı deniz suyu tipik olarak yaklaşık on milyon deniz virüsü içerir. Bu virüslerin çoğu bakteriyofajlar bulaşan ve yok eden deniz bakterileri ve büyümesini kontrol et fitoplankton dibinde deniz besin ağı. Bakteriyofajlar bitkiler ve hayvanlar için zararsızdır, ancak deniz ekosistemlerinin düzenlenmesi için gereklidir. İçin anahtar mekanizmalar sağlarlar okyanus karbonunu geri dönüştürmek ve besinler. Olarak bilinen bir süreçte viral şant Ölü bakteri hücrelerinden salınan organik moleküller taze bakteri ve alg büyümesini uyarır. Özellikle bakterilerin virüsler tarafından parçalanması (liziz ) geliştirdiği gösterilmiştir nitrojen döngüsü ve fitoplankton büyümesini teşvik eder. Viral aktivite ayrıca biyolojik pompa süreç karbon tutucular derin okyanusta. Okyanuslardaki solunum miktarını artırarak, virüsler dolaylı olarak atmosferdeki karbondioksit miktarını yaklaşık 3 oranında düşürmekten sorumludur. gigatonnes yıllık karbon miktarı
Deniz mikroorganizmaları toplamın yaklaşık% 70'ini oluşturur deniz biyokütlesi. Deniz virüslerinin her gün bu biyokütlenin% 20'sini öldürdüğü tahmin edilmektedir. Virüsler, zararlıların hızla yok edilmesinden sorumlu ana ajanlardır. alg çiçekleri çoğu zaman başkalarını öldüren Deniz yaşamı. Okyanuslardaki virüslerin sayısı, daha az sayıda konakçı organizmanın bulunduğu, açık denizde ve suyun derinliklerinde azalır. Virüsler önemli bir doğal araçtır genlerin aktarılması artan farklı türler arasında genetik çeşitlilik ve evrimi yönlendirir. Virüslerin, dünyadaki yaşamın son evrensel ortak atası zamanında bakteri, arkeler ve ökaryotların çeşitlenmesinden önce erken evrimde merkezi bir rol oynadığı düşünülüyor. Virüsler, hala dünyadaki keşfedilmemiş genetik çeşitliliğin en büyük alanlarından biridir.
Arka fon

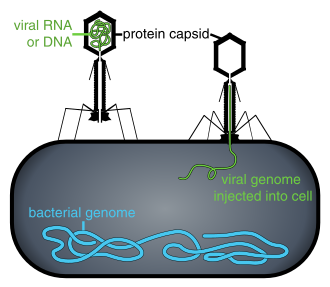
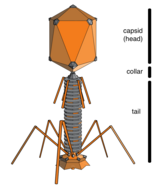
Virüsler artık kadim olarak kabul ediliyor ve yaşamın farklılaşmasından önce kökenleri var. üç alan.[6] Yaşamın olduğu her yerde bulunurlar ve muhtemelen canlı hücreler ilk evrimleştiğinden beri var olmuşlardır.[7] Virüslerin kökenleri evrimsel yaşam tarihi belirsizdir çünkü fosil oluşturmazlar. Moleküler teknikler virüslerin DNA veya RNA'sını karşılaştırmak için kullanılır ve nasıl ortaya çıktıklarını araştırmak için yararlı bir araçtır.[8] Bazı virüslerde olabilir gelişti itibaren plazmitler - hücreler arasında hareket edebilen DNA parçaları - diğerleri bakterilerden evrimleşmiş olabilir. Evrimde, virüsler önemli bir yatay gen transferi hangi artar genetik çeşitlilik.[9]
Görüşler, virüslerin bir tür hayat veya canlı organizmalarla etkileşime giren organik yapılar.[10] Bazıları tarafından bir yaşam formu olarak kabul edilirler çünkü genetik materyal taşırlar, kendiliğinden birleşerek kendilerinin birden fazla kopyasını oluşturarak çoğalırlar ve evrim geçirirler. Doğal seçilim. Bununla birlikte, genellikle yaşam sayılması için gerekli görülen hücresel yapı gibi temel özelliklerden yoksundurlar. Bu niteliklerin tümüne sahip olmamakla birlikte bazılarına sahip oldukları için, virüsler çoğalıcı olarak tanımlanmıştır.[11] ve "hayatın kıyısındaki organizmalar" olarak.[12]
Okyanustaki virüslerin varlığı, elektron mikroskobu ve epifloresan mikroskobu ekolojik su örnekleri ve daha sonra metagenomik kültürlenmemiş viral numunelerin örneklenmesi.[13][14] Deniz virüsleri, mikroskobik olmasına ve esasen Bilim insanları yakın zamana kadar, okyanustaki en bol ve çeşitli biyolojik varlıklardı. Virüslerin tahmini bolluğu 10'dur30 okyanusta veya 1 ile 100.000x10 arasında6 başına mililitre.[4] Deniz virüslerinin kantifikasyonu başlangıçta transmisyon elektron mikroskobu kullanılarak gerçekleştirildi, ancak epifloresans veya akış sitometrisi.[15]
Bakteriyofajlar

Bakteriyofajlar, genellikle sadece aradı fajlarvirüsler parazit bakteri. Deniz fajları parazit deniz bakterileri gibi siyanobakteriler.[16] Bunlar, deniz ortamlarında en bol biyolojik varlık olan çeşitli virüsler grubudur, çünkü konakçıları, bakterileri, tipik olarak denizdeki sayısal olarak baskın hücresel yaşamdır. Okyanuslarda bakteri olduğundan on kat daha fazla faj vardır.[17] mililitre deniz suyu başına 250 milyon bakteriyofaj düzeyine ulaşıyor.[18] Bu virüsler, belirli bakterilere bağlanarak enfekte eder. yüzey reseptör molekülleri ve sonra hücreye girme. Kısa bir süre içinde, bazı durumlarda sadece dakikalar içinde, bakteriyel polimeraz viral mRNA'yı proteine çevirmeye başlar. Bu proteinler ya hücre içinde yeni viryonlar, yeni viryonların birleşmesine yardımcı olan yardımcı proteinler ya da hücre lizizinde rol oynayan proteinler haline gelirler. Viral enzimler, hücre zarının parçalanmasına yardımcı olur ve enjeksiyondan yirmi dakika sonra üç yüz fajı kopyalayabilen fajlar vardır.[19]
Bakteriler, yabancı DNA'yı yok eden enzimler üreterek kendilerini bakteriyofajlardan korurlar. Bu enzimler, kısıtlama endonükleazları, bakteriyofajların bakteri hücrelerine enjekte ettiği viral DNA'yı kesin.[20] Bakteriler ayrıca kullanan bir sistem içerir CRISPR bakterilerin geçmişte temas kurduğu virüslerin genomlarının parçalarını tutmak için diziler, virüsün çoğalmasını bir şekilde engellemelerine izin verir. RNA interferansı.[21][22] Bu genetik sistem bakterilere Edinilmiş bağışıklık enfeksiyona.[23]
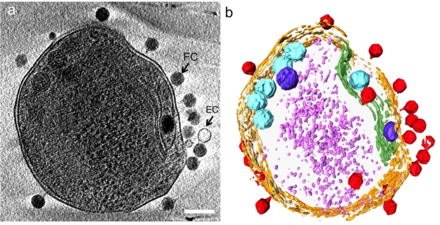
(b) hücre duvarını turuncu, plazma membranı açık sarı, tilakoid membranı yeşil, karboksizomları camgöbeği, polifosfat gövdesi mavi, hücrenin yanlarında veya üstünde adsorbe olmuş fajlar kırmızı ile vurgulanarak görselleştirilen aynı görüntü ve sitoplazmik granüller (muhtemelen çoğunlukla ribozomlar) açık mor renktedir.[24]

denizle etkileşim Proklorokok MED4 bakteri
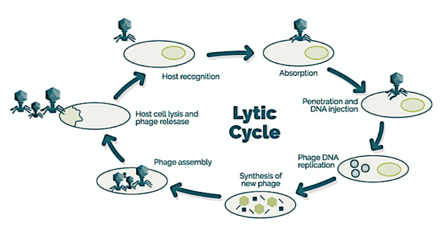
→ bağlanma: faj, kendisini konakçı hücrenin yüzeyine bağlar
→ penetrasyon: faj DNA'sını hücre zarından enjekte eder
→ transkripsiyon: konakçı hücrenin DNA'sı bozulur ve hücrenin metabolizması
faj biyosentezini başlatmaya yöneliktir
→ biyosentez: faj DNA'sı hücre içinde çoğalır
→ olgunlaşma: çoğaltılmış materyal, tamamen oluşturulmuş viral fajlar halinde toplanır
→ liziz: yeni oluşan fajlar enfekte hücreden salınır
(bu süreçte kendisi yok edilir) yeni konakçı hücreler aramak için [25]
Mikroplar, Dünya'nın ekosistemlerini sürdüren besin dönüşümlerini yönlendirir,[26] ve bu mikropları enfekte eden virüsler, hem mikrobiyal popülasyon boyutunu hem de çeşitliliği modüle eder.[27][24] siyanobakteri Proklorokok, en bol oksijenli fototrof Dünya üzerinde, küresel birincil karbon üretiminin önemli bir kısmına katkıda bulunur ve sıklıkla mililitrede 100.000 hücre yoğunluğuna ulaşır. oligotrofik ve ılıman okyanuslar.[28] Bu nedenle, viral (siyanofaj) enfeksiyonu ve parçalanması Proklorokok küreselin önemli bir bileşenini temsil eder karbon döngüsü. Siyanofajlar, konakçı ölümlerini tetiklemedeki ekolojik rollerine ek olarak, çekirdek fotosentez genleri de dahil olmak üzere genleri birlikte kullanarak ve değiştirerek konakçılarının metabolizmasını ve evrimini etkiler.[24]


Uzun zamandır, kuyruklu fajlar düzenin Caudovirales organizma sayısı ve çeşitliliği bakımından deniz ekosistemlerine hakim görünüyordu.[16] Bununla birlikte, daha yeni araştırmaların bir sonucu olarak, kuyruklu olmayan virüsler, çok sayıda derinliğe ve okyanus bölgelerine hakim gibi görünmektedir.[29] Bu kuyruklu olmayan fajlar ayrıca deniz bakterilerini de enfekte eder ve aileleri içerir. Corticoviridae,[30] Inoviridae,[31] Mikroviridae [32] ve Autolykiviridae.[33][34][35][36]
Archaeal virüsler
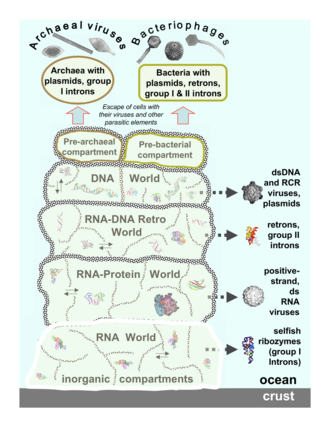
Archaean virüsleri içinde çoğalır Archaea: Bunlar, alışılmadık ve bazen benzersiz şekillere sahip çift sarmallı DNA virüsleridir.[37][38] Bu virüsler, en ayrıntılı olarak, termofilik arkeler, özellikle siparişler Sulfolobales ve Termoprotealler.[39] Bu virüslere karşı savunmalar, tekrarlayan DNA Virüslerin genleri ile ilgili olan arkay genomları içindeki diziler.[40][41] Çoğu archaea, virüslere karşı uyarlanabilir bir savunma olarak CRISPR-Cas sistemlerine sahiptir. Bunlar, archaea'nın daha sonra RNA girişimine benzer bir işlem kullanarak virüs tarafından sonraki enfeksiyonları hedeflemek ve ortadan kaldırmak için kullanılan viral DNA bölümlerini tutmasını sağlar.[42]
Mantar virüsleri
Mikovirüsler, mikofajlar olarak da bilinir, enfekte eden virüslerdir mantarlar. Mantar hücrelerinin enfeksiyonu, hayvan hücrelerinin enfeksiyonundan farklıdır. Mantarların kitinden yapılmış sert bir hücre duvarı vardır, bu nedenle çoğu virüs bu hücrelerin içine ancak hücre duvarına travma geçirdikten sonra girebilir.[43]
- Görmek; Nerva, L .; Ciuffo, M .; Vallino, M .; Margaria, P .; Varese, G.C .; Gnavi, G .; Turina, M. (2016). "Bir deniz mantarları koleksiyonundan viral ve plazmit simbiyotlarının tespiti ve karakterizasyonu için çoklu yaklaşımlar". Virüs Araştırması. 219: 22–38. doi:10.1016 / j.virusres.2015.10.028. hdl:2318/1527617. PMID 26546154.
Ökaryot virüsleri

Deniz protistleri
2015 yılına kadar, etkileyen yaklaşık 40 virüs deniz protistleri çoğu mikroalg virüsleri olmak üzere izole edilmiş ve incelenmiştir.[44] Bu deniz protist virüslerinin genomları oldukça çeşitlidir.[45][46] Deniz yosunları ailedeki virüsler bulaşabilir Phycodnaviridae. Bunlar büyüktür (100–560 kb) çift sarmallı DNA virüsleri ile ikosahedral şekilli kapsidler. 2014 yılına kadar, aile içinde altı cinse bölünmüş 33 tür tespit edildi,[47][48] olarak bilinen bir süper büyük virüs grubuna ait olan nükleositoplazmik büyük DNA virüsleri. Kanıtlar 2014 yılında yayınlandı. Phycodnaviridae daha önce inandığı gibi, sadece alg türlerinden ziyade insanları enfekte edebilir.[49] Bu aile altındaki çoğu cins, hücre reseptörüyle konakçı hücreye girer endositoz ve çekirdekte çoğalır.

Phycodnaviridae alg konaklarının büyümesini ve üretkenliğini düzenleyerek önemli ekolojik roller oynarlar. Alg türleri böyle Heterosigma akashiwo ve cins Chrysochromulina balıkçılığa zarar verebilecek yoğun çiçeklenmeler oluşturarak su ürünleri yetiştiriciliği endüstrisinde kayıplara neden olabilir.[50] Heterosigma akashiwo virüsü (HaV), bu alg türlerinin ürettiği toksik kırmızı gelgitlerin tekrarlanmasını önlemek için mikrobiyal bir ajan olarak kullanılması önerilmiştir.[51] kokolithovirüs Emiliania huxleyi virüsü 86, dev bir çift sarmallı DNA virüsü, her yerde bulaşır kokolitofor Emiliania huxleyi.[47][48] Bu virüs, deniz virüsleri arasında bilinen en büyük genomlardan birine sahiptir.[52] Phycodnaviridae tatlı su ve deniz yosunu türlerinin ölümüne ve parçalanmasına neden olur, suya organik karbon, nitrojen ve fosfor bırakarak mikrobiyal döngü için besin sağlar.[53]
Virüs-prokaryot oranı, VPR, genellikle virüsler ve konakçılar arasındaki ilişkinin bir göstergesi olarak kullanılır. Çalışmalar, virüsün deniz mikrobiyal üretkenliği, ölüm oranı ve biyojeokimyasal döngü üzerindeki etkisini dolaylı olarak çıkarmak için VPR'yi kullandı.[54] Bununla birlikte, bu yaklaşımları yaparken, bilim adamları yüzey okyanusunda gözlemlenen ortalama VPR olan 10: 1 VPR olduğunu varsayarlar.[54][17] Gerçek VPR, konuma bağlı olarak büyük ölçüde değişir, bu nedenle VPR, tedavi edildiği şekliyle viral aktivite veya bolluk için doğru vekil olmayabilir.[54][55]
Deniz omurgasızları
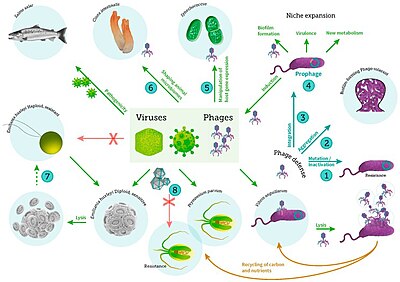
bakteri, fitoplankton ve balıkların viral enfeksiyonu dahil[56]
Deniz omurgasızları viral hastalıklara karşı hassastır.[57][58][59] Deniz yıldızı israf hastalığı bir hastalığı denizyıldızı ve düzensiz olarak ortaya çıkan ve etkilenenlerin kitlesel ölümlerine neden olan diğer birkaç ekinoderm.[60] Bu hastalıktan etkilenen yaklaşık 40 farklı deniz yıldızı türü vardır. 2014 yılında hastalığın şu anda bilinen tek sarmallı bir DNA virüsü ile ilişkili olduğu öne sürüldü. deniz yıldızı ile ilişkili densovirüs (SSaDV); ancak deniz yıldızı israf hastalığı tam olarak anlaşılamamıştır.[61]
Deniz omurgalıları
Balık özellikle enfeksiyonlara yatkındır rabdovirüsler kuduz virüsünden farklı ancak bununla ilgili. Somon, turna balığı, levrek, levrek, sazan ve morina gibi türlerde en az dokuz tür rabdovirüs ekonomik olarak önemli hastalıklara neden olur. Semptomlar anemi, kanama, uyuşukluk ve suyun sıcaklığından etkilenen ölüm oranını içerir. Kuluçkahanelerde hastalıklar genellikle sıcaklığın 15–18 ° C'ye çıkarılmasıyla kontrol edilir.[62]:442–443 Tüm omurgalılar gibi, balıklar da acı çekiyor herpes virüsleri. Bu eski virüsler, konakçılarıyla birlikte evrimleşmiştir ve oldukça türe özgüdür.[62]:324 Balıklarda kanserli tümörlere ve adı verilen kanserli olmayan büyümelere neden olurlar. hiperplazi.[62]:325
1984 yılında bulaşıcı somon anemisi (ISAv) Norveç'te bir Atlantik somonu kuluçkahane. Salgındaki balıkların yüzde sekseni öldü. Viral bir hastalık olan ISAv, şu anda Atlantik somon yetiştiriciliğinin yaşayabilirliği için büyük bir tehdit oluşturuyor.[63] Adından da anlaşılacağı gibi, şiddetli anemi enfekte balık. Memelilerden farklı olarak, balıkların kırmızı kan hücreleri DNA'ya sahiptir ve virüslerle enfekte olabilir. Yönetim stratejileri, bir aşı geliştirmeyi ve hastalığa karşı genetik direnci iyileştirmeyi içerir.[64]
Deniz memelileri aynı zamanda deniz viral enfeksiyonlarına karşı hassastır. 1988 ve 2002'de binlerce liman mühürleri Avrupa'da öldürüldü fosin distemper virüsü.[65] Dahil olmak üzere birçok başka virüs kalisivirüsler, herpes virüsleri, adenovirüsler ve parvovirüsler, deniz memelileri popülasyonlarında dolaşır.[66]
Dev deniz virüsleri
Çoğu virüs uzunluğu yaklaşık 20 ila 300 nanometre arasında değişir. Bu, yaklaşık 400 nanometreden başlayan bakteri uzunluğu ile karşılaştırılabilir. Ayrıca orada dev virüsler sık sık aranır kızlarıtipik olarak yaklaşık 1000 nanometre (bir mikron) uzunluğundadır. Tüm dev virüsler, filum Nükleocytoviricota (NCLDV) ile birlikte Poxvirüsler Bunlardan en büyüğü bilinen Tupanvirüs. Bu dev virüs cinsi, 2018 yılında derin okyanusta ve bir soda gölünde keşfedildi ve toplam uzunluğu 2,3 mikrona kadar ulaşabilir.[67]
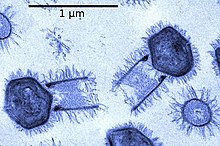
 Dev mimivirüs
Dev mimivirüs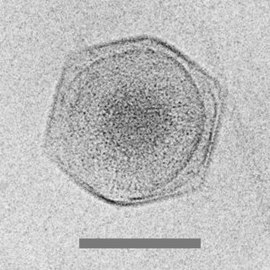
Kriyo-elektron mikrografı of CroV dev virüs [68]
ölçek çubuğu = 0.2µm
Dev virüslerin keşfi ve ardından karakterize edilmesi, evrimsel kökenleri ile ilgili bazı tartışmaları tetikledi. Kökenlerine ilişkin iki ana hipotez, ya küçük virüslerden evrimleştikleri, ev sahibi organizmalardan DNA aldıkları ya da çok karmaşık organizmalardan üreme için kendi kendine yeterli olmayan mevcut biçime evrimleştikleri şeklindedir.[69] Dev virüslerin ne tür karmaşık organizmalardan sapmış olabileceği de bir tartışma konusudur. Bir öneri, başlangıç noktasının aslında dördüncü bir noktayı temsil etmesidir. alan adı hayatın,[70][71] ancak bu büyük ölçüde azaltılmıştır.[72][73]
Virofajlar
Virofajlar küçük, çift sarmallı DNA virüsleridir. ortak enfeksiyon nın-nin dev virüsler. Virofajlar, kendi replikasyonları için birlikte enfekte eden dev virüsün viral replikasyon fabrikasına güvenirler. Virofajların özelliklerinden biri, parazit birlikte bulaşan virüsle ilişki. Çoğaltma için dev virüse bağımlılıkları genellikle dev virüslerin etkisiz hale gelmesine neden olur. Virofaj, konakçı organizmanın iyileşmesini ve hayatta kalmasını iyileştirebilir. Diğerlerinin aksine uydu virüsleri virofajların parazit onların birlikte bulaşan virüsleri üzerindeki etkisi. Virofajların dev bir virüsü etkisiz hale getirdiği ve böylece konakçı organizmanın durumunu iyileştirdiği gözlemlenmiştir.
Bilinen tüm virofajlar aile içinde gruplanır Lavidaviridae ("büyük virüse bağımlı veya ilişkili" den + -viridae ).[74] İlk virofaj, bir soğutma kulesi 2008'de Paris, Fransa'da. Birlikte bulaşan dev virüsü ile keşfedildi. Acanthamoeba castellanii mamavirüs (ACMV). Virofaj seçildi Sputnik ve replikasyonu tamamen ACMV ve sitoplazmik replikasyon mekanizmasının ko-enfeksiyonuna dayanıyordu. Sputnik'in ayrıca ACMV üzerinde inhibe edici bir etkiye sahip olduğu ve konağın hayatta kalmasını iyileştirdiği keşfedildi. Diğer karakterize edilmiş virofajlar arasında Sputnik 2, Sputnik 3, Zamilon ve Mavirüs.[75][76]
Bu virofajların çoğu analiz edilerek keşfedildi metagenomik veri kümeleri. Metagenomik analizde, DNA dizileri, belirli önemli kalıpları ve özellikleri ortaya çıkaran çoklu biyoinformatik algoritmalarla çalıştırılır. Bu veri kümelerinde dev virüsler ve virofajlar var. 17-20 civarında dizileri arayarak ayrılırlar.kbp halihazırda dizilenmiş virofajlara benzerlik gösteren uzun. Bu virofajlar, doğrusal veya dairesel çift sarmallı DNA genomlarına sahip olabilir.[77] Kültürdeki virofajlar, yaklaşık 40 ila 80 nanometre uzunluğunda olan ikosahedral kapsid partiküllerine sahiptir.[78] Virofaj parçacıkları o kadar küçüktür ki, bu parçacıkları görüntülemek için elektron mikroskobu kullanılmalıdır. Metagenomik dizi temelli analizler, yaklaşık 57 tam ve kısmi virofaj genomunu tahmin etmek için kullanılmıştır.[79] ve Aralık 2019'da, 27 farklı taksonomik sınıftan insan bağırsağı, bitki rizosfer ve karasal alt yüzey dahil olmak üzere çeşitli habitatlardan 328 yüksek kaliteli (tam veya neredeyse tamamlanmış) genom tespit etmek.[80]

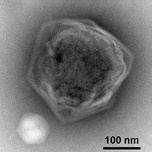
Dev bir deniz virüsü CroV enfekte eder ve ölüme neden olur liziz deniz zooflagellate Kafeterya roenbergensis.[82] Bu kıyı ekolojisini etkiler çünkü Kafeterya roenbergensis suda bulunan bakterilerle beslenir. Düşük sayılar olduğunda Kafeterya roenbergensis yaygın CroV enfeksiyonları nedeniyle, bakteri popülasyonları katlanarak artar.[83] Etkisi CroV doğal popülasyonları hakkında C. roenbergensis bilinmeyen kalır; bununla birlikte, virüsün çok konağa özgü olduğu ve diğer yakın ilişkili organizmaları enfekte etmediği bulunmuştur.[84] Kafeterya roenbergensis de ikinci bir virüsle enfekte olmuştur. Mavirüs virofajı CroV ile ko-enfeksiyon sırasında.[75] Bu virüs, CroV replikasyonuna müdahale eder ve bu da C. roenbergensis hücreler. Mavirüs hücrelerin genomuna entegre olabilir C. roenbergensis ve böylece nüfusa bağışıklık kazandırır.[76]
Deniz virüslerinin rolü
Deniz virüsleri son zamanlarda kapsamlı bir şekilde incelenmesine rağmen, birçok ekosistem işlevi ve döngüsünde kritik roller üstlendikleri zaten bilinmektedir. Deniz virüsleri bir dizi önemli ekosistem servisleri ve deniz ekosistemlerinin düzenlenmesi için gereklidir.[3] Deniz bakteriyofajları ve diğer virüsler, biyojeokimyasal döngüler küresel olarak sağlayın ve düzenleyin mikrobiyal biyolojik çeşitlilik, döngüsü karbon deniz yoluyla besin ağları ve bakteri oluşumunu önlemek için gereklidir. nüfus patlamaları.[85]
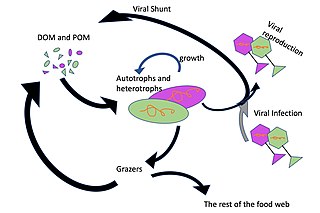
Viral şant

Okyanustaki virüsler için baskın konaklar, bakteriler gibi deniz mikroorganizmalarıdır.[13] Bakteriyofajlar bitkiler ve hayvanlar için zararsızdır ve deniz ve tatlı su ekosistemlerinin düzenlenmesi için gereklidir.[87] önemli ölüm ajanlarıdır fitoplankton temeli besin zinciri su ortamlarında.[88] Sudaki mikrobiyal topluluklardaki bakterileri enfekte eder ve yok ederler ve en önemli mekanizmalardan biridir. karbon geri dönüşümü ve deniz ortamlarında besin döngüsü. Ölü bakteri hücrelerinden salınan organik moleküller, taze bakteri ve yosun büyümesini uyarır. viral şant.[89]
Böylelikle deniz virüslerinin besin döngülerinde verimini artırarak önemli bir rol oynadığı düşünülmektedir. biyolojik pompa. Virüsler neden liziz canlı hücreler, yani hücre zarlarını parçalarlar. Bu, aşağıdaki gibi bileşikleri serbest bırakır amino asitler ve nükleik asitler, yüzeye yakın yerlerde geri dönüştürülme eğilimindedir.
Viral aktivite ayrıca biyolojik pompanın yeteneğini de arttırır. ayırıcı karbon derin okyanusta.[66] Lizis, hücre duvarlarında bulunan ve muhtemelen daha derin sulara ihraç edilen, karbon bakımından zengin olan daha fazla sindirilemeyen malzeme salar. Bu nedenle, viral şant tarafından daha derin sulara ihraç edilen materyal muhtemelen türetildiği materyalden daha fazla karbon zengindir.[90][91] Okyanuslardaki solunum miktarını artırarak, virüsler dolaylı olarak atmosferdeki karbondioksit miktarını yaklaşık üç kat azaltmaktan sorumludur. gigatonnes yıllık karbon miktarı.[66] Bakterilerin virüsler tarafından parçalanmasının da nitrojen döngüsünü artırdığı ve fitoplankton büyümesini uyardığı gösterilmiştir.[92]
viral şant yol, engelleyen bir mekanizmadır (prokaryotik ve ökaryotik ) deniz mikrobik partikül organik madde (POM) yukarı geçişten trofik seviyeler onları geri dönüştürerek çözünmüş organik madde (DOM), mikroorganizmalar tarafından kolaylıkla alınabilir. Viral şant, tek bir deniz mikrop türünün mikro çevreye hakim olmasını engelleyerek mikrobiyal ekosistem içinde çeşitliliğin korunmasına yardımcı olur.[93] Viral şant yolu tarafından geri dönüştürülen DOM, diğer ana deniz DOM kaynakları tarafından üretilen miktarla karşılaştırılabilir.[94]

Virüsler, deniz ortamlarında en bol bulunan biyolojik varlıktır.[5] Ortalama olarak, tipik bir çay kaşığı deniz suyunda yaklaşık on milyon tane bulunur.[96] Bu virüslerin çoğu bakteriyofajlar heterotrofik bakterileri enfekte etmek ve siyanofajlar siyanobakterileri enfekte ediyor. Virüsler, mikroplara kıyasla görece bollukları nedeniyle mikrobiyal döngüdeki mikroorganizmaları kolayca enfekte eder.[97] Prokaryotik ve ökaryotik ölüm oranı, karbon besin geri dönüşümüne katkıda bulunur. hücre parçalanması. Nitrojen (özellikle amonyum) rejenerasyonunun da kanıtları vardır. Bu besin geri dönüşümü, mikrobiyal büyümeyi uyarmaya yardımcı olur.[98] Küresel okyanuslarda fitoplanktondan birincil üretimin% 25 kadarı, mikrobiyal döngü içinde viral şantlama yoluyla geri dönüştürülebilir.[99]
Alg çoğalmalarını sınırlamak
Mikroorganizmalar, deniz biyokütlesinin yaklaşık% 70'ini oluşturur.[100] Virüslerin her gün bu biyokütlenin% 20'sini öldürdüğü ve okyanuslarda bakteri ve arkelerin olduğundan 15 kat daha fazla virüs olduğu tahmin edilmektedir. Virüsler, zararlıların hızla yok edilmesinden sorumlu ana ajanlardır. alg çiçekleri,[101] bu genellikle diğer deniz yaşamını öldürür.[102] Bilim adamları denizin potansiyelini araştırıyor siyanofajlar önlemek veya tersine çevirmek için kullanılacak ötrofikasyon. Okyanuslardaki virüslerin sayısı, daha az sayıda konakçı organizmanın bulunduğu, açık denizde ve suyun derinliklerinde azalır.[103]
Gen transferi
Deniz bakteriyofajları genellikle şunları içerir: yardımcı metabolik genler, viral enfeksiyon sırasında konakçı metabolizmasını destekleyerek viral replikasyonu sürdürdüğü düşünülen konakçıdan türetilen genler.[104] Bu genler, karbon, fosfor, kükürt ve nitrojen dahil olmak üzere birçok biyojeokimyasal döngüyü etkileyebilir.[105][106][107][108]
Virüsler önemli bir doğal araçtır genlerin aktarılması artan farklı türler arasında genetik çeşitlilik ve evrimi yönlendirir.[9] Virüslerin, bakteriler, arkeler ve ökaryotlar çeşitlenmeden önce, erken evrimde merkezi bir rol oynadığı düşünülmektedir. son evrensel ortak ata Dünyadaki yaşamın.[109] Virüsler, hala Dünya üzerindeki keşfedilmemiş genetik çeşitliliğin en büyük rezervuarlarından biridir.[103]
Deniz habitatları
Kıyı boyunca
Deniz kıyı habitatları, kara ve okyanus arasındaki arayüzde bulunur. Bu olasıdır RNA virüsleri bu ortamlarda önemli roller oynar.[110]
Okyanus yüzeyinde

Deniz yüzeyi habitatları, atmosfer ile okyanus arasındaki arayüzde bulunur. biyofilm benzeri Okyanus yüzeyindeki habitat, yüzeyde yaşayan mikroorganizmaları barındırır. Neuston. Mikro tabakadaki virüsler, sözde Virioneuston, son zamanlarda potansiyel olarak önemli ekolojik etkilere sahip sınır yüzey katmanlarında gizemli biyolojik varlıklar olarak araştırmacıların ilgisini çekmiştir. Bu geniş hava-su arayüzü, küresel yüzey alanının% 70'inden fazlasını kapsayan büyük hava-su değişim süreçlerinin kesişme noktasında yer aldığından, bunun büyük olasılıkla, deniz biyojeokimyasal döngüleri, üzerinde mikrobiyal döngü ve gaz değişimi ve ayrıca deniz besin ağı yapısı, deniz yüzeyi mikro tabakasından kaynaklanan hava kaynaklı virüslerin küresel dağılımı ve insan sağlığı.[111]
Su sütununda
Deniz viral aktivitesi, potansiyel bir açıklamayı sunar. plankton paradoksu öneren George Hutchinson 1961'de.[112] Planktonun paradoksu, birçok plankton türünün, okyanusun sınırlı kaynakların yaratması gereken küçük bölgelerde tanımlanmış olmasıdır. rekabetçi dışlama, bir arada bulunan türlerin sayısını sınırlandırır.[112] Konakçılarla potansiyel temas arttıkça viral enfeksiyon arttıkça deniz virüsleri bu etkide rol oynayabilir.[4] Virüsler bu nedenle çok fazla büyüyen plankton türlerinin popülasyonlarını kontrol edebilir ve çok çeşitli türlerin bir arada yaşamasına izin verebilir.[4]
Çökeltilerde
Deniz bakteriyofajları önemli bir rol oynar. derin deniz ekosistemler. 5x10 arası var12 ve 1x1013 derin deniz çökeltilerinde metrekare başına fajlar ve bunların bolluğu, sayısı ile yakından ilişkilidir. prokaryotlar çökeltilerde bulundu. Çökeltilerde bulunan prokaryotların% 80'inin ölümünden sorumludurlar ve bu ölümlerin neredeyse tamamı hücre kaynaklı liziz (patlama). Bu, canlı hücrelerden gelen nitrojen, karbon ve fosforun çözünmüş organik maddeye ve döküntüye dönüştürülmesini sağlar ve derin deniz tortularında yüksek oranda besin dönüşümüne katkıda bulunur. Biyojeokimyasal döngülerde derin deniz sedimanlarının önemi nedeniyle, deniz bakteriyofajları karbon, azot ve fosfor döngüleri. Bu etkileri daha kesin olarak aydınlatmak için daha fazla araştırma yapılması gerekiyor.[113]
Hidrotermal menfezlerde
Virüsler bir parçası hidrotermal havalandırma mikrobiyal topluluğu ve bu ekosistemlerdeki mikrobiyal ekoloji üzerindeki etkileri, gelişmekte olan bir araştırma alanıdır.[114] Virüsler, okyanustaki en bol yaşamdır ve en büyük genetik çeşitlilik rezervuarını barındırır.[101] Enfeksiyonları genellikle ölümcül olduğundan, önemli bir ölüm kaynağı oluştururlar ve bu nedenle biyolojik oşinografik süreçler üzerinde yaygın bir etkiye sahiptirler. evrim ve biyojeokimyasal döngü okyanusun içinde.[103] Bununla birlikte, havalandırma habitatlarında bulunan virüslerin daha fazla benimsediğini gösteren kanıtlar bulunmuştur. karşılıklı -den parazit içinde bulundukları aşırı ve değişken çevrede hayatta kalabilmek için evrimsel strateji.[115] Derin deniz hidrotermal menfezlerinin yüksek viral üretimi gösteren çok sayıda virüse sahip olduğu bulundu.[116] Diğer deniz ortamlarında olduğu gibi, derin deniz hidrotermal virüsleri bolluğunu ve çeşitliliğini etkiler prokaryotlar ve bu nedenle mikrobiyal biyojeokimyasal döngüyü şu şekilde etkiler: Lysing ev sahiplerinin çoğalmasını sağlar.[117] Bununla birlikte, bir ölüm ve popülasyon kontrolü kaynağı olarak rollerinin aksine, virüslerin, aşırı ortamlarda prokaryotların hayatta kalmasını arttırdığı ve genetik bilgi rezervuarı görevi gördüğü varsayılmıştır. Virosferin, çevresel stresler altında mikroorganizmalarla etkileşimlerinin, bu nedenle konakçı genlerin yayılması yoluyla mikroorganizmanın hayatta kalmasına yardımcı olduğu düşünülmektedir. yatay gen transferi.[118]
Kutup bölgeleri
Dağıtım
Virüsler büyük ölçüde ana bilgisayara özeldir.[119] Bir deniz virüsünün, virüsün yaşadığı aynı bölgede yaşayanlar gibi, aynı anda meydana gelen organizmaları etkileme olasılığı daha yüksektir.[120] Bu nedenle, biyocoğrafya bir virionun enfekte etme kabiliyetinde önemli bir faktördür.
Spatiotemporal ve diğer çevresel gradyanlar boyunca viral popülasyonlardaki bu varyasyon bilgisi, transmisyon elektron mikroskobu (TEM) ile belirlendiği üzere viral morfoloji ile desteklenir. Kuyruklu olmayan virüsler, birden çok derinlikte ve okyanus bölgelerinde baskın gibi görünmektedir ve bunu Caudovirales izlemektedir. miyovirüsler, podovirüsler ve sifovirüsler.[29] Ancak ailelere ait virüsler Corticoviridae,[121] Inoviridae[122] ve Mikroviridae[123] ayrıca çeşitli deniz bakterilerini enfekte ettiği bilinmektedir. Metagenomik kanıtlar, mikrovirüslerin (ikosahedral ssDNA fajları) özellikle deniz habitatlarında yaygın olduğunu göstermektedir.[123]
Viral çeşitliliği değerlendirmeye yönelik metagenomik yaklaşımlar, genellikle referans sekans eksikliğinden dolayı sınırlıdır ve birçok sekansı açıklamasız bırakır.[124] Bununla birlikte, viral içerikler, tipik olarak bir deniz suyu numunesinin 0.02 um filtrasyonundan sonra üretilen bir viral fraksiyonun doğrudan sekanslanması yoluyla veya bir mikrobiyal metagenomdan viral kontigleri veya viral genomları tanımlamak için biyoinformatik yaklaşımlar yoluyla üretilir. VirSorter gibi varsayılan viral bölgeleri tanımlamak için yeni araçlar[125] ve VirFinder,[126] Viral bolluk modellerinin, konakçı aralığının ve deniz bakteriyofajının fonksiyonel içeriğinin değerlendirilmesine olanak sağlar.[127][128]
Ayrıca bakınız
Referanslar
- ^ Bonnain, C .; Breitbart, M .; Buck, K.N. (2016). "Ferrojan at hipotezi: okyanustaki demir-virüs etkileşimleri". Deniz Bilimlerinde Sınırlar. 3: 82. doi:10.3389 / fmars.2016.00082. S2CID 2917222.
- ^ Bu Küçük Organizmaların Bazı Gerçekten Garip Şekilleri Var National Geogaphic ", 12 Kasım 2016.
- ^ a b Shors T (2008). Virüsleri Anlamak. Jones ve Bartlett Yayıncılar. s. 5. ISBN 978-0-7637-2932-5.
- ^ a b c d Brussaard CP, Baudoux A, Rodríguez-Valera F (2016). Stal LJ, Cretoiu MS (editörler). Deniz Virüsleri. Deniz Mikrobiyomu. Springer Uluslararası Yayıncılık. s. 155–183. doi:10.1007/978-3-319-33000-6_5. ISBN 9783319329987.
- ^ a b c d Koonin EV, Senkevich TG, Dolja VV (2006). "Antik Virüs Dünyası ve hücrelerin evrimi". Biyoloji Doğrudan. 1: 29. doi:10.1186/1745-6150-1-29. PMC 1594570. PMID 16984643.CS1 Maint: birden çok isim: yazarlar listesi (bağlantı)
 Materyal, bir altında bulunan bu kaynaktan kopyalandı. Creative Commons Attribution 2.0 Uluslararası Lisansı.
Materyal, bir altında bulunan bu kaynaktan kopyalandı. Creative Commons Attribution 2.0 Uluslararası Lisansı. - ^ Mahy WJ ve Van Regenmortel MHV (editörler). Masa Genel Viroloji Ansiklopedisi. Oxford: Academic Press; 2009. ISBN 0-12-375146-2. s. 28.
- ^ Iyer LM, Balaji S, Koonin EV, Aravind L (2006). "Nükleo-sitoplazmik büyük DNA virüslerinin evrimsel genomiği". Virüs Araştırması. 117 (1): 156–84. doi:10.1016 / j.virusres.2006.01.009. PMID 16494962.CS1 Maint: birden çok isim: yazarlar listesi (bağlantı)
- ^ Sanjuán R, Nebot MR, Chirico N, Mansky LM, Belshaw R (Ekim 2010). "Viral mutasyon oranları". Journal of Virology. 84 (19): 9733–48. doi:10.1128 / JVI.00694-10. PMC 2937809. PMID 20660197.CS1 Maint: birden çok isim: yazarlar listesi (bağlantı)
- ^ a b Canchaya C, Fournous G, Chibani-Chennoufi S, Dillmann ML, Brüssow H (2003). "Yanal gen transferinin ajanları olarak faj". Mikrobiyolojide Güncel Görüş. 6 (4): 417–24. doi:10.1016 / S1369-5274 (03) 00086-9. PMID 12941415.CS1 Maint: birden çok isim: yazarlar listesi (bağlantı)
- ^ Koonin EV, Starokadomskyy P (2016). "Virüsler canlı mı? Çoğalıcı paradigması, eski ama yanlış yönlendirilmiş bir soruya kesin ışık tutuyor". Biyolojik ve Biyomedikal Bilimler Tarihi ve Felsefesi Çalışmaları. 59: 125–134. doi:10.1016 / j.shpsc.2016.02.016. PMC 5406846. PMID 26965225.
- ^ Koonin, E. V .; Starokadomskyy, P. (7 Mart 2016). "Virüsler canlı mı? Çoğalıcı paradigması, eski ama yanlış yönlendirilmiş bir soruya kesin ışık tutuyor". Biyolojik ve Biyomedikal Bilimler Tarihi ve Felsefesi Çalışmaları. 59: 125–34. doi:10.1016 / j.shpsc.2016.02.016. PMC 5406846. PMID 26965225.
- ^ Rybicki, EP (1990). "Yaşamın sınırındaki organizmaların sınıflandırılması veya virüs sistematiği ile ilgili sorunlar". Güney Afrika Bilim Dergisi. 86: 182–186.
- ^ a b Mann NH (Mayıs 2005). "Fajın üçüncü çağı". PLOS Biyoloji. 3 (5): e182. doi:10.1371 / journal.pbio.0030182. PMC 1110918. PMID 15884981.
- ^ Wommack KE, Hill RT, Muller TA, Colwell RR (Nisan 1996). "Güneş ışığının bakteriyofaj canlılığı ve yapısı üzerindeki etkileri". Uygulamalı ve Çevresel Mikrobiyoloji. 62 (4): 1336–41. doi:10.1128 / AEM.62.4.1336-1341.1996. PMC 167899. PMID 8919794.
- ^ Marie D, Brussaard CP, Thyrhaug R, Bratbak G, Vaulot D (Ocak 1999). "Akış sitometrisi ile kültürde ve doğal örneklerde deniz virüslerinin sayımı". Uygulamalı ve Çevresel Mikrobiyoloji. 65 (1): 45–52. doi:10.1128 / AEM.65.1.45-52.1999. PMC 90981. PMID 9872758.
- ^ a b Mann, NH (2005-05-17). "Fajın Üçüncü Çağı". PLOS Biyoloji. 3 (5): 753–755. doi:10.1371 / journal.pbio.0030182. PMC 1110918. PMID 15884981.
- ^ a b Wommack KE, Colwell RR (Mart 2000). "Virioplankton: su ekosistemlerindeki virüsler". Mikrobiyoloji ve Moleküler Biyoloji İncelemeleri. 64 (1): 69–114. doi:10.1128 / MMBR.64.1.69-114.2000. PMC 98987. PMID 10704475.
- ^ Bergh O, Børsheim KY, Bratbak G, Heldal M (Ağustos 1989). "High abundance of viruses found in aquatic environments". Doğa. 340 (6233): 467–68. Bibcode:1989Natur.340..467B. doi:10.1038/340467a0. PMID 2755508. S2CID 4271861.
- ^ Shors pp. 595–97
- ^ Bickle TA, Krüger DH (June 1993). "Biology of DNA restriction". Mikrobiyolojik İncelemeler. 57 (2): 434–50. doi:10.1128/MMBR.57.2.434-450.1993. PMC 372918. PMID 8336674.
- ^ Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, et al. (Mart 2007). "CRISPR provides acquired resistance against viruses in prokaryotes". Bilim. 315 (5819): 1709–12. Bibcode:2007Sci...315.1709B. doi:10.1126/science.1138140. hdl:20.500.11794/38902. PMID 17379808. S2CID 3888761.
- ^ Brouns SJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJ, Snijders AP, et al. (Ağustos 2008). "Small CRISPR RNAs guide antiviral defense in prokaryotes". Bilim. 321 (5891): 960–64. Bibcode:2008Sci...321..960B. doi:10.1126/science.1159689. PMC 5898235. PMID 18703739.
- ^ Mojica FJ, Rodriguez-Valera F (September 2016). "The discovery of CRISPR in archaea and bacteria". FEBS Dergisi. 283 (17): 3162–69. doi:10.1111/febs.13766. hdl:10045/57676. PMID 27234458.
- ^ a b c d Murata, K.; Zhang, Q .; Galaz-Montoya, J.G.; Fu, C.; Coleman, M.L.; Osburne, M.S.; Schmid, M.F.; Sullivan, M.B.; Chisholm, S.W.; Chiu, W. (2017). "Visualizing adsorption of cyanophage P-SSP7 onto marine Proklorokok". Bilimsel Raporlar. 7: 44176. Bibcode:2017NatSR...744176M. doi:10.1038/srep44176. PMC 5345008. PMID 28281671.
- ^ How do bacteriophages reproduce? Barselona Üniversitesi. Erişim tarihi: 12 Temmuz 2020.
- ^ Falkowski, P.G .; Fenchel, T.; Delong, E.F. (2008). "The microbial engines that drive Earth's biogeochemical cycles". Bilim. 320 (5879): 1034–1039. Bibcode:2008Sci...320.1034F. doi:10.1126/science.1153213. PMID 18497287. S2CID 2844984.
- ^ Brum, J.R.; Sullivan, M.B. (2015). "Rising to the challenge: accelerated pace of discovery transforms marine virology". Doğa İncelemeleri Mikrobiyoloji. 13 (3): 147–159. doi:10.1038/nrmicro3404. PMID 25639680. S2CID 32998525.
- ^ Bouman, H.A.; Ulloa, O.; Scanlan, D.J.; Zwirglmaier, K.; Li, W.K.; Platt, T.; Stuart, V.; Barlow, R.; Leth, O.; Clementson, L.; Lutz, V. (2006). "Oceanographic basis of the global surface distribution of Proklorokok ecotypes". Bilim. 312 (5775): 918–921. Bibcode:2006Sci...312..918B. doi:10.1126/science.1122692. PMID 16690867. S2CID 20738145.
- ^ a b Brum JR, Schenck RO, Sullivan MB (September 2013). "Global morphological analysis of marine viruses shows minimal regional variation and dominance of non-tailed viruses". ISME Dergisi. 7 (9): 1738–51. doi:10.1038/ismej.2013.67. PMC 3749506. PMID 23635867.
- ^ Krupovic M, Bamford DH (2007). "Putative prophages related to lytic tailless marine dsDNA phage PM2 are widespread in the genomes of aquatic bacteria". BMC Genomics. 8: 236. doi:10.1186/1471-2164-8-236. PMC 1950889. PMID 17634101.
- ^ Xue H, Xu Y, Boucher Y, Polz MF (2012). "High Frequency of a Novel Filamentous Phage, VCYϕ, within an Environmental Vibrio cholerae Population". Uygulamalı ve Çevresel Mikrobiyoloji. 78 (1): 28–33. doi:10.1128/AEM.06297-11. PMC 3255608. PMID 22020507.
- ^ Roux S, Krupovic M, Poulet A, Debroas D, Enault F (2012). "Virom okumalarından toplanan 81 yeni tam genomdan oluşan bir koleksiyon yoluyla Microviridae viral ailesinin evrimi ve çeşitliliği". PLOS ONE. 7 (7): e40418. Bibcode:2012PLoSO...740418R. doi:10.1371 / journal.pone.0040418. PMC 3394797. PMID 22808158.
- ^ Kauffman, Kathryn M.; Hussain, Fatima A.; Yang, Joy; Arevalo, Philip; Brown, Julia M.; Chang, William K.; Vaninsberghe, David; Elsherbini, Joseph; Sharma, Radhey S.; Cutler, Michael B.; Kelly, Libusha; Polz, Martin F. (2018). "A major lineage of non-tailed dsDNA viruses as unrecognized killers of marine bacteria". Doğa. 554 (7690): 118–122. Bibcode:2018Natur.554..118K. doi:10.1038/nature25474. PMID 29364876. S2CID 4462007.
- ^ Scientists Find New Type of Virus in World’s Oceans: Autolykiviridae, on: sci-news, 25 January 2018
- ^ Never-Before-Seen Viruses With Weird DNA Were Just Discovered in The Ocean, on: scienceuyarmak, 25 January 2018
- ^ NCBI: Autolykiviridae (family) – unclassified dsDNA viruses
- ^ Lawrence CM, Menon S, Eilers BJ, Bothner B, Khayat R, Douglas T, Young MJ (2009). "Arkael virüslerin yapısal ve işlevsel çalışmaları". Biyolojik Kimya Dergisi. 284 (19): 12599–603. doi:10.1074 / jbc.R800078200. PMC 2675988. PMID 19158076.
- ^ Prangishvili D, Forterre P, Garrett RA (2006). "Viruses of the Archaea: a unifying view". Doğa İncelemeleri Mikrobiyoloji. 4 (11): 837–48. doi:10.1038/nrmicro1527. PMID 17041631. S2CID 9915859.CS1 Maint: birden çok isim: yazarlar listesi (bağlantı)
- ^ Prangishvili D, Garrett RA (2004). "Exceptionally diverse morphotypes and genomes of crenarchaeal hyperthermophilic viruses". Biyokimya Topluluğu İşlemleri. 32 (Pt 2): 204–8. doi:10.1042/BST0320204. PMID 15046572.
- ^ Mojica FJ, Díez-Villaseñor C, García-Martínez J, Soria E (February 2005). "Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements". Moleküler Evrim Dergisi. 60 (2): 174–82. Bibcode:2005JMolE..60..174M. doi:10.1007/s00239-004-0046-3. PMID 15791728. S2CID 27481111.
- ^ Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV (March 2006). "A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action". Biyoloji Doğrudan. 1: 7. doi:10.1186/1745-6150-1-7. PMC 1462988. PMID 16545108.
- ^ van der Oost J, Westra ER, Jackson RN, Wiedenheft B (July 2014). "Unravelling the structural and mechanistic basis of CRISPR-Cas systems". Doğa İncelemeleri Mikrobiyoloji. 12 (7): 479–92. doi:10.1038/nrmicro3279. PMC 4225775. PMID 24909109.
- ^ Dimmock NJ, Easton AJ, Leppard K (2007). Introduction to Modern Virology (Altıncı baskı). Blackwell Publishing. s.70. ISBN 978-1-4051-3645-7.
- ^ Tomaru Y, Kimura K and Nagasaki K (2015) "Marine Protist Viruses". In: Ohtsuka S, Suzaki T, Horiguchi T, Suzuki N, Not F (eds) Marine Protists pages 501–517. Springer, Tokyo. doi:10.1007/978-4-431-55130-0_20. ISBN 978-4-431-55130-0.
- ^ Hyman, Paul; Abedon, Stephen T. (2012). "Smaller Fleas: Viruses of Microorganisms". Scientifica. 2012: 1–23. doi:10.6064/2012/734023. PMC 3820453. PMID 24278736..
 Materyal, bir altında bulunan bu kaynaktan kopyalandı. Creative Commons Attribution 3.0 International License.
Materyal, bir altında bulunan bu kaynaktan kopyalandı. Creative Commons Attribution 3.0 International License. - ^ Short, S.M. (2012). "The ecology of viruses that infect eukaryotic algae". Çevresel Mikrobiyoloji. 14 (9): 2253–2271. doi:10.1111/j.1462-2920.2012.02706.x. PMID 22360532.
- ^ a b "Viral Bölge". ExPASy. Alındı 15 Haziran 2015.
- ^ a b ICTV. "Virüs Taksonomisi: 2014 Sürümü". Alındı 15 Haziran 2015.
- ^ Yolken, RH; et al. (2014). "Chlorovirus ATCV-1 is part of the human oropharyngeal virome and is associated with changes in cognitive functions in humans and mice". Proc Natl Acad Sci U S A. 111 (45): 16106–16111. Bibcode:2014PNAS..11116106Y. doi:10.1073/pnas.1418895111. PMC 4234575. PMID 25349393.
- ^ Tomaru, Yuji; Shirai, Yoko; Nagasaki, Keizo (2008-08-01). "Ecology, physiology and genetics of a phycodnavirus infecting the noxious bloom-forming raphidophyte Heterosigma akashiwo". Balıkçılık Bilimi. 74 (4): 701–711. doi:10.1111/j.1444-2906.2008.01580.x. S2CID 23152411.
- ^ Nagasaki, Keizo; Tarutani, Kenji; Yamaguchi, Mineo (1999-03-01). "Growth Characteristics of Heterosigma akashiwo Virus and Its Possible Use as a Microbiological Agent for Red Tide Control". Uygulamalı ve Çevresel Mikrobiyoloji. 65 (3): 898–902. doi:10.1128/AEM.65.3.898-902.1999. PMC 91120. PMID 10049839.
- ^ Bilinen en büyük viral genomlar Giantviruses.org. Erişim: 11 Haziran 2020.
- ^ Sigee, David (2005-09-27). Freshwater Microbiology: Biodiversity and Dynamic Interactions of Microorganisms in the Aquatic Environment. John Wiley & Sons. ISBN 9780470026472.
- ^ a b c Wigington CH, Sonderegger DL, Brussaard CP, Buchan A, Finke JF, Fuhrman J, Lennon JT, Middelboe M, Suttle CA, Stock C, Wilson WH (2015-08-26). "Re-examining the relationship between virus and microbial cell abundances in the global oceans". bioRxiv: 025544. bioRxiv 10.1101/025544. doi:10.1101/025544.
- ^ Parikka KJ, Le Romancer M, Wauters N, Jacquet S (May 2017). "Deciphering the virus-to-prokaryote ratio (VPR): insights into virus-host relationships in a variety of ecosystems". Cambridge Philosophical Society'nin Biyolojik İncelemeleri. 92 (2): 1081–1100. doi:10.1111/brv.12271. PMID 27113012. S2CID 3463306.
- ^ Middelboe, M.; Brussaard, C. (2017). "Marine viruses: key players in marine ecosystems". Virüsler. 9 (10): 302. doi:10.3390/v9100302. PMC 5691653. PMID 29057790.
- ^ TJohnson, P.T. (1984). "Viral diseases of marine invertebrates". Helgoländer Meeresuntersuchungen. 37 (1–4): 65–98. Bibcode:1984HM.....37...65J. doi:10.1007/BF01989296. S2CID 30161955.
- ^ Renault T (2011) "Viruses infecting marine molluscs" In: Hurst CJ (Ed) Studies in Viral Ecology, Volume 2: Animal Host Systems, John Wiley & Sons. ISBN 9781118024584.
- ^ Arzul, I.; Corbeil, S.; Morga, B.; Renault, T. (2017). "Viruses infecting marine molluscs" (PDF). Omurgasız Patoloji Dergisi. 147: 118–135. doi:10.1016/j.jip.2017.01.009. PMID 28189502.
- ^ Dawsoni, Solaster. "Sea Star Species Affected by Wasting Syndrome." Pacificrockyintertidal.org Seastarwasting.org (n.d.): n. pag. Ecology and Evolutionary Biology. Ağ.
- ^ "Sea Star Wasting Syndrome | MARINe". eeb.ucsc.edu. Alındı 2018-06-03.
- ^ a b c Murphy, FA; Gibbs, EPJ; Horzinek, MC; Studdart MJ (1999). Veterinary Virology. Boston: Akademik Basın. ISBN 978-0-12-511340-3.
- ^ New Brunswick to help Chile beat disease Fish Information and Services
- ^ Fact Sheet - Atlantic Salmon Aquaculture Research Arşivlendi 29 Aralık 2010, Wayback Makinesi Balıkçılık ve Okyanuslar Kanada. Erişim tarihi: 12 Mayıs 2009.
- ^ Hall AJ, Jepson PD, Goodman SJ, Harkonen T (2006). "Phocine distemper virus in the North and European Seas – data and models, nature and nurture". Biyolojik Koruma. 131 (2): 221–29. doi:10.1016/j.biocon.2006.04.008.
- ^ a b c Suttle CA (October 2007). "Marine viruses – major players in the global ecosystem". Doğa İncelemeleri Mikrobiyoloji. 5 (10): 801–12. doi:10.1038/nrmicro1750. PMID 17853907. S2CID 4658457.
- ^ Abrahão, Jônatas; Silva, Lorena; Silva, Ludmila Santos; Khalil, Jacques Yaacoub Bou; Rodrigues, Rodrigo; Arantes, Thalita; Assis, Felipe; Boratto, Paulo; Andrade, Miguel; Kroon, Erna Geessien; Ribeiro, Bergmann; Bergier, Ivan; Seligmann, Herve; Ghigo, Eric; Colson, Philippe; Levasseur, Anthony; Kroemer, Guido; Raoult, Didier; Scola, Bernard La (27 February 2018). "Tailed giant Tupanvirus possesses the most complete translational apparatus of the known virosphere". Doğa İletişimi. 9 (1): 749. Bibcode:2018NatCo...9..749A. doi:10.1038/s41467-018-03168-1. PMC 5829246. PMID 29487281.
- ^ Xiao, C.; Fischer, M.G.; Bolotaulo, D.M.; Ulloa-Rondeau, N.; Avila, G.A.; Suttle, C.A. (2017). "Cryo-EM reconstruction of the Cafeteria roenbergensis virus capsid suggests novel assembly pathway for giant viruses". Bilimsel Raporlar. 7 (1): 5484. Bibcode:2017NatSR...7.5484X. doi:10.1038/s41598-017-05824-w. PMC 5511168. PMID 28710447.
- ^ Bichell RE. "In Giant Virus Genes, Hints About Their Mysterious Origin". Her şey düşünüldü.
- ^ Van Etten JL (July–August 2011). "Giant Viruses". Amerikalı bilim adamı. 99 (4): 304–311. doi:10.1511/2011.91.304.
- ^ Legendre M, Arslan D, Abergel C, Claverie JM (January 2012). "Genomics of Megavirus and the elusive fourth domain of Life". İletişimsel ve Bütünleştirici Biyoloji. 5 (1): 102–6. doi:10.4161/cib.18624. PMC 3291303. PMID 22482024.
- ^ Schulz F, Yutin N, Ivanova NN, Ortega DR, Lee TK, Vierheilig J, Daims H, Horn M, Wagner M, Jensen GJ, Kyrpides NC, Koonin EV, Woyke T (April 2017). "Giant viruses with an expanded complement of translation system components" (PDF). Bilim. 356 (6333): 82–85. Bibcode:2017Sci...356...82S. doi:10.1126/science.aal4657. PMID 28386012. S2CID 206655792.
- ^ Bäckström D, Yutin N, Jørgensen SL, Dharamshi J, Homa F, Zaremba-Niedwiedzka K, Spang A, Wolf YI, Koonin EV, Ettema TJ (March 2019). "Virus Genomes from Deep Sea Sediments Expand the Ocean Megavirome and Support Independent Origins of Viral Gigantism". mBio. 10 (2): e02497-02418. doi:10.1128/mBio.02497-18. PMC 6401483. PMID 30837339.
- ^ Duponchel, S; Fischer, MG (March 2019). "Viva lavidaviruses! Five features of virophages that parasitize giant DNA viruses". PLOS Patojenleri. 15 (3): e1007592. doi:10.1371/journal.ppat.1007592. PMC 6428243. PMID 30897185.
- ^ a b Fischer MG, Suttle CA (April 2011). "A virophage at the origin of large DNA transposons". Bilim. 332 (6026): 231–4. Bibcode:2011Sci...332..231F. doi:10.1126/science.1199412. PMID 21385722. S2CID 206530677.
- ^ a b Fischer MG, Hackl (December 2016). "Host genome integration and giant virus-induced reactivation of the virophage mavirus". Doğa. 540 (7632): 288–91. Bibcode:2016Natur.540..288F. doi:10.1038/nature20593. PMID 27929021. S2CID 4458402.
- ^ Katzourakis, Aris; Aswad, Amr (2014). "The origins of giant viruses, virophages and their relatives in host genomes". BMC Biyoloji. 12: 2–3. doi:10.1186/s12915-014-0051-y. PMC 4096385. PMID 25184667.
- ^ Krupovic, Mart; Kuhn, Jens; Fischer, Metthias (Fall 2015). "A classification system for virophages and satellite viruses" (PDF). Viroloji Arşivleri. 161 (1): 233–247. doi:10.1007/s00705-015-2622-9. PMID 26446887. S2CID 14196910 - Springer aracılığıyla.
- ^ Roux, Simon; Chan, Leong-Keat; Egan, Rob; Malmstrom, Rex R.; McMahon, Katherine D.; Sullivan, Matthew B. (2017). "Ecogenomics of virophages and their giant virus hosts assessed through time series metagenomics". Doğa İletişimi. 8 (1): 858. Bibcode:2017NatCo...8..858R. doi:10.1038/s41467-017-01086-2. ISSN 2041-1723. PMC 5636890. PMID 29021524.
- ^ Paez-Espino, David; Zhou, Jinglie; Roux, Simon; Nayfach, Stephen; Pavlopoulos, Georgios A.; Schulz, Frederik; McMahon, Katherine D.; Walsh, David; Woyke, Tanja; Ivanova, Natalia N .; Eloe-Fadrosh, Emiley A.; Tringe, Susannah G.; Kyrpides, Nikos C. (2019-12-10). "Diversity, evolution, and classification of virophages uncovered through global metagenomics". Mikrobiyom. 7 (1): 157. doi:10.1186/s40168-019-0768-5. PMC 6905037. PMID 31823797.
- ^ Duponchel, S; Fischer, MG (2019). "Viva lavidaviruses! Five features of virophages that parasitize giant DNA viruses". PLOS Pathog. 15 (3): e1007592. doi:10.1371/journal.ppat.1007592. PMC 6428243. PMID 30897185..
 Materyal, bir altında bulunan bu kaynaktan kopyalandı. Creative Commons Attribution 4.0 Uluslararası Lisansı.
Materyal, bir altında bulunan bu kaynaktan kopyalandı. Creative Commons Attribution 4.0 Uluslararası Lisansı. - ^ Fischer, M. G.; Allen, M. J.; Wilson, W. H.; Suttle, C. A. (2010). "Giant virus with a remarkable complement of genes infects marine zooplankton" (PDF). Ulusal Bilimler Akademisi Bildiriler Kitabı. 107 (45): 19508–19513. Bibcode:2010PNAS..10719508F. doi:10.1073/pnas.1007615107. PMC 2984142. PMID 20974979.
- ^ Matthias G. Fischer; Michael J. Allen; William H. Wilson; Curtis A. Suttle (2010). "Giant virus with a remarkable complement of genes infects marine zooplankton" (PDF). Ulusal Bilimler Akademisi Bildiriler Kitabı. 107 (45): 19508–19513. Bibcode:2010PNAS..10719508F. doi:10.1073/pnas.1007615107. PMC 2984142. PMID 20974979.
- ^ Massana, Ramon; Javier Del Campo; Christian Dinter; Ruben Sommaruga (2007). "Crash of a population of the marine heterotrophic flagellate Cafeteria roenbergensis by viral infection". Çevresel Mikrobiyoloji. 9 (11): 2660–2669. doi:10.1111/j.1462-2920.2007.01378.x. PMID 17922751. S2CID 30191542.
- ^ Waldor MK, Friedman DI, Adhya SL, eds. (2005). Phages: their role in bacterial pathogenesis and biotechnology. Washington DC: ASM Press. pp.450. ISBN 978-1-55581-307-9.
- ^ Käse, Laura; Geuer, Jana K. (2018). "Phytoplankton Responses to Marine Climate Change – an Introduction". YOUMARES 8 – Oceans Across Boundaries: Learning from each other. pp. 55–71. doi:10.1007/978-3-319-93284-2_5. ISBN 978-3-319-93283-5.
- ^ Weitz JS, Wilhelm SW (2013). "An ocean of viruses". Bilim insanı. 27 (7): 35–39.
- ^ Suttle CA (September 2005). "Viruses in the sea". Doğa. 437 (7057): 356–61. Bibcode:2005Natur.437..356S. doi:10.1038/nature04160. PMID 16163346. S2CID 4370363.
- ^ Wilhelm SW, Suttle CA (1999). "Viruses and nutrient cycles in the sea: viruses play critical roles in the structure and function of aquatic food webs". BioScience. 49 (10): 781–88. doi:10.2307/1313569. JSTOR 1313569.
- ^ Suttle CA (October 2007). "Marine viruses—major players in the global ecosystem". Doğa İncelemeleri Mikrobiyoloji. 5 (10): 801–12. doi:10.1038/nrmicro1750. PMID 17853907. S2CID 4658457.
- ^ Suttle CA (September 2005). "Viruses in the sea". Doğa. 437 (7057): 356–61. Bibcode:2005Natur.437..356S. doi:10.1038/nature04160. PMID 16163346. S2CID 4370363.
- ^ Shelford EJ, Suttle CA (2018). "Virus-mediated transfer of nitrogen from heterotrophic bacteria to phytoplankton". Biyojeoloji. 15 (3): 809–15. Bibcode:2018BGeo...15..809S. doi:10.5194/bg-15-809-2018.
- ^ Weinbauer, Markus G.; et al. (2007). "Synergistic and antagonistic effects of viral lysis and protistan grazing on bacterial biomass, production and diversity". Çevresel Mikrobiyoloji. 9 (3): 777–788. doi:10.1111/j.1462-2920.2006.01200.x. PMID 17298376.
- ^ Robinson, Carol, and Nagappa Ramaiah. "Microbial heterotrophic metabolic rates constrain the microbial carbon pump." The American Association for the Advancement of Science, 2011.
- ^ Heinrichs, M.E., Mori, C. and Dlugosch, L. (2020) "Complex Interactions Between Aquatic Organisms and Their Chemical Environment Elucidated from Different Perspectives". İçinde: YOUMARES 9-The Oceans: Our Research, Our Future , pages 279–297. Springer. doi:10.1007/978-3-030-20389-4_15.
- ^ Dávila-Ramos S, Castelán-Sánchez HG, Martínez-Ávila L, Sánchez-Carbente MD, Peralta R, Hernández-Mendoza A, et al. (2019). "A Review on Viral Metagenomics in Extreme Environments". Mikrobiyolojide Sınırlar. 10: 2403. doi:10.3389/fmicb.2019.02403. PMC 6842933. PMID 31749771.
- ^ Fuhrman, Jed A. (1999). "Marine viruses and their biogeochemical and ecological effects". Doğa. 399 (6736): 541–548. Bibcode:1999Natur.399..541F. doi:10.1038/21119. ISSN 0028-0836. PMID 10376593. S2CID 1260399.
- ^ Tsai, An-Yi, Gwo-Ching Gong, and Yu-Wen Huang. "Importance of the Viral Shunt in Nitrogen Cycling in Synechococcus Spp. Growth in Subtropical Western Pacific Coastal Waters." Terrestrial, Atmospheric & Oceanic Sciences25.6 (2014).
- ^ Wilhelm, Steven W.; Suttle, Curtis A. (1999). "Viruses and nutrient cycles in the sea: viruses play critical roles in the structure and function of aquatic food webs". BioScience. 49 (10): 781–788. doi:10.2307/1313569. JSTOR 1313569.
- ^ Bar-On, YM; Phillips, R; Milo, R (2018). "Dünyadaki biyokütle dağılımı". PNAS. 115 (25): 6506–6511. doi:10.1073 / pnas.1711842115. PMC 6016768. PMID 29784790.
- ^ a b Suttle, Curtis A. (2005). "Viruses in the sea". Doğa. 437 (7057): 356–361. Bibcode:2005Natur.437..356S. doi:10.1038/nature04160. ISSN 0028-0836. PMID 16163346. S2CID 4370363.
- ^ www.cdc.gov. Harmful Algal Blooms: Red Tide: Home [Retrieved 2014-12-19].
- ^ a b c Suttle, Curtis A. (October 2007). "Marine viruses — major players in the global ecosystem". Doğa İncelemeleri Mikrobiyoloji. 5 (10): 801–812. doi:10.1038/nrmicro1750. ISSN 1740-1526. PMID 17853907. S2CID 4658457.
- ^ Breitbart M, Thompson L, Suttle C, Sullivan M (2007-06-01). "Exploring the Vast Diversity of Marine Viruses" (PDF). Oşinografi. 20 (2): 135–139. doi:10.5670/oceanog.2007.58.
- ^ Hurwitz BL, U'Ren JM (June 2016). "Viral metabolic reprogramming in marine ecosystems". Mikrobiyolojide Güncel Görüş. 31: 161–168. doi:10.1016/j.mib.2016.04.002. PMID 27088500.
- ^ Hurwitz BL, Hallam SJ, Sullivan MB (November 2013). "Metabolic reprogramming by viruses in the sunlit and dark ocean". Genom Biyolojisi. 14 (11): R123. doi:10.1186/gb-2013-14-11-r123. PMC 4053976. PMID 24200126.
- ^ Anantharaman K, Duhaime MB, Breier JA, Wendt KA, Toner BM, Dick GJ (May 2014). "Sulfur oxidation genes in diverse deep-sea viruses". Bilim. 344 (6185): 757–60. Bibcode:2014Sci...344..757A. doi:10.1126/science.1252229. hdl:1912/6700. PMID 24789974. S2CID 692770.
- ^ Roux S, Hawley AK, Torres Beltran M, Scofield M, Schwientek P, Stepanauskas R, et al. (Ağustos 2014). "Ecology and evolution of viruses infecting uncultivated SUP05 bacteria as revealed by single-cell- and meta-genomics". eLife. 3: e03125. doi:10.7554/elife.03125. PMC 4164917. PMID 25171894.
- ^ Forterre P, Philippe H (1999). "The last universal common ancestor (LUCA), simple or complex?". Biyolojik Bülten. 196 (3): 373–5, discussion 375–7. doi:10.2307/1542973. JSTOR 1542973. PMID 11536914.
- ^ Culley, A.I.; Lang, A.S.; Suttle, C.A. (2006). "Metagenomic analysis of coastal RNA virus communities". Bilim. 312 (5781): 1795–1798. Bibcode:2006Sci...312.1795C. doi:10.1126/science.1127404. PMID 16794078. S2CID 20194876.
- ^ a b Rahlff, Janina (2019). "The Virioneuston: A Review on Viral–Bacterial Associations at Air–Water Interfaces". Virüsler. 11 (2): 191. doi:10.3390/v11020191. PMC 6410083. PMID 30813345..
 Materyal, bir altında bulunan bu kaynaktan kopyalandı. Creative Commons Attribution 4.0 Uluslararası Lisansı.
Materyal, bir altında bulunan bu kaynaktan kopyalandı. Creative Commons Attribution 4.0 Uluslararası Lisansı. - ^ a b Hutchinson GE (1961). "The Paradox of the Plankton". Amerikan Doğa Uzmanı. 95 (882): 137–145. doi:10.1086/282171. JSTOR 2458386.
- ^ Danovaro R, Dell'Anno A, Corinaldesi C, Magagnini M, Noble R, Tamburini C, Weinbauer M (August 2008). "Major viral impact on the functioning of benthic deep-sea ecosystems". Doğa. 454 (7208): 1084–7. Bibcode:2008Natur.454.1084D. doi:10.1038/nature07268. PMID 18756250. S2CID 4331430.
- ^ Anderson, Rika E.; Brazelton, William J.; Baross, John A. (2011). "Is the genetic landscape of the deep subsurface biosphere affected by viruses?". Mikrobiyolojide Sınırlar. 2: 219. doi:10.3389/fmicb.2011.00219. ISSN 1664-302X. PMC 3211056. PMID 22084639.
- ^ Anderson, Rika E.; Sogin, Mitchell L.; Baross, John A. (2014-10-03). "Evolutionary Strategies of Viruses, Bacteria and Archaea in Hydrothermal Vent Ecosystems Revealed through Metagenomics". PLOS ONE. 9 (10): e109696. Bibcode:2014PLoSO...9j9696A. doi:10.1371/journal.pone.0109696. ISSN 1932-6203. PMC 4184897. PMID 25279954.
- ^ Ortmann, Alice C.; Suttle, Curtis A. (August 2005). "High abundances of viruses in a deep-sea hydrothermal vent system indicates viral mediated microbial mortality". Derin Deniz Araştırmaları Bölüm I: Oşinografik Araştırma Makaleleri. 52 (8): 1515–1527. Bibcode:2005DSRI...52.1515O. doi:10.1016/j.dsr.2005.04.002. ISSN 0967-0637.
- ^ Breitbart, Mya (2012-01-15). "Marine Viruses: Truth or Dare". Deniz Bilimi Yıllık İncelemesi. 4 (1): 425–448. Bibcode:2012ARMS....4..425B. doi:10.1146/annurev-marine-120709-142805. ISSN 1941-1405. PMID 22457982.
- ^ Goldenfeld, Nigel; Woese, Carl (January 2007). "Biology's next revolution". Doğa. 445 (7126): 369. arXiv:q-bio/0702015. Bibcode:2007Natur.445..369G. doi:10.1038/445369a. ISSN 0028-0836. PMID 17251963. S2CID 10737747.
- ^ Leggett HC, Buckling A, Long GH, Boots M (October 2013). "Generalism and the evolution of parasite virulence". Ekoloji ve Evrimdeki Eğilimler. 28 (10): 592–6. doi:10.1016/j.tree.2013.07.002. PMID 23968968.
- ^ Flores CO, Valverde S, Weitz JS (March 2013). "Multi-scale structure and geographic drivers of cross-infection within marine bacteria and phages". ISME Dergisi. 7 (3): 520–32. doi:10.1038/ismej.2012.135. PMC 3578562. PMID 23178671.
- ^ Krupovic M, Bamford DH (July 2007). "Putative prophages related to lytic tailless marine dsDNA phage PM2 are widespread in the genomes of aquatic bacteria". BMC Genomics. 8: 236. doi:10.1186/1471-2164-8-236. PMC 1950889. PMID 17634101.
- ^ Xue H, Xu Y, Boucher Y, Polz MF (January 2012). "High frequency of a novel filamentous phage, VCY φ, within an environmental Vibrio cholerae population". Uygulamalı ve Çevresel Mikrobiyoloji. 78 (1): 28–33. doi:10.1128/AEM.06297-11. PMC 3255608. PMID 22020507.
- ^ a b Roux S, Krupovic M, Poulet A, Debroas D, Enault F (2012). "Virom okumalarından toplanan 81 yeni tam genomdan oluşan bir koleksiyon yoluyla Microviridae viral ailesinin evrimi ve çeşitliliği". PLOS ONE. 7 (7): e40418. Bibcode:2012PLoSO...740418R. doi:10.1371 / journal.pone.0040418. PMC 3394797. PMID 22808158.
- ^ Hurwitz BL, Sullivan MB (2013). "The Pacific Ocean virome (POV): a marine viral metagenomic dataset and associated protein clusters for quantitative viral ecology". PLOS ONE. 8 (2): e57355. Bibcode:2013PLoSO...857355H. doi:10.1371/journal.pone.0057355. PMC 3585363. PMID 23468974.
- ^ Roux S, Enault F, Hurwitz BL, Sullivan MB (2015-05-28). "VirSorter: mining viral signal from microbial genomic data". PeerJ. 3: e985. doi:10.7717/peerj.985. PMC 4451026. PMID 26038737.
- ^ Ren J, Ahlgren NA, Lu YY, Fuhrman JA, Sun F (July 2017). "VirFinder: a novel k-mer based tool for identifying viral sequences from assembled metagenomic data". Mikrobiyom. 5 (1): 69. doi:10.1186/s40168-017-0283-5. PMC 5501583. PMID 28683828.
- ^ Paez-Espino D, Eloe-Fadrosh EA, Pavlopoulos GA, Thomas AD, Huntemann M, Mikhailova N, et al. (Ağustos 2016). "Uncovering Earth's virome". Doğa. 536 (7617): 425–30. Bibcode:2016Natur.536..425P. doi:10.1038/nature19094. PMID 27533034. S2CID 4466854.
- ^ Coutinho FH, Silveira CB, Gregoracci GB, Thompson CC, Edwards RA, Brussaard CP, et al. (Temmuz 2017). "Marine viruses discovered via metagenomics shed light on viral strategies throughout the oceans". Doğa İletişimi. 8: 15955. Bibcode:2017NatCo...815955C. doi:10.1038/ncomms15955. PMC 5504273. PMID 28677677.






